遺伝病研究における次世代技術の使用が普及している。 exomeおよび全ゲノムシーケンシングが利用可能になると、データの解析と解釈が必要になる。遺伝病の研究に使えるVarSifter [論文より ref.1]、GEMINI [ref.2]、GeneTalk [ref.3]、CanvasDB [ref.4]、ExomeSuite [ref.5]、wKGGSeq [ref.6]などの既存のツールがあり、突然変異を引き起こす私たちの病気を見つけることができる。これらのツールのほとんどは、劣性および優性の症例に対処できるが、複合ヘテロ接合およびde novo変異を分析することができる公的に利用可能なプログラムがない。
GeneTalkは、特に複合ヘテロ接合向けに設計されているが、フルバージョンは商用である。パブリックバージョンは複合ヘテロ接合解析をサポートしていない。 GEMINIおよびwKGGSeqは、変異が段階的になっている場合にのみ複合ヘテロ接合体モデルを支持し、そうでなければ二重ヒット戦略を用いる。さらに、wKGGSeqは、KGGSeqによって病原性であると予測される変異のみを考慮する。 ExomeSuiteはダブルヒット戦略も使用する。第3.2.1節では、ダブルヒット戦略が複合ヘテロ接合解析を正確に実行しないことを示し、誤りを増加させる例を提供する。 VarSifterを用いた複合ヘテロ接合症例の取り扱いも容易ではない。著者らは、既存のツールの複合ヘテロ接合解析の結果を比較した(一部略)。
ここでは、与えられた基準に従って候補バリアントを効果的にフィルタリングする、継承モデルベースのツールであるFMFilterを提供する。著者らの知る限り、FMFilterは、複合ヘテロ接合モデルとDE novoモデルを適切に扱うことができる最初の公的に利用可能なツールである(論文執筆時点)。これは、FMFilterと、2つの公開された複合ヘテロ接合症例[ref.7,8]における強力なフィルタリングオプションを使用して承認されている。また、プログラムには使いやすいグラフィカルユーザーインターフェイスがあり、プログラミングに関する知識は必要なくなっている。このプログラムは、効率的なメモリ処理技術を考慮して設計されており、通常のコンピュータを使用して非常に大きなバリアントファイルを処理するために効果的に使用できる。
FMFilterの利点は、既存のツールと比較して必要とされる計算リソースの量の削減である。これにより、研究者はパーソナルコンピュータでツールを実行できる。主な制約はI / O境界だが、プログラムのメモリ使用量はごくわずかである。したがって、バリアントファイルサイズはプログラムの使用を制限しない。この機能により、FMFilterは、既存のツールのほとんどが開くことができないか、多くの読み込みと実行時間を必要とする場所で、大きなVCFファイルを簡単に処理できる。論文の表1では、HDDおよびSSDのストレージを考慮したプログラムの性能を示す。 FMFilterの使用法を解釈するために、異なる病気モデルから採取したサンプルvcfを検討した。サンプルには、パブリックデータ(pigo.vcf [ref.3]および1000ゲノムのvcf)およびin houseのケースの両方が含まれる。巨大ファイル例として、1000ゲノムプロジェクトからの15ギガバイトのVCFファイルと28ゲノムからなる100ギガバイトの社内VCFファイルを検討した。
公式ページ
http://fmfilter.sourceforge.net
マニュアル
http://fmfilter.sourceforge.net/manual.html
インストール
ラン
ここではwindows版を説明する。
上記からダウンロードしたexeファイルを実行する。

支持に従いインストールする。
起動する。
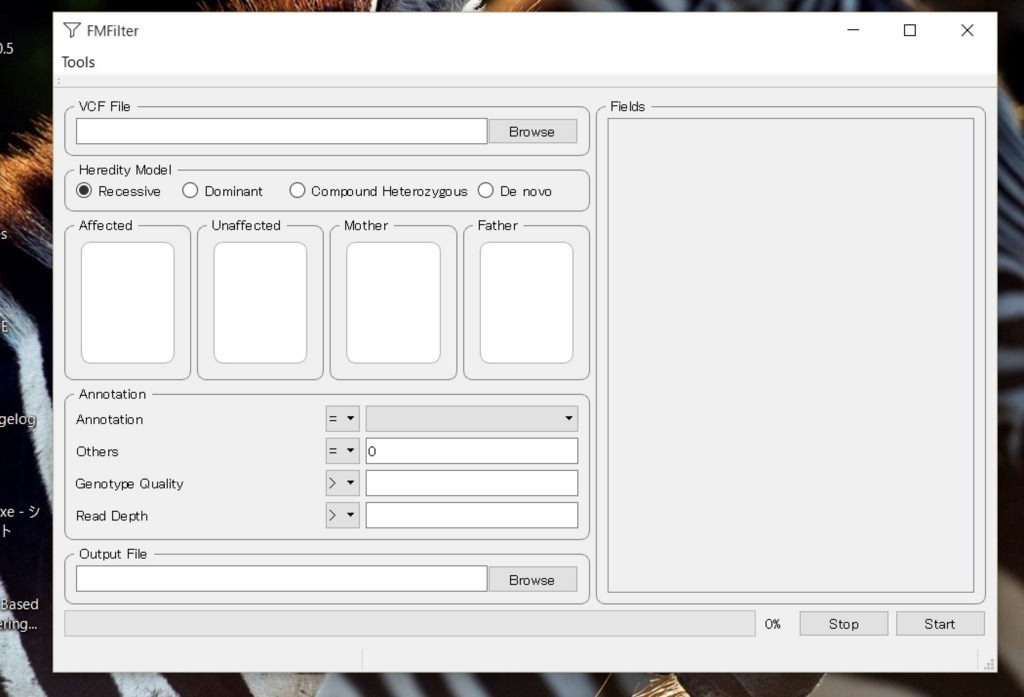
BrowseからVCFを読み込む。UK10K プロジェクト(nature)のexampleデータなどが利用できればよかったが(UK10K Study Samples)、ダウンロードにあたり登録申請が手間そうだったので諦めた。ここでは1000genomesのVCFを読み込んだ。1000genomesは家族、系統などの情報も消されているが、ここでは仮に両親と子供(患者)、unaffectedが以下のIDとする。複数選択は続けてクリックする。
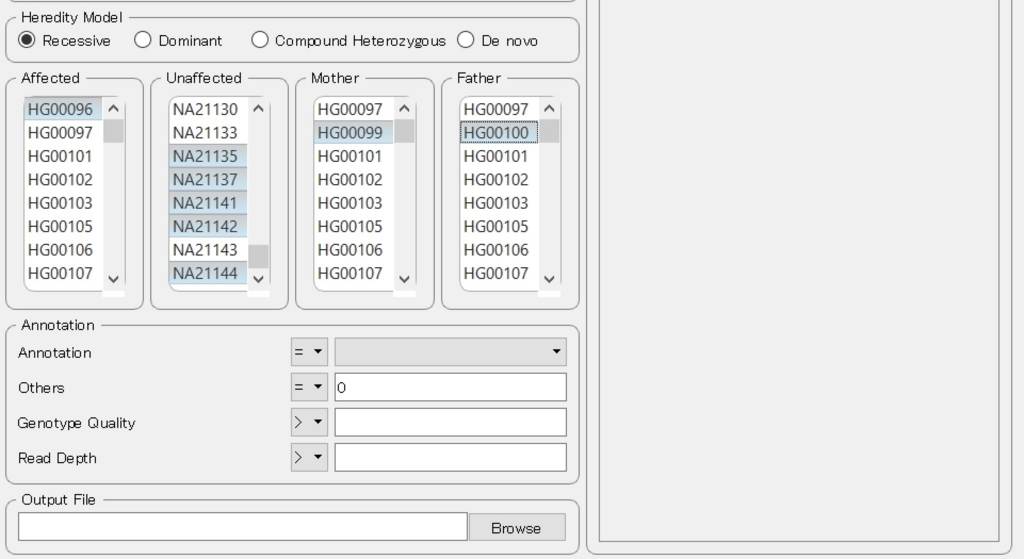
発症していない両親(ヘテロキャリア)、発症した子供から、ホモの劣勢形質の原因遺伝子の変異を検出したいので、Recessiveを選択(両親にない新規変異ならばDe novo)。
ファミリーのトリオやカルテットの解析ではなくて影響ありと影響なしの2群比較なら、affectedとunaffectedだけ指定する。複合ヘテロ接合体(compound heterozygous)の解析ならCompound Heterozygousを選ぶ(See manual)。
ほかのフィルター条件追加できる。カバレッジ、Genotype qualityを指定できる。Othersは0だと、該当患者のみの出力になり、それ以外の数値だと最大で指定した数値までの患者以外のhitも許容するようになるらしい。populationsのデータ解析で役に立つとマニュアルに書かれている。
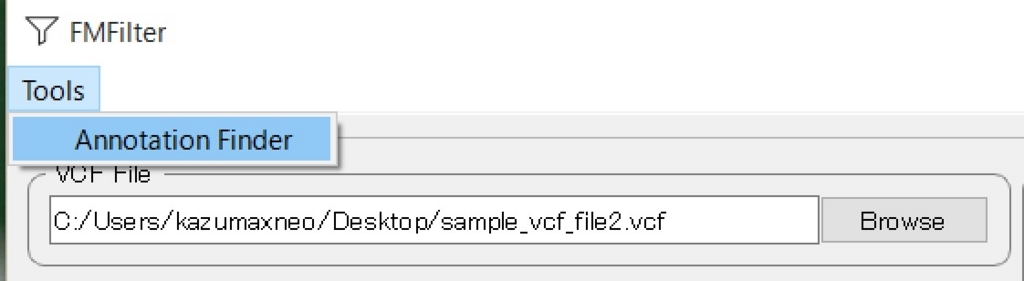
Annotationの項目でフィルタイングするには、ToolメニューからAnnotation Finderを起動し、既知バリアントにアノテーションをつけたVCFを作って、それを読み込む必要がある。(Tools => Annotation Finder)。
右側のウィンドウは、GTフィールドに記載があれば表示される。たとえばGATKではこのフィールドがある(VCF)。

マニュアルより転載。
ほかに、大規模プロジェクトのバリアントデータの頻度を利用してフィルタリングするMAFのウィンドウも用意されている(上の写真の右下)。MAF(リンク)のフィルタリングを行うには、Tools => Annotation Finderのアノテーション段階で、大規模プロジェクトのバリアント情報をVCFに取り込んでおく必要がある。
準備ができたら、右下のstartでランする。おわると指定したパスにフィルタリングされったVCFが出力される。
注 論文で書かれている1000genome のVCFをchr1の10%だけ読み込んだが(およそ1.8GB)、エラーを起こした。GT yieldがないためか?(未検討)。
引用
FMFilter: A fast model based variant filtering tool.
Akgün M, Faruk Gerdan Ö, Görmez Z, Demirci H.
J Biomed Inform. 2016 Apr;60:319-27.