2019 1/17 タイトル修正
FasParser1 abstract
シーケンスデータを操作するために、「FasParser」と呼ばれるコンピュータソフトウェアパッケージを開発した。これはパーソナルコンピュータで様々な分析を行うことができるソフトウエアで、その機能には、カウント、2配列間の違いを調べること、2間の重複領域を同定すること、IDや長さによるソート、複数の遺伝子座の連結、ヌクレオチド配列のアミノ酸への翻訳、およびいくつかの異なるフォーマットでアライメントを構築すること、ならびにいくつかの抽出およびフィいる多リングが含まれる。 これらの機能の多くはバッチモードで実行することができる。これは大きなデータセットの分析に非常に役立つ。 このパッケージは誰でも使用でき、プログラミング経験のないシーケンス分析研究者向けに設計されている。 FasParserのGUI版はhttps://github.com/Sun-Yanbo/FasParserから無料でダウンロードできる。
FasParser2 abstract
FasParserは、シーケンスと配列をバッチモードで操作するグラフィカルなプラットフォームである。 これは、プログラミングの経験がなくても大きなシーケンシングデータセットを扱う生物学者にとって特に有用である。 ここでは、シーケンス操作を容易にすることができる数多くの新しい機能セットと改良を加えた、新バージョンの'FasParser2'を紹介する。 その主な追加機能には、(i)バッチ処理能力を大幅に向上させる再設計されたグラフィカルインタフェース、(ii)複数配列間のアライメント時に低アライメント領域を除去するトリミング機能、(iii) 「悪い」配列(あまりに短い配列または多すぎる配列)、 (iv) biological sequenceを表示して編集するためのエディタツール、ならびに (v) ポジティブセレクションおよびプライマー設計のためのインターフェース、がある。
Windows専用となっている。基本的な機能が多いが、複数のファイルを入れてチェックをつければ、順番に処理して自動でファイル出力してくれます。このバッチモードの使い勝手が良く、研究や業務の内容次第ですが、うまく使えば自動化の道が開けます(そうゆう意味ではCLC genomicsやGeneiousに似てます)。
このブログでいつも紹介しているように、本ツールも無料で使えます(Donateボタンから寄付ができます。開発を支援したければ寄付を検討して下さい)。
FasParserに関するツイート。
インストール
mac os 10.13のparallels13で動かしているwindows 10 proでテストした。
本体(FasParser2)Github(PDFマニュアルもあり)
リリースからsetup.exeをダウンロードする。
ラン
1、Editer
遺伝子配列やアミノ酸配列を編集できる。使い方は一般的な遺伝子編集ソフトと同じなので、説明は割愛する。

2、ID
editer以外の機能は使い方がやや独特なので動画を用意した。FasParser2を使い。バクテリアゲノムのFASTAファイルからヘッダーのIDを抽出する作業を行なっている。
Runを押すと抽出が始まり、終わると同時に別ファイルが出力される。よくみると、左のほうにアイコンができていることがわかる。動画の最後ではそれを開いている。
3、Alignment
Alignment
Alignのマルチプルアライメントを選択する。

IDと同様、別ウィンドウが出現する。
マルチプルアライメントを実行したいサンプルにチェックをつける。後から追加したければ、このウィンドウにドラッグすることでさらに追加できる。

マルチプルアライメントのツールはmafftを選択。ここでは、multi-fastaファイルとしてmafft(紹介)のexampleファイル(リンク1dna、リンク2protein)を使う。準備できたら中央のRunボタンをクリックしてジョブを開始する。58のrRNA遺伝子のmulti-FASTAの解析にはMuscleで1分ほどかかった。.muscleファイルができる。
Alignment: Trimming
終わるとマルチプルアライメント結果のファイルができる。このままウィンドウを閉じずに低クオリティ領域のトリミングを実行できる。マルチプルアライメントのラン後、中央部分左手のTrimingボタンをクリックする。

DNAのアライメントならCodonからDNAに切り替える。

マルチプルアライメントより時間がかかる。終わると、アライメントが不十分な部位がトリミングされる(デフォルトではかなり強力にトリミングされるので、処理前のファイルも確認してください)。
マルチプルアライメント結果をビューアで確認する。ここでは大量のデータでも高速動作するAlivew(リンク)(Larsson A., 2014)を使う。

windows版もあるが、mac版をダウンロードする(ダウンロードリンク)。
.muscle.fasファイルを読み込む。
トリミング前

トリミング後

一定のウィンドウサイズで分析するため、連続して保存されているアミノ酸残基だけ残る。
Alignment: identity
この機能は、3つのアイデンティティ計算を提供する:アラインメント内の最小ペアワイズアイデンティティ(最小); 最大ペア単位の同一性(最大)。 平均的な同一性(平均)。

計算はすぐに終わります。
Alignment: Formats
真ん中のFormatsボタンでは、マルチプルアライメント結果を FASTA、PHYLIP (Phylipi and Phylips)、PAML、NEXUSフォーマットに変換できる。この機能は分子系統樹を描くときに役に立つ。

4、Merge
multi-FASTAの結合。
5、Sort
指定した順番でソートする(abc順、長さ順など)。

分析の都合で改行が取り除かれて出力される。

6、Filter
サイズ、キーワード、コドン、などでmulti-fastaの特定の配列だけ抽出できる。
ヘッダー(ID)にplasmidが含まれる配列だけ抽出。

1000bp以上-6000bp以下の配列だけ抽出。

他にギャップ(-)の割合でフィルタリングしたり、指定した配列との相同性でフィルタリングすることができます(テスト時は相同性フィルタリングでクラッシュした)。
7、DNA2AA
DNAをアミノ酸に翻訳する。muliti-fastaのDNA配列を一度に翻訳することができる。

8、ORF
DNAからコード領域を見つける。longestかget all the potential ORFか選択できる。

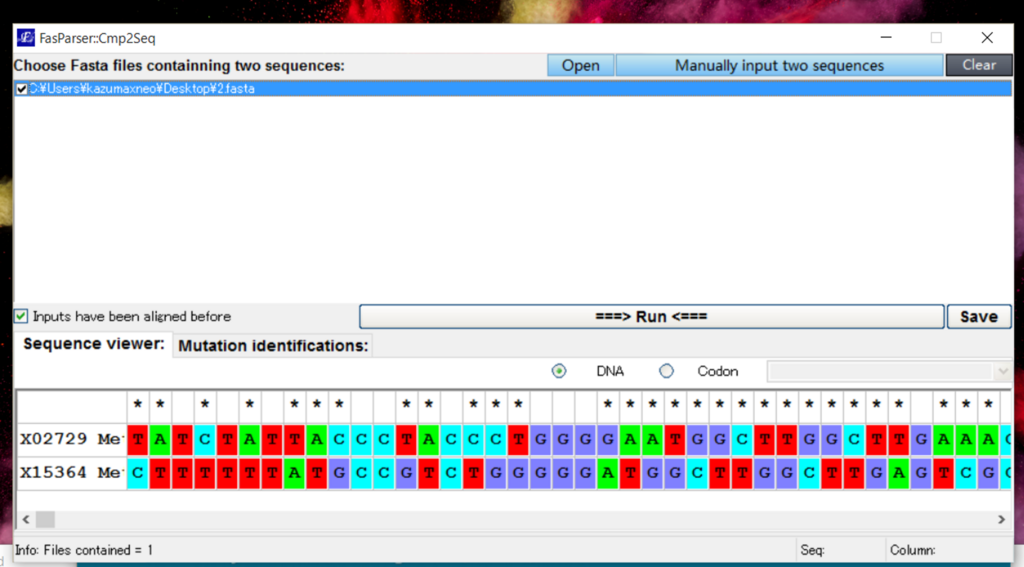
9、Comp
Comp-2Seq
2配列のfastaを入力として、配列を比較する(Runを押してアライメント実行)。DNAでもアミノ酸でも比較可能。
Comp-2Align
異なるアライナーのアライメント結果を比較する。
10、Primer
配列をペーストすれば、primer3を使い、設定した条件でプライマーを自動設計してくれる。

テスト時はprimer3 APIが見つからないとしてエラーになった。
環境設定はウィンドウ右下のSettingから行う。他にもいくつかの機能があります。詳細はマニュアルPDFで確認して下さい。
引用
FasParser2: a graphical platform for batch manipulation of tremendous amount of sequence data
Bioinformatics. 2018 Jul 15;34(14):2493-2495
Yan-Bo Sun
FasParser: a package for manipulating sequence data.
Yan-Bo Sun
Zool Res. 2017 Mar 18;38(2):110-112
AliView: a fast and lightweight alignment viewer and editor for large data sets
Larsson A
Bioinformatics. 2014 Nov 15;30(22):3276-8