随時更新
情報が増えてきたので、これまで紹介してきたfasta、fastqの分析、変換(圧縮)、修復ツールをまとめておく。
- アダプタートリミング
- seqkit fastq / fastaの操作ツール seqkit seqkitに最近追加されたコマンド
- seqtk fastq / fastaの操作ツール seqtk
- BBtools 多機能なNGSの管理ツール BBtools
- ATGCのカウント FASTAやFASTQの塩基数をカウント
- Multi-fastaの分割 Mulit-FASTAの分割 (split)
- 高速なk-merカウント 高速なk-merカウントツール KMC
- Low/High k-merを含むfastqの除去 diginormによるシーケンスデータの軽量化
- fastqの圧縮 高速で高効率なfastqの圧縮ツール DSRC
- fastqの検証 FastQValidatorでfastqデータを検証
- fastqの修復 embossのseqretでFASTAを修復
- オーバーラップするfastqのマージ fastqの操作ツール illumina-utils
- 似た配列のクラスタリング cd-hitで似た配列をクラスタリング
- Fastq => Fasta 変換 フォーマット変換 Fastq=> Fasta
- Fastq => BED 変換 フォーマット変換 FASTA => BED
- Genbank => Fasta 変換 フォーマット変換 genbank => fasta
- Bam => Fasta変換 フォーマット変換 bam=> Fastq アライメントされなかったリードの取り出し方等
- fastqのトリミング トリミングツール カテゴリーの記事一覧
- fastqのシミュレーション 次世代リードのシミュレーター カテゴリーの記事
- エラー訂正 エラーコレクション カテゴリーの記事
- マルチプルアライメント マルチプルアライメント カテゴリーの記事
- ナノポアのFAST5 => Fasta 変換 Oxford NanoporeリードのFAST5 => FASTA / FASTQ変換
- 結果の統合 様々なツールの分析結果を1つに集約して分析する MulitiQC
- ペアリードのマージ エラーを除去しながらペアリードをマージする CASPER
- fastqの汎用処理ツール fastqの処理ツール fqtools
- Ngs crumbs NGSの スモールユーティリティツール Ngs crumbs
- fastqutils FASTQ、BED、BAMを操作するNGSUtilsその3 fastqutils
- Goldilocks FASTA分析に使えるpythonライブラリ Goldilocks
- BBSketch minHashを使いfasta / fastqから生物種を高速推定する BBSketch
- 高速なfastqの前処理パイプライン fastp 高速なfastqの前処理パイプライン fastp
- fastqから素早くインサートサイズの平均を出す fastqから素早くインサートサイズを計算する
- fastq-dumpを並列処理する fastq-dumpを並列で行い高速化するスクリプト pfastq-dump
- fastqの分析ツール シンプルなfastq、sam、bamの分析ツール fastqp
- fasta/fastqの簡単な要約統計 fasta、fastqの簡単なstatisticsを出す Seqstats
- configのリファレンス順ソート mauveを使いcontigをリファレンスfasta順に並べ替え
- カバレッジ、GC を素早くグラフにする カバレッジやGCを可視化するwebツールgbtlite
- fastqをランダムな配列に変換 fastqの配列をランダムに変化 fastq-anonymous
- 複数リファレンスへのmapping マッピングやフィルタリングする FastQ Screen
- webで手持ちのゲノムと遺伝子のblast検索実行 blast検索できるwebツール SimpleSynteny
- fastq-dumpを高速化 並列実行して高速化する parallel-fastq-dump
- ペアエンドのトリミング、マージを一括して行う ClipAndMerge
- 高速なペアエンドのマージツール ペアエンドfastqをマージする flash2
- フォーマット変換、fastaのパース seqmajic
- fastqの高効率圧縮 FASTQの圧縮/解凍を行う Spring
- 順番のずれたfastqの高速同期 ペアエンドfastqを同期する Fastq-pair
- FASTA形式に変換 様々なフォーマットをFASTA形式に変換 any2fasta
- 変異導入 点変異やSVを導入するEMBOSSのmsbar
- 低複雑性領域のマスク low complexity領域をマスクする komplexity
- fasta/fastq/bamのユーティリティツール fxtools
- vsearch 多機能な配列処理ツール VSEARCH
- fastqの分析結果をJSON形式で出力 JSON形式で出力 fastq-scan
- multi-fastaのGCや長さを出力 EMBOSSのinfoseqコマンド
- fastqをソートして圧縮やindexingを助ける Clumpify
- BLASTデータベースのFASTAを分割する AlignBucket
- アミノ酸配列に翻訳する gotranseq
- fastqのクオリティ分析を行う Quack
- 複数のFASTAファイルをNNN...で連結する CombineFasta
- ターゲットに関係ある/関係ない、配列を取り出す mirabait
- FASTA配列のオンラインツールボックス FaBox
- 前処理しながらターゲット配列(ウィルス、病原性細菌など)を検出 fastv
- 一定のカバレッジまでリードをランダムサンプリング rasusa
- fastqのためのgrep fqgrep
- 複数の配列(multi-fastaファイルなど)を結合 EMBOSSのunionコマンド
- Demultiplexingを行う fgbioのDemuxFastqsコマンド
- fastqをソートして扱いやすくする BBMapパッケージのClumpify
- 核酸配列をアミノ酸配列に翻訳 gotranseq
- Multi FASTAのGC含量や長さを表示 EMBOSSのinfoseqコマンド
- 追加配列のアノテーションを含めるようにfastaとgff3を改変 reform
- 配列ファイルを堅牢かつ再現性よく操作するためのユーティリティ群 SeqFu
- ライフサイエンスのための包括的なフォーマットコンバーター BioConvert
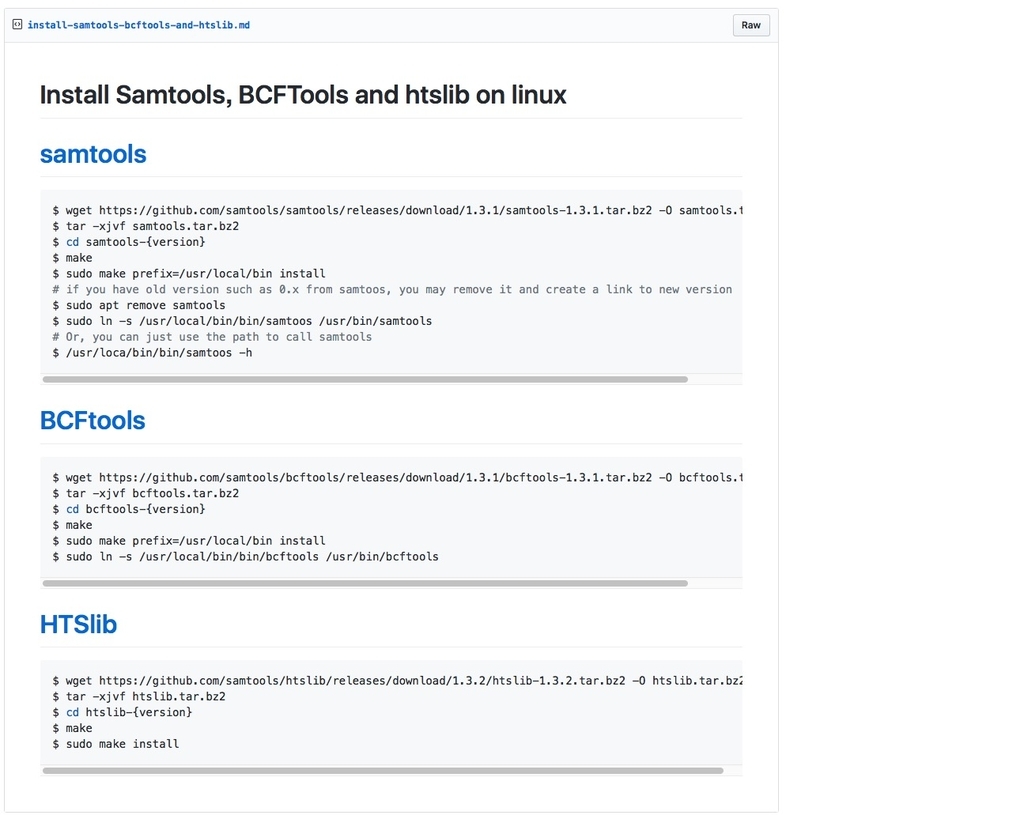
samtools/bcftools/htslibの導入はこちらを参考にしてください。綺麗にまとまってます。
Install samtools, bcftools and htslib on linux · GitHub
#Anaconda環境ならバージョン指定するだけで導入できます。(バージョンチェック)
conda install -c bioconda samtools==1.9
conda install -c bioconda bcftools==1.9
#htslibだけ入れたいなら
conda install -c bioconda htslib==1.9
高速化のTIPS
たくさんのデータに同じ処理をするなら、GNU parallelを使って並列化することで簡単に高速化できる。
例えばseqkit statsを全てのfastqに対して実行することを考える。ワイルドカードでも良いが、データが多いとかなり時間がかかってしまう。
seqkit stats -a -T *fq.gz > stats.txt
GNU parallelで並列化する("sudo apt-get install parallel")。
#listを作成
ls *fq.gz > list
#10並列(tailで1行目を捨ててエラー出力を追記)
cat list | parallel -j 10 'seqkit stats -a -T {} |tail -n 1 1>> stats.txt 2>&1'
*HDDだとI/Oが足を引っ張る可能性があります。並列処理数を抑えて下さい。
(parallel --citation => will cite、で引用するのを忘れない事)
ヘッダの連番へのリネーム
perl -ane 'if(/\>/){$a++;print ">name_$a\n"}else{print;}' input.fa > rename.fa
#or
awk '/>/ {$0 = ">rename_" ++n} 1' input.fa > rename.fa
またはseqkit renameを使う
#rename duplicateしたIDをユニークな名前に修正。duplicateしていると判断されると"_2"などがつく。
seqkit rename -n input.fa > renamed.fa
- -n check duplicated by full name instead of just id.
fastq => fasta
awk '(NR - 1) % 4 < 2' test.fq | sed 's/@/>/' > test.fa
#seqkit
seqkit fq2fa in.fq > out.fa
#8並列(他のコマンドでももちろん利用可能、CPUとメモリがたくさん利用できる時だけ)。linuxのGNU paralel使用。
ls *.fq | parallel -j 8 'seqkit fq2fa {} > {.}.fa'
fastq => fasta変換して、さらに連番へのリネームも同時実行
awk 'NR % 4 == 2 {print ">" ++n "\n" $0}' input.fq > output.fa
multi-lineのfastaからシングルラインのfastaに 修正(*1より)。
perl -pe '/^>/ ? print "\n" : chomp' input.fasta > output.fasta
指定領域を取り出す。
samtools faidx ref.fa chr2:1000-25000 > output.fa
(multi-)fastaの配列の改行を除き配列は1行で出力。(Biostarスレッドより)。
awk '/^>/ { if(NR>1) print ""; printf("%s\n",$0); next; } { printf("%s",$0);} END {printf("\n");}' input.fa > output.fa
multi-fastaの配列を合体させて強制的に1つに配列にする(Biostarスレッドより)。
cat multi_seq.fasta | sed -e '1!{/^>.*/d;}' | sed ':a;N;$!ba;s/\n//2g' > one_seq.fasta
#またはgrep ーVで>以外の行を抽出、seqretで修復
grep -V ">" multi_seq.fasta > out
seqret out one_seq.fasta
N(NNN...)を含む配列だけ除く。
#Nを1塩基以上含む配列は除く
cutadapt --quiet --max-n 1 input.fa > out.fa
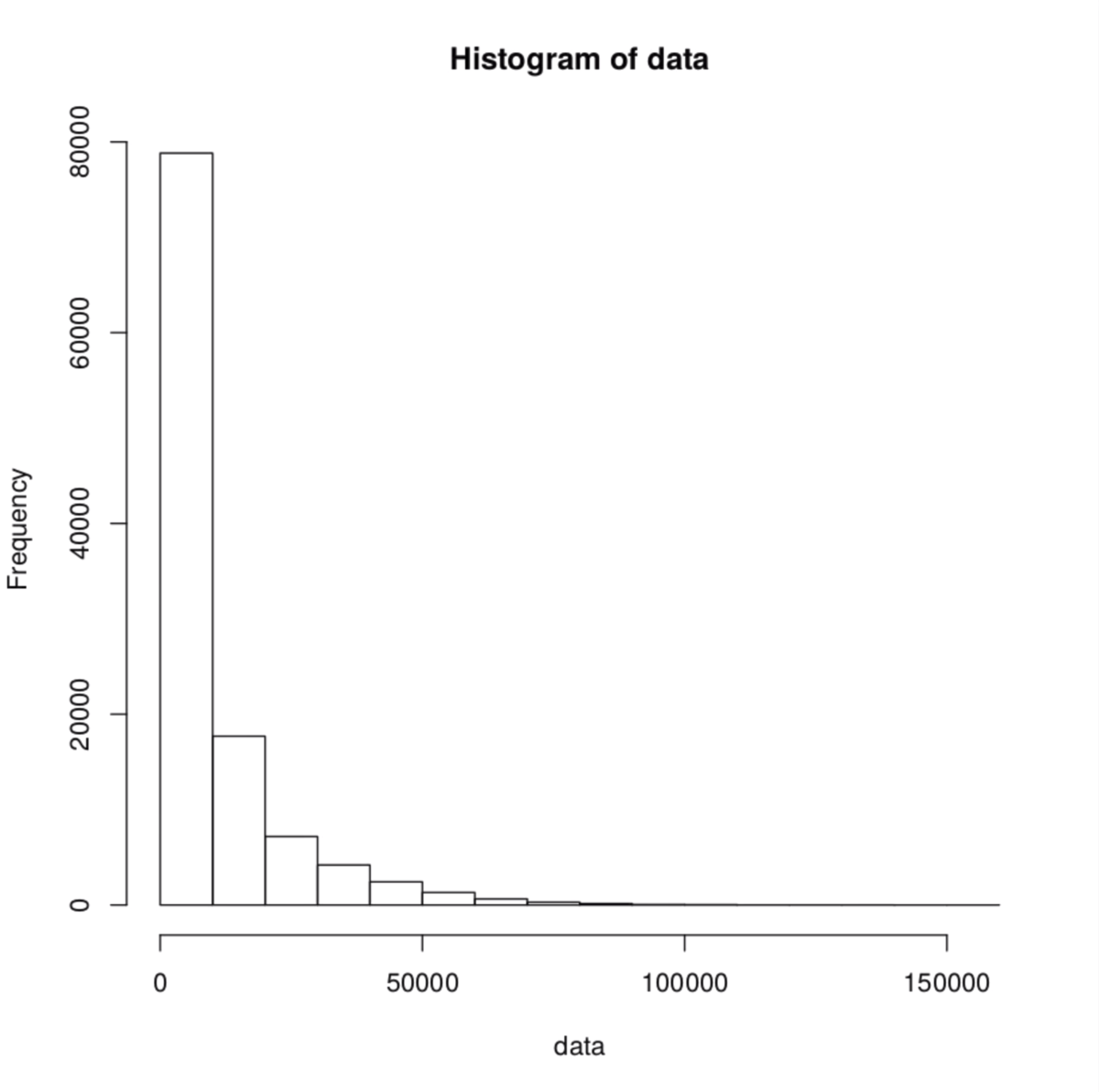
リード長分布
samtools faidx input.fasta
cut -f2 input.fasta.fai | Rscript -e 'data <- as.numeric (readLines ("stdin")); summary(data); hist(data)'
フォーマット変換
NGSなどのリードは対応していないが、小さめのFASTAデータセットであれば、オンラインで様々なフォーマットに変換できる。何千配列もあるような大きなデータは投げないよう注意する。
1、Sequence conversion
http://sequenceconversion.bugaco.com/converter/biology/sequences/

FASTAからNEXUS、Philip、clustal、EMBLなど様々なフォーマットに変換可能。
2、Sequence format converter
こちらも様々な出力に対応。
SMS
https://www.bioinformatics.org/sms2/
こちらも昔から保守されている。
-- seqkit --
カレントディレクトリの全fa.gzファイルを分析。
seqkit stats *.fa.gz
reverse complementary (相補鎖にしてさらにリバースにする)
seqkit seq -pr input.fa > output.fa
1000bp以上の配列だけ取り出す。
seqkit seq -m 1000 scaffolds.fa > output.fasta
1000bp以下の配列だけ取り出す。
seqkit seq -M 1000 scaffolds.fa > output.fasta
最初の100-bpを取り出す。
seqkit subseq -r 1:100 contigs.fa > left.fa
最後の100-bpを取り出す(マイナスで後ろからカウント)。
seqkit subseq -r -100:-1 contig.fa > right.fa
gtfで指定した領域の配列だけ取り出す。
seqkit subseq --gtf cDNA.gtf genome.fa > cdna.fa
100-bp windowでゲノム配列を切り出す(GC含量カウントに使ったりする)。
seqkit sliding -s 1 -W 100 genome.fa > 100bp_windows.fa
GC含量をカウント。
seqkit fx2tab genome.fa| head -n 1 | seqkit tab2fx | seqkit sliding -s 1 -W 100 | seqkit fx2tab -n -g > GC.txt
tab形式に変換し、GCなどの情報を表示。
seqkit fx2tab input.fa > output.fa
環状ゲノムのスタート位置を変える。
seqkit restart -i 100 genome.fa > genome_restart.fa
指定した配列を取り出す。opera_contig_216907~というヘッダの配列とopera_contig_162971~というヘッダの2配列のみ取り出す。
cat input.fasta |seqkit grep -p opera_contig_216907 -p opera_contig_162971
gzip圧縮ファイルならcat => zcatに変更。
長さでソートし、10000-bp以上の配列だけ出力。
seqkit sort --quiet -r -l conitg.fa -L 10000 > sorted.fa
-- BBtools --
contig.fastaを分析する。
stats.sh in=contigs.fa gc=gc.txt gcformat=4
N50などの基礎的な情報が出力される。
user$ stats.sh in=contigs.fa gc=gc.txt gcformat=4
A C G T N IUPAC Other GC GC_stdev
0.2841 0.2276 0.2531 0.2352 0.0000 0.0000 0.0000 0.4807 0.0583
Main genome scaffold total: 42
Main genome contig total: 42
Main genome scaffold sequence total: 0.004 MB
Main genome contig sequence total: 0.004 MB 0.000% gap
Main genome scaffold N/L50: 9/131
Main genome contig N/L50: 9/131
Main genome scaffold N/L90: 42/41
Main genome contig N/L90: 42/41
Max scaffold length: 352
Max contig length: 352
Number of scaffolds > 50 KB: 0
% main genome in scaffolds > 50 KB: 0.00%
Minimum Number Number Total Total Scaffold
Scaffold of of Scaffold Contig Contig
Length Scaffolds Contigs Length Length Coverage
-------- -------------- -------------- -------------- -------------- --------
All 42 42 3,520 3,520 100.00%
50 19 19 2,547 2,547 100.00%
100 11 11 2,009 2,009 100.00%
250 1 1 352 352 100.00%
gc含量は別ファイルに出力される。
たくさんファイルがあるなら、並列処理することを考える。たとえばGNU parallelを使って、カレントのfastaファイルを対象にstatsコマンドを走らせる(別々に保存したい)。
ls *fasta | parallel --jobs 8 'seqkit stats {} >{}_stas.txt' 2>error_log
出力
FASTQ。
-- parallel-fastq-dump --
SRAのfastqを高速ダウンロード、例えばペアエンドデータSRRxxxxxxをダウンロードしてout_dir/にペアエンドfastq.gz出力。
parallel-fastq-dump --threads 8 --split-files --gzip --outdir out_dir --sra-id SRRxxxxxx
-- seqkit --
カレントディレクトリの全fqファイルを分析。
seqkit stats *.fq
フルネームが重複したリードを除去。
seqkit rmdup -n input.fq
順番をバラバラにする。
seqkit shuffle input.fastq > shuffled.fastq
fastqをfastaに変換
seqkit fq2fa input.fastq > output.fa
duplicateしたIDをユニークな名前に修復。
seqkit rename input.fastq > renamed.fastq
カレントディレクトリのfastqファイルから、配列が一致する配列を抽出。
seqkit common -s *fq > common_seq.fq
リードを10%ランダムサンプリング。
seqkit sample -p 0.1 input.fastq > output.fastq
fastqを均等に10ファイルに分けて出力。
seqkit split -p 10 input.fastq
-- seqtk --
ペアリードを1つのファイルにまとめインターレースにする。
seqtk mergepe R1.fq R2.fq > paired.fq
ILLUMINA 1.3+フォーマットをfastaに変換し、同時にquality20以下を小文字にする。
seqtk seq -aQ64 -q20 input.fq > output.fa
クオリティを分析。
seqtk fqchk input.fq > output.txt
-- BBtools --
・様々な統計データを出力。
bbmap.sh in1=reads1.fq in2=reads2.fq bhist=bhist.txt qhist=qhist.txt aqhist=aqhist.txt lhist=lhist.txt ihist=ihist.txt ehist=ehist.txt qahist=qahist.txt indelhist=indelhist.txt mhist=mhist.txt gchist=gchist.txt idhist=idhist.txt scafstats=scafstats.txt
phread quality scoreを一般的なASCII+33からASCII +64に変換する。
reformat.sh in=reads.fq out=reads.fq qin=33 qout=64
fastq => fasta+qualへの変換。
reformat.sh in=reads.fq out=reads.fa qfout=reads.qual
インターレースのペアリードデータを2つのファイルに分ける。
reformat.sh in=interlace.fq out1=read1.fq out2=read2.fq
ペアリードデータをインターレースファイルにする。
reformat.sh in1=read1.fq in2=read2.fq out=reads.fq
ペアリードファイルがずれていないか検証。
reformat.sh in=reads.fq vint
ペアリードファイルの修復。
repair.sh -Xmx20g in1=broken1.fq in2=broken2 out1=fixed1.fq out2=fixed2.fq outs=singletons.fq repair
ペアリードのマージ。
bbmerge.sh in1=reads1.fq in2=reads2.fq out=merged.fq outu1=unmerged1.fq outu2=unmerged2.fq ihist=ihist.txt
オーバーラップするペアリードのエラー修復。
bbmerge.sh in=interlace.fq out=corrected.fq ecco mix
1や2がついていないペアリードファイルにペアを識別する1 or 2を入れる。
reformat.sh in1=pair1.fq in2=pair2.fq out1=renamed1.fq out2=renamed2.fq addslash=t int=f
70文字ごとに改行する。(foldコマンドと同じ)
reformat.sh in=reads.fa out=wrapped.fa fastawrap=70
小文字の塩基を大文字にする。
reformat.sh in=reads.fq out=out.fq tuc
名前が全てユニークか確認する。
reformat.sh in=reads.fq out=out.fq uniquenames
クオリテイスコアを特定の範囲に限定する(処理でクオリティがおかしくなってしまった時に使う)。
reformat.sh in=reads.fq out=out.fq mincalledquality=2 maxcalledquality=41
k-merのgraphを利用し、マージされなかったfastqを延長しマージを試みる。
bbmerge-auto.sh in=reads.fq out=merged.fq outu=unmerged.fq ihist=ihist.txt ecct extend2=20 iterations=5
リード長よりインサートサイズが短いペアリードからアダプター配列を見つける。
bbmerge.sh in1=reads1.fq in2=reads2.fq outa=adapters.fa
続けて見つかったアダプター(e.g., GATCGGAAGAGCACACGTCTGAACTCCAGTC )を除く。
bbmerge.sh in1=reads1.fq in2=read2.fq out=merged.fq adapter1=GATCGGAAGAGCACACGTCTGAACTCCAGTC
bbnorm.sh in1=reads1.fq in2=reads2.fq out=normalized.fq target=100 min=5
duplication sequencesの除去。
dedupe.sh in=X.fa out=Z.fa outd=duplicates.fa
99%以上同一の配列を除去。
dedupe.sh in=X.fa out=Y.fa minidentity=99
200bp以上同一の配列を1塩基のミスマッチまで許容して除去。
dedupe.sh in=X.fq pattern=cluster%.fq ac=f am=f s=1 mo=200 c pc csf=stats.txt outbest=best.fq fo c mcs=3 cc dot=graph.dot
PacBioの16S全長シーケンスデータのクラスタリング1。全長読めてない配列を除去。
reformat.sh in=reads_of_insert.fastq out=filtered.fq minlen=1420 maxlen=1640 maq=20 qin=33
1420-bp以下のリードと1640-bp以上のリード(アーティファクト)を除去する。また平均クオリティが20以下のリードも除去する。
PacBioの16S全長シーケンスデータのクラスタリング2。99%以上同一の配列をクラスタリング。
dedupe.sh in=filtered.fq csf=stats_e26.txt outbest=best_e26.fq qin=33 usejni=t am=f ac=f fo c rnc=f mcs=3 k=27 mo=1420 ow cc pto nam=4 e=26 pattern=cluster_%.fq dot=graph.dot
3'側のアダプタートリミング。
bbduk.sh in=reads.fq out=clean.fq ref=adapters.fa ktrim=r k=23 mink=11 hdist=1 tpe tbo
平均クオリティが10以下のリードをフィルタリング(除去)。
bbduk.sh in=reads.fq out=clean.fq maq=10
k-merフィルタリング。
bbduk.sh in=reads.fq out=unmatched.fq outm=matched.fq ref=phix.fa k=31 hdist=1 stats=stats.txt
指定ファイルに対して31-merで探索を行い、1塩基のミスマッチを許容して(hdist=1)マッチするリードがあれば、除去される。ここではイルミナのスパイク(バクテリオファージ、5.5-kb)に対して検索している。
k-merマスキング1。
bbduk.sh in=ecoli.fa out=ecoli_masked.fa ref=salmonella.fa k=25 ktrim=N
Quality recalibration
bbduk.sh in=reads.fq out=recalibrated.fq recalibrate sam=mapped.sam
samファイルを元にベースクオリティスコアを再校正したfastqを出力する。
Matching degenerate sequences such as primers
bbduk.sh in=reads.fq out=matching.fq literal=ACGTTNNNNNGTC copyundefined k=13 mm=f
degenerateな配列を探索する。例えばメタ16Sの可変性領域を網羅的に探す
ユニークなリード数を示したヒストグラムをかく。
bbcountunique.sh in=reads out=histogram.txt
指定リファレンスファイルに対して31-merで探索を行い、1塩基のミスマッチを許容して(hdist=1)マッチするリードがあれば、分離する。イルミナのスパイク(バクテリオファージ、5.5-kb)に対して検索をかけている。マッチしたペアリードとマッチしなかったペアリードをそれぞれペアの順番を同期したまま出力する(=4つのfastqが出力される)。
bbduk.sh in1=pair_1.fq in2=pair_2.fq \
out1=unmatched_1.fq out2=unmatched_2.fq \
outm1=matched_1.fq outm2=matched_2.fq \
ref=phix.fa k=31 hdist=1 stats=stats.txt
- outm=<file> (outmatch) Write reads here that contain kmers matching the database.
- ref=<file,file> Comma-delimited list of reference files.
- hammingdistance=0 (hdist) Maximum Hamming distance for ref kmers (subs only). Memory use is proportional to (3*K)^hdist.
連結したリファレンス配列を指定すれば、バテクリアの汚染除去などにも使用できる。ただし扱いには注意する。
-- fixfastq -- (github)
#build
git clone https://github.com/davidvi/cleanFastq.git
cd cleanFastq/
g++ -std=c++11 fixFastq.cpp -o fixFastq
sudo cp fixFastq /usr/local/bin/
#run
fixFastq broken.fastq > fixed.fastq
--fqtools--
basetab 塩基をカウント
fqtools basetab input.fq
qualtab クオリティスコアをカウント
fqtools qualtab input.fq
count リード数をカウント
fqtools count input.fq
sequence 配列だけ表示(配列のみのcat)
fqtools sequence input.fq > output
header ヘッダーだけ表示(ヘッダーのみのcat)
fqtools header input.fq > output
quality クオリティスコアだけ表示(クオリティスコアのみのcat)
fqtools quality input.fq > output
validate 配列ATGCATGCを探す(grep、agrepに近い)。
fqtools find -s ATGCATGC input.fq
trim トリミング
#先頭10bp
fqtools trim -s 10 input.fq > trimmmed.fq
#100bpになるまでカット
fqtools trim -l 100 input.fq > trimmmed.fq
-- ngs crumbs--
ペアリードからインターレースのfastqを作る。
interleave_pairs R1.fq R2.fq > merged.fq
インターレースからペアリードに分離する。
interleave_pairs merged.fq -o R1.fq R2.fq
ペアじゃないリードを除く。
pair_matcher interelace.fq -o output.fq -p orphan.fq
fastqのフォーマットを変更する(fasta、fastq、fastq-illumina)。
convert_format input.fastq -f fasta -o output.fa
--fastqutils--
properpairs(リンク)ペアエンドでペアになっていないリードを除く。
fastqutils properpairs pair1.fq pair2.fq output_pair1.fq output_pair2.fq
revcomp(リンク)逆相補鎖(reverse complementary)に変換。
fastqutils revcomp input.fq > output.fq
sort(リンク)nameやポジションでソートする。
fastqutils sort input.fq > sort.fq
split(リンク)指定数に分割する。
#3つに分割する。
fastqutils split input.fq output 3
convertqual(リンク)イルミナのクオリティ値をsanger scaleに変換。
fastqutils convertqual input.fq > output.fq
csencode(リンク)color spaceをATGCに変換。
fastqutils csencode input.fq > output.fq
fromfasta(リンク)qualとfastaからfastqを作成。
fastqutils fromfasta input.csfasta input.qual > output.fq
fromqseq(リンク)illumina export.txtからfastqを作成。
fastqutils fromqseq export.txt
stats(リンク)statistics
fastqutils stats input.fq > summary
出力。全部位の情報も出る。
$ fastqutils stats forward.fq
Done! (0:58)
Space: basespace
Pairing: Fragmented
Quality scale: Illumina
Number of reads: 224924
Length distribution
Mean: 230.287452651
StdDev: 49.7016332922
Min: 20
25 percentile: 204
Median: 235
75 percentile: 262
Max: 301
Total: 224924
Quality distribution
pos mean stdev min 25pct 50pct 75pct max count
1 33.9262595366 0.436774185711 30 34 34 34 38 224924
2 33.8028489623 1.54960128452 10 34 34 34 38 224924
3 33.8104426384 1.54959912975 10 34 34 34 38 224924
4 33.8467082214 1.44051755323 10 34 34 34 38 224924
5 33.8715655066 1.56917725475 10 34 34 34 38 224924
6 37.6461204674 1.84245487446 10 38 38 38 38 224924
7 37.622272412 1.94525249168 10 38 38 38 38 224924
8 37.6478232647 1.86618349415 10 38 38 38 38 224924
9 37.6512333055 1.85726711398 10 38 38 38 38 224924
10 37.6626060358 1.79494590177 10 38 38 38 38 224924
11 37.6324714126 1.89571282258 9 38 38 38 38 224924
12 37.6014342622 1.99703097792 10 38 38 38 38 224924
13 37.6435818321 1.8427123548 10 38 38 38 38 224924
14 37.6336940478 1.88442047255 10 38 38 38 38 224924
15 37.6378332237 1.85404372502 9 38 38 38 38 224924
16 37.6625215628 1.78640763134 9 38 38 38 38 224924
17 37.6524514947 1.80913909303 10 38 38 38 38 224924
18 37.6461649268 1.81338343748 9 38 38 38 38 224924
sff2fastq
Roche 454の.sff => fastq変換
git clone git://github.com/indraniel/sff2fastq.git;
cd sff2fastq
make
ln -s sff2fastq /usr/local/bin/
#実行
cat input.sff | sff2fastq > file.fastq
Ming Tangさんの素晴らしいワンライナー集(bioinformatics-one-liners)
追記
--seqmajic--(リンク)
fastaフォーマットをphyフォーマットに変換(アライメント実行後のfastaを使う)
seqmagick convert input.fa output.phy
stockholm format(wiki)に変換(アライメント実行後のfastaを使う)
seqmagick convert input.fa output.sto
multi-fastaから先頭5配列を出力
seqmagick convert --head 5 input.fa firs_five_seq.fa
multi-fastaからラスト5配列を出力。ただし、5配列のうち、100bp以下は出力されない(--head 5 => --min-length 100 )。
seqmagick convert --tail 5 --min-length 100 input.fa last_five_seq.fa
multi-fastaの100bp以上の配列を対象に、最後から5配列を出力(--min-length 100 => --head 5 )
seqmagick convert --min-length 100 --tail 5 input.fa last_five_seq.fa
multi-fastaの100bp以上の配列を対象に、それぞれの配列の1bpから5bpまでのポジションを抽出
seqmagick convert --min-length 100 --cut 1:5 input.fa pos1-5.fa
DNA <=> RNA変換
#DNA => RNA
seqmagick convert --transcribe dna2rna input_dna.fa output_rna.fa
#RNA => DNA
seqmagick convert --transcribe rna2dna input_rna.fa output_dna.fa
- --transcribe {dna2rna,rna2dna}
- --translate {dna2protein,rna2protein,dna2proteinstop,rna2proteinstop}
duplicationを除く(配列をチェックしている。ヘッダーが違っても配列が100%同一だと除かれる)
seqmagick convert --deduplicate-sequences input.fa output.fa
追記
fastaからpseudo-fastqを作成。
UCSCのkent utilitiesのfaToFastqを使う。
git clone git://github.com/ENCODE-DCC/kentUtils.git
cd kentUtils
make
#rsyncでbinのパスを通す
sudo rsync -a -P ./bin/ /usr/local/bin/kentUtils/
faToFastqだけならcondaでも入る(リンク)。
クオリティ"<"のfastqに見せかける。
faToFastq -qual=\< input.fa output.fq
>head output.fq
$ head output.fq
@DEAMERNANOPORE_20161206_FNFAB49164_MN16450_sequencing_run_MA_821_R9_4_NA12878_12_06_16_71094_ch374_read7088_strand.fast5
ATTCGTTTGGCTGGGTTCAGGATGCGTGTTGCTCGCTCCCGTTCCATTCATCACTGCTAAATTCTGTCCTCCCTTCATCACTCTTTTCGTTTCCATTCCCGTTTCCACTCCGATTCCCGTTCCTTCCTGCGTTCCATCACTCCGCTCCGTTGCTCCATTGCTCCGTTCCGTTAGGCTCCCGTTTCGTTCCACTCATCCGTTTCTGATCCATCACTCTGCTCCATTGCCGAGTCCACTCCCATTCAGATTCAGTTCTGCCCTCCGTTCCATTTCCGCTCATCATTCCAATCCGCCGCATGGGTTCCATTCCACTCAGGTTCCGTTCCGTTGCTCCGTTCCATTGCCTTCCGTTCGATCGGTTAGTTCCTCCCGTTTCCAGGTTTCCATCCACTCCGTTTCTGTTTCATCTGCTCCTTTCCTCCGTTCCACTTCATTCATTCCTGTCTCTCATCACTACATCCCAGTCACTCCTTTCAGTTCCTCCACTCCACTCCCGATTTCCTCTCCAGTCGGCTCCATCCGTTCCATTCCACTCATCCG
+
<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<<
--BBSketch--(リンク)データに含まれる生物種を超高速推定(fastqも使えます)
sendsketch.sh in=contigs.fa
sketchファイルで結果は問い合わされ、 数秒で結果が返ってくる。
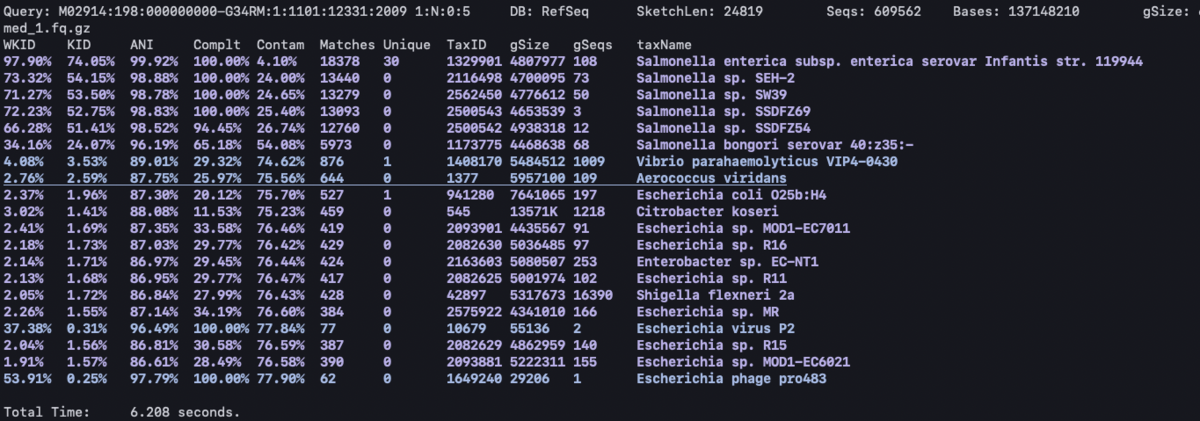
- KID (or kmer identity) is simply the percent of the kmers that matched
- WKID is the weighted kmer identity, which has been adjusted to compensate for genome size.
- matches is the number of matching kmers between the query and reference sketch
--fastp--(リンク)
fastq出力を指定しなければレポートファイル(htmlとjson)のみ出力される。
fastp -i pair1.fq -I pair2.fq
- -I read2 input file name (string [=])
- -O read2 output file name (string [=])
- -3 enable per read cutting by quality in tail (3'), default is disabled (WARNING: this will interfere deduplication for SE data)
- -w worker thread number, default is 3 (int [=3])
出力の一部。

--fastqp--(リンク)
fastqの分析。inputファイルだけ指定すればランできる。
fastqp input.fastq.gz
出力ディレクトリはgz圧縮される。解凍して中を開く。複数のmetricsで分析が行われている。
出力の一部。

各コマンドの詳細はTopのツールのリンクで確認してください。
引用
BBMap (aligner for DNA/RNAseq) is now open-source and available for download.
SeqKit: A Cross-Platform and Ultrafast Toolkit for FASTA/Q File Manipulation
Wei Shen, Shuai Le, Yan Li , Fuquan Hu
PLOS ONE. 2016. doi:10.1371/journal.pone.0163962.
GNU Parallel 20150322
http://dx.doi.org/10.5281/zenodo.16303
*1
Question: Multiline Fasta To Single Line Fasta
https://www.biostars.org/p/9262/