Microbiome Community Detector (MiCoDe) は、ベイジアン加重確率的ブロックモデルを用いて微生物分類群のネットワークをコミュニティにクラスタリングする、無料の使いやすいウェブツールである。MiCoDeはデータを自動的にフィルタリングし、高次元性、構成性、ゼロインフレ、非線形性といったマイクロバイオーム高スループットシーケンシングデータの課題を考慮する。MiCoDeは厳密な統計的非教師あり学習モデルに基づいているものの、当ウェブツールはあらゆる研究者が容易に利用できる。ユーザーはtaxonomic abundance dataを含むCSVファイルをアップロードするだけでよく、行はサンプルに対応し、列は分類群に対応する。その後、データ変換、ネットワーク推定、コミュニティ数に関するいくつかの選択を行い、オンライン解析を実行する。選択に迷った場合は、推奨設定であるデフォルト設定を選択できる。本論文では、MiCoDeの動機、方法論、実装、結果について議論する。また、ユーザーによるMiCoDeの適応方法や将来的な進化の可能性についても考察する。当ソフトウェアは、マイクロバイオームコミュニティ検出のための貴重なツールである。
MiCoDeはhttps://lce.biohpc.swmed.edu/micode/でオンライン無料提供されており、インストール不要かつブラウザ非依存である。ユーザーはGitHub(https://github.com/klutz920/MiCoDe)で公開されているRコードを使用してローカル環境でも利用できる。
https://lce.biohpc.swmed.edu/micode/にアクセスする。
HPより: Microbiome Community Detector(MiCoDe)は、マイクロバイオームデータから推定された共存ネットワークのコミュニティ構造を調査するために使用できるウェブツールである。MiCoDeは、重み付きエッジを持つグラフで表される完全連結ネットワークに、ベイズ非パラメトリック加重確率ブロックモデル(WSBM)またはベイズ非パラメトリック加重確率無限ブロックモデル(WSIBM)のいずれかを適合させる。
Analysis
サンプルと分類群に対応する行と列を持つ分類学的アバンダンステーブル(CSV形式)をアップロードする。
example1

行はサンプル、列は分類群
複数のオプションが用意されている。データの前処理(MCLR、CLR、構成法)、分類群間関連性の測定法(SPR、スピアマン、ピアソン相関係数)、コミュニティ数(WSIBM使用時は「自動」を選択、WSBM使用時は2~20の範囲で事前指定)など選択できる(論文Figure1より)。

デフォルトの設定は、特にアバンダンスデータが非線形かつゼロ過剰性が高い場合に推奨される設定であるため選ばれている。MCLR変換は組成性の問題も除去する。ベイズモデルは高次元データの処理にも適している(論文Methodより)。
3つのExample.csvをダウンロードできる。

3つについては論文の結果のセクションでそれぞれ説明されているが、実データとなっている、
検証コードを入力してサブミットする。テスト時は1分程度で結果が表示された。
出力例
MiCoDeは、ノードおよび重み付きエッジからなる視覚的ネットワークとして視覚化され、K個の異なるコミュニティ別にクラスタリングされた分類群間の相互作用が示される。図でノードは分類群に対応し、エッジは2つの分類群間の相関が指定された閾値を超えることを示している。ノードの大きさは、そのノードに接続されるエッジの数が増えるにつれて大きくなる。エッジの数は、相関閾値が低くなるにつれて増加する。
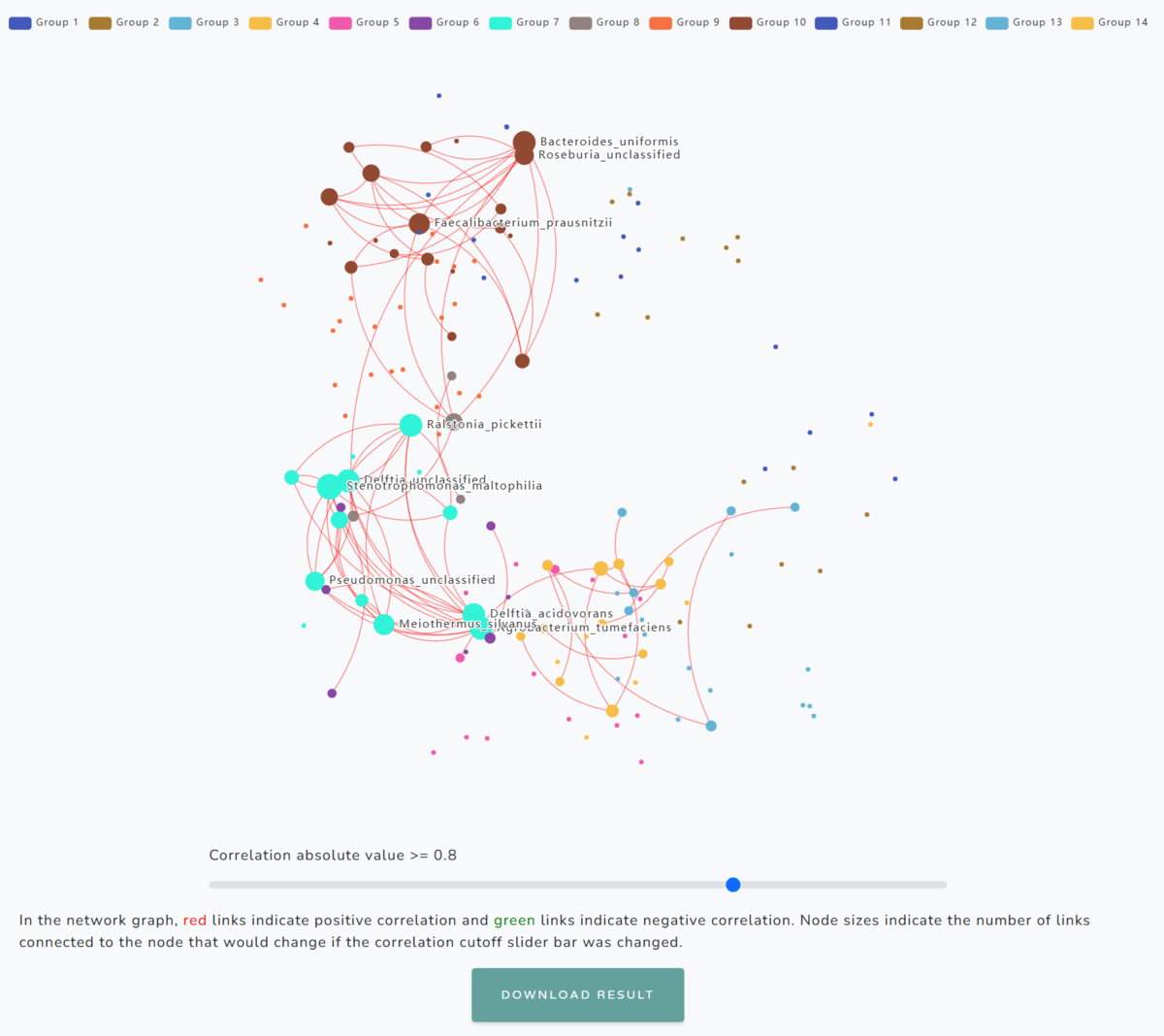
上の注釈にあるように、同じ色は同じコミュニティ内のノードを表している(同一コミュニティに属する分類群を識別できるようにするため)。
下のバーから、相関の大きさに基づいてエッジを動的にフィルタリングできる。
Correlation absolute value >= 0.8 => >= 0.5

赤いリンクは正の相関を示し、緑のリンクは負の相関を示している。
ノードまたはエッジにカーソルを合わせると、2つの分類群間の相関係数や特定の分類群に接続されたエッジ数などが表示される。

ズームイン・ズームアウト、スクロールも可能。

凡例内のコミュニティ名をクリックすると、それらのコミュニティをネットワークから除外できる。Group 10を除外した。

再度クリックするとネットワークに再表示される。
結果はCSVとしてダウンロードできる。クラスタリング結果と相関行列のCSVとなっている。

cluster_result.csv

分類群はアルファベット順に並んでいる。群集インデックスでソートすることで、同じ群集に割り当てられた分類群を確認でき、生物学的な意味を推察するのに役立つ(論文より)。
corr_matrix.csv

すべての分類群ペア間の相関係数を含んでいる。
結果の画面中央に表示されるリンクを使用すると、分析を再実行せずに後から結果にアクセスできる。

論文より
- マイクロバイオームネットワークにおいて、ノードは分類群を表し、存在するエッジは任意の2分類群間の数値的または非数値的な関連を表す。エッジが相関係数などの数値を持つグラフは重み付きグラフ(weighted graph)と呼ばれる。群集(コミュニティ)とは、グラフ内のノードのうち、同一群集内で密に結合し、他の群集内のノードとは疎に結合しているノード群を指す。
- MiCoDeでは、分類群存在量データを解析に適した形に補正するために、変換手法を用いた前処理や、関連指標を用いた微生物ネットワークの重み付きグラフ推定を行うオプションが利用可能である。MiCoDeのインターフェースは無料で、ユーザーフレンドリーであり、インストールやプログラミングを必要とせず、特定のブラウザにも依存しない。現在、群集検出のためのウェブツールは他に存在しない。MiCoDeは現在、プログラミングやネットワーク解析の専門知識を持たない研究者を含むすべての研究者が利用可能な唯一の群集検出ウェブツールである。
- MiCoDeの目的は、n個のサンプルからシーケンスされたp個の分類群を、相互排他的なK個の群集(コミュニティ)にクラスタリングすることである。MiCoDeはこの目的を3つのステップで達成する。
引用
MiCoDe: a web tool for performing microbiome community detection using a Bayesian weighted stochastic block model Open Access
Kevin C Lutz, Shengjie Yang, Tejasv Bedi, Michael L Neugent, Nikita Madhavaram, Bo Yao, Xiaowei Zhan, Nicole J De Nisco, Qiwei Li
Bioinformatics, Volume 41, Issue 7, July 2025
