ゲノム、トランスクリプトーム、プロテオームデータの解釈に不可欠なエンリッチメント解析は、メタボロミクス分野へも拡大している。さらに、メタ解析やマルチオミクス研究に見られるように、異なる研究やオミクスプラットフォームからのデータを統合する統合エンリッチメント解析への需要が高まっている。こうした増大するニーズに対応するため、WebGestaltをアップデートし、代謝物および複数の分析対象リストに対するエンリッチメント解析機能を追加した。また、これらの更新に対応するため、解析速度を大幅に向上させ、ユーザーインターフェースを一新し、新たなパスウェイ可視化機能を導入した。特に、Rustバックエンドの採用により、遺伝子セットエンリッチメント解析時間は270.64秒から12.41秒へ95%短縮、ネットワークトポロジーベース解析は159.59秒から17.31秒へ89%短縮された。この性能向上はRパッケージと新たに導入したPythonパッケージの両方で利用できる。さらに、WebGestaltデータベース内のデータを各ソースの最新状態に反映させるとともに、パスウェイ・ネットワーク・遺伝子シグネチャのコレクションを拡充した。2024年版WebGestaltは、メタボロミクスへの新規対応、マルチオミクス解析機能の効率化、顕著な性能向上を実現し、大きな飛躍を遂げている。これらの更新内容の詳細はhttps://www.webgestalt.orgで確認できる。
R version of WebGestalt
https://www.webgestalt.org/にアクセスする。
上のメニューからは2019年バージョンにもアクセスできる。
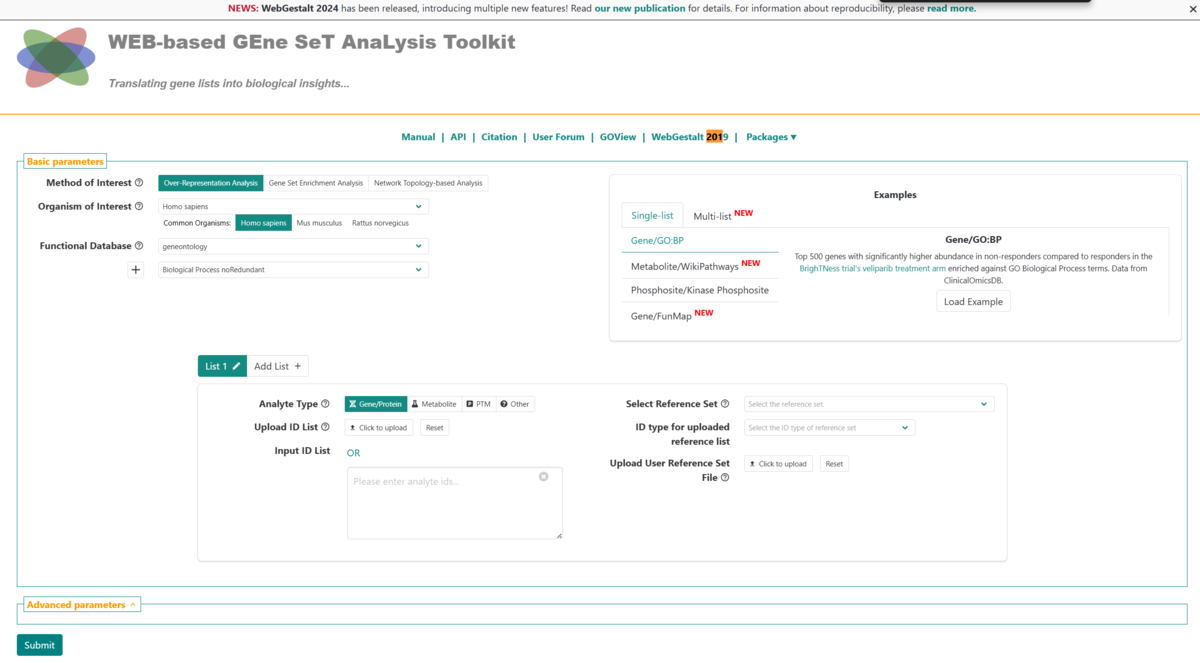
生物を選び、方法を選択する。Over Representation Analysis(ORA)、Gene Set EnrichmentAnalysis (GSEA) 、Network Topology-based Analysis (NTA) が選べる。
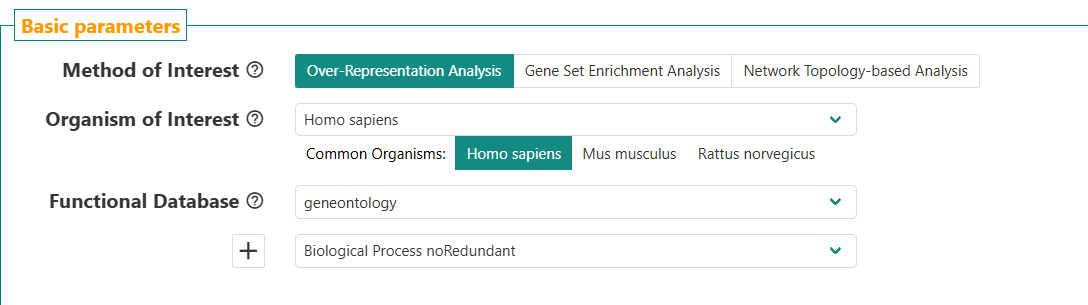
Functional Database を選択する。Gene Ontology (GO)、疾患、表現型、なども選べる。otherを選ぶと、カスタムデータベースをアップロードして使用することができる。

Functional Databaseの種類を選択後、続いてKEGGやReactome、WikiPathwaysなどデータベースを選択する。例えばpathwayなら以下からデータベースを択できる。

+マークを押すことで、複数のFunctional Databaseを指定できる(ユーザーカスタムデータベースは1個まで)。

下のパネルで、ID typeとデータを指定する。下の画像では見えていないが、右端のパネルからデモデータを読み込むこともできる。
Single list (demo)
BrighTNess試験のベリパリブ治療群において、反応者と比較して非反応者で有意に高発現した上位500遺伝子が貼り付けられている。

Multi-list1 (demo)
マルチオミクスの場合、タブごとにオミクスデータを貼り付ける。追加するには、Add Listをクリックする。デモデータ1では、Liu、Hugolo、Gideという3つのメラノーマ関連研究データセットが読み込まれる。

画像のように、ボタンがStudy名となっている。それぞれのタブに3つのソースからGene IDがそれぞれ貼り付けられている。
Multi-list2 (demo)
より複雑なマルチオミクスの例。Multi-omics/WikiPathwaysのデモデータ。DMSO処理対照群と比較し、FCCP処理Hela細胞において有意に高発現したRNA、タンパク質、代謝物のマルチオミクス解析(FDR<0.01)、WikiPathwaysに基づくエンリッチメント解析(Quirosらによるデータ)が読み込まれている。

左端のボタンからそれぞれ、有意に高発現したRNA、有意に高発現したタンパク質、代謝物のマルチオミクス解析(FDR<0.01)の遺伝子もしくはタンパク質リストとなる。
Advanced parameters
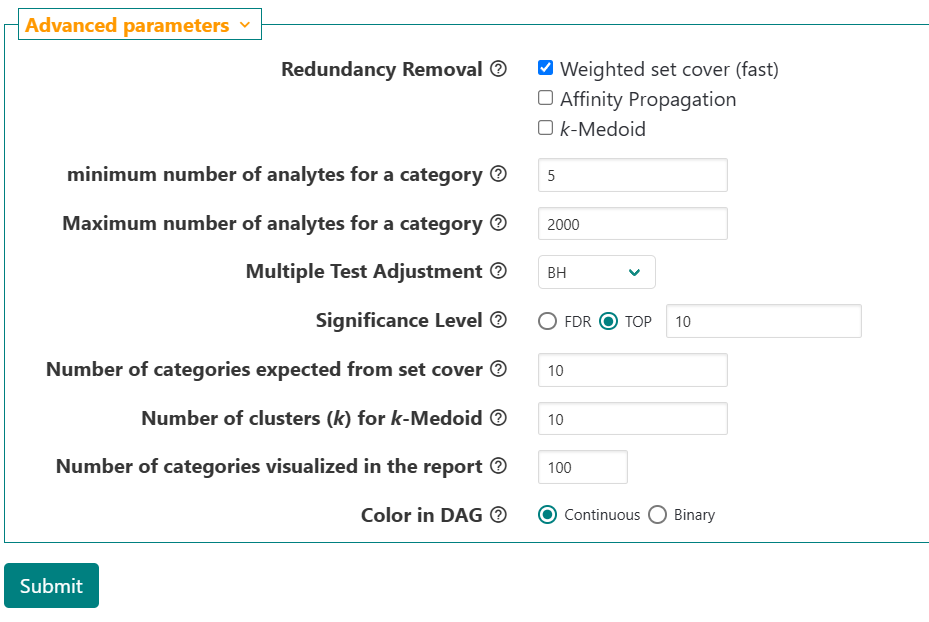
最後にサブミットする。デモデータだと数十秒で結果が表示された。
出力例


グラフは棒グラフから表、Volcano plotに切り替え可能。

MultiOmicsだと、オミクスごとにまとめられる。切り替えるにはタブをクリックする。

論文とHPより
- pathway analysisは、メタボロミクスデータセットの機能的解釈を行う上で不可欠なツールとなっている。しかしこの解析には、代謝物同定の標準化の欠如、個々のpathway データベースにおける代謝物の不完全な網羅、そして広く用いられている ORA手法における適切なバックグラウンド集合の選択といった重大な課題が存在している。これらの課題に対処するために、WebGestalt 2024 では、多様な代謝物IDタイプへの対応、複数のpathwayデータベースの利用、そして ORA および GSEAの両手法の採用によって、ヒトメタボロミクスデータにおける経路解析を強化している。
- WebGestalt は、さまざまなデータソースから取得したIDマッピング情報、遺伝子セット、およびネットワークを統合し、自身のデータベースに組み込んでいる。この統合により、ユーザーは入力として異なる種類のIDを使用しつつ、幅広い知識領域にわたるエンリッチメント解析を実行できる。
- 2024年の更新では、既存データが各ソースの最新状態を反映するよう更新された。さらに、新しいデータが追加され、利用可能なアナライト/IDタイプ、経路、ネットワーク、遺伝子シグネチャの種類が拡張された。これにより、エンリッチメント解析に利用可能なセットの総数は 663,247 に達した(論文補足表S1)。
- 今回の更新では特にメタボロミクス対応に重点が置かれている。メタボロミクス解析における最大の課題の一つは、標準化の欠如である。メタボロミクス研究で使用されるIDタイプは多岐にわたり、解析の大きな障壁となっている。柔軟な代謝物ID入力を可能にするため、WebGestaltでは公開データベース RaMP-DB (7) に由来する16種類の代謝物IDタイプへの対応を導入した。
- メタボロミクスを用いたORAには、独自のリファレンスセットを用意することを強く推奨する。このリファレンスセットには、検出された全ての代謝物を包含する必要がある(遺伝子リスト選択のパネルでSelect Reference Setから指定する)。
コメント
2019アップデートを一度紹介しているので、今回はより簡単に紹介しました。
引用
WebGestalt 2024: faster gene set analysis and new support for metabolomics and multi-omics Open Access
John M Elizarraras, Yuxing Liao, Zhiao Shi, Qian Zhu, Alexander R Pico, Bing Zhang
Nucleic Acids Research, Volume 52, Issue W1, 5 July 2024, Pages W415–W421
関連