ヒト遺伝子のバリアントレパートリーの多くは、一塩基多型(SNV)とsmall indelで構成されているが、構造多型(SV)は依然として私たちの変異DNAの主要な部分を占めている。SVの検出は、各カテゴリーのSVを検出するために異なる技術(アレイCGH、SNPアレイ、核型分析、オプティカルゲノムマッピングなど)を使用する必要があるか、適切な解像度(全ゲノムシーケンシング)を得る必要があるため、しばしば複雑な課題であった。パンゲノム解析の爆発的な増加により、ヒト遺伝学者はSVを蓄積しているが、その解釈は依然として時間と労力を要する困難な作業である。AnnotSVウェブサーバー(https://www.lbgi.fr/AnnotSV/)は、以下の目的で効率的なツールとなることを目指している:(i) ヒト疾患の文脈におけるSVの潜在的な病原性を注釈付け・解釈すること、(ii) 特定された全SVから潜在的な偽陽性バリアントを識別すること、(iii) 患者の変異レパートリーを可視化すること。AnnotSVウェブサーバーの最新開発内容は以下の通りである:(i) アノテーションソースとランキングの更新、(ii) 多様な利用(解析、パイプライン)を可能にする3つの新規出力フォーマット、(iii) インタラクティブなCircosビューを含む2つの新規ユーザーインターフェース。
Tutorials (YouTube channel)
https://www.lbgi.fr/AnnotSV/tutorials
3 - How to run AnnotSV online?
5 - Case study: Known pathogenic SV and RE overlapped
https://www.lbgi.fr/AnnotSV/ にアクセスする。
ここでは左上のAnnotSVを見ていく。
AnnotSV
AnnotSVはSVの潜在的な病原性を解釈したり、SVの潜在的な偽陽性を除外するのに有用なアノテーションを提供するAnnotSV webサーバーの中核のツール (PubMed)。
AnnotSV使用するにはSVのVCFをアップロードする。ここではexampleデータを指定。

AnnotSVは入力データとアノテーション特徴との間でゲノム上の重複領域を特定する。重複の判定基準は、SVとアノテーションの間で相互(reciprocal)または非相互(non-reciprocal)として設定できる。reciprocalとは、SVの長さのx%以上がアノテーション領域と重なり、かつアノテーション領域のy%以上がSVと重なっていることを意味していると思われる。non-reciprocalはSVが少しでもアノテーション領域と重複していればアノテーションされる。
ヒトゲノムのGRCh37またはGRCh38ビルドのいずれかを用いて実行可能です。
、アノテーション処理を実行します。
AnnotSVは複数のゲノムリソース(RefSeq, OMIM, DGV, gnomAD, ClinVar, DECIPHER, Ensembl Regulatoryなど)を統合していて、各SVがどの遺伝子や既知SVバリアントに関係するか自動でアノテーションを付与してランク付けする。また、DGV(MacDonald et al., 2014)から得られた重複バリアントに関連する対立遺伝子頻度も報告する。
結果はTSVCFやアノテーションが付与されたVCF、またHTMLの表としてダウンロードできる。

また、その場でHTMLの表を開くことも可能。
表はknotAnnotSVで視覚化されている。
Expanded mode
色はSVのタイプを表す。また、色の濃さは機能喪失に対する影響の大きさを表し、遺伝子全体の重なり度は3’ 側が重なっている場合は右半分が色づくなどの意味を持っている。詳細はレポジトリ (Github)で説明されている。
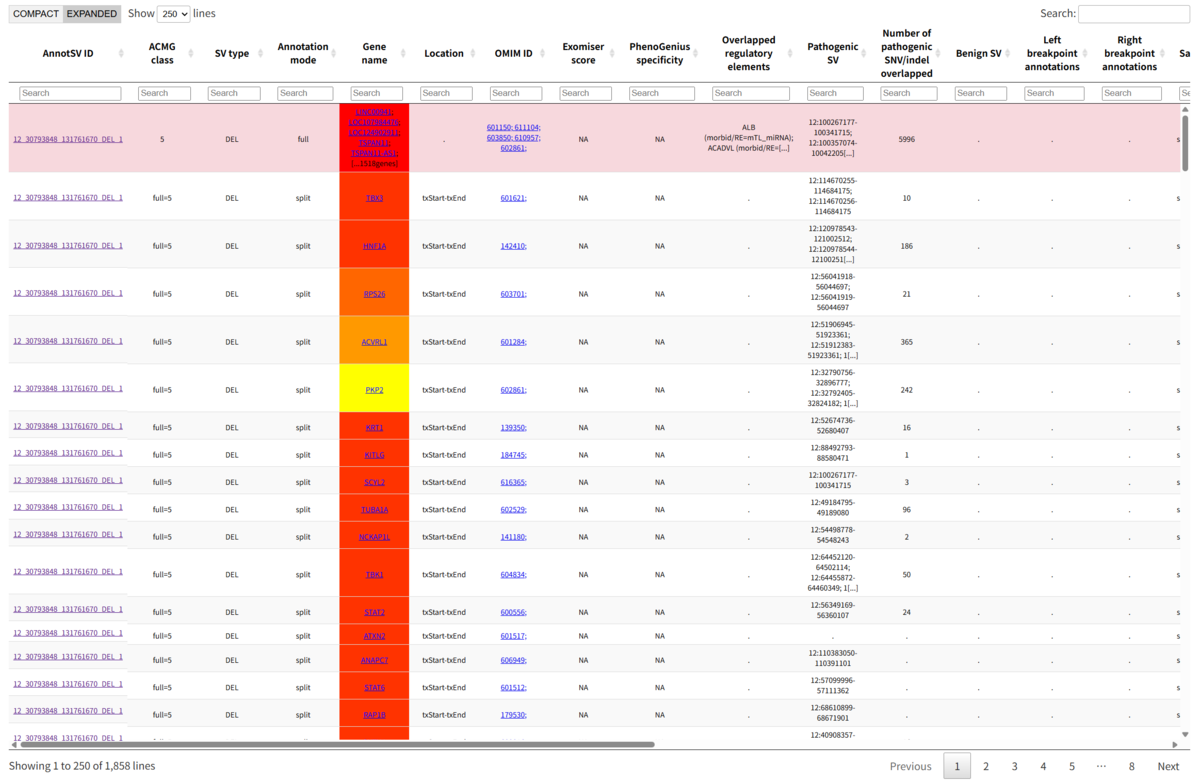
表の1列目はAnnotSVの固有のID、2列目はACMG(American College of Medical Genetics)によってランク付けされた病的評価クラスで、1-5(あるいはNA)となっている。3列目はSVの種類でDELやINなど、4列目はAnnotation modeでfull (SV全長ベース)かsplit(各遺伝子ごとのアノテーション)、5列目はSVが発生している遺伝子名、6列目はLocation、7列目はOMIM(Online Mendelian Inheritance in Man)IDで疾患との関連付けに使われる。8列目はExomiser scoreでExomiserによる優先度スコア(ヒト表現型に基づく病的可能性評価、表ではNA)、9列目はPhenoGenius specificityで、PhenoGeniusで計算された表現型特異性スコア(ヒト表現型とSV/遺伝子の関連度を示す、表ではNA)。10列目はOverlapped regulatory elementsでこれはSVと重なった調節領域(enhancer, promoterなど)を示している。11列目はPathogenic SVで病原性SVが重なっている場合の情報を表す。12列目はNumber of pathogenic SNV/indel overlappedでSVが重なる領域に存在する既知病原性SNVやindel数を表す。14-15列目はLeft breakpoint annotations / Right breakpoint annotationsでSVの左端と右端のブレイクポイントに重なるアノテーション情報。16列目はそのSVが検出されたSamples_ID、さらに右の列にはクオリティ値や各サンプルごとのhomo/heteroかの情報が示されている。
Compact mode

上の画像では、上の5つがピンクでこれはDELを表す。その下の7個続く水色はDUPを表す。一番下の濃いピンクはINSを表す。
SV rankingについては以下のように説明されている。
https://www.lbgi.fr/AnnotSV/ranking
 0.99 以上 クラス 5 Pathogenic(病原性)
0.99 以上 クラス 5 Pathogenic(病原性)
0.90 ~ 0.98 クラス 4 Likely pathogenic(病原性の可能性が高い)
−0.89 ~ 0.89 クラス 3 Variant of uncertain significance(意義不明変異)
−0.98 ~ −0.90 クラス 2 Likely benign(良性の可能性が高い)
−0.99 以下 クラス 1 Benign(良性)
バリアントはcirocs plotとして視覚化することもできる(link)。
https://github.com/bioinfo-chru-strasbourg/vcf2circos
引用
The AnnotSV webserver in 2023: updated visualization and ranking
Véronique Geoffroy, Jean-Baptiste Lamouche, Thomas Guignard, Samuel Nicaise, Arnaud Kress, Sophie Scheidecker, Antony Le Béchec, Jean Muller Author Notes
Nucleic Acids Research, Volume 51, Issue W1, 5 July 2023, Pages W39–W45
AnnotSV and knotAnnotSV: a web server for human structural variations annotations, ranking and analysis
Geoffroy V, Guignard T, Kress A, Gaillard JB, Solli-Nowlan T, Schalk A, Gatinois V, Dollfus H, Scheidecker S, Muller J.
NAR. 2021 May 22.
AnnotSV: an integrated tool for structural variations annotation
Véronique Geoffroy, Yvan Herenger, Arnaud Kress, Corinne Stoetzel, Amélie Piton, Hélène Dollfus, Jean Muller
Bioinformatics, Volume 34, Issue 20, October 2018, Pages 3572–3574
