本著者らは最近、腸内微生物叢の状態から疾患発生の可能性を判定し健康状態を評価する指標として、糞便メタゲノムに基づく「腸内微生物叢ウェルネス指数(GMWI)」を導入した。このウェルネス指数の算出は、健康に有益な種と健康に有害な種の相対的豊かさに依存する。心強いことに、GMWIは既に症例群と対照群の腸内マイクロバイオーム差異に焦点を当てた様々な研究で活用されている。本稿では、糞便ショットガンメタゲノム分類プロファイルからGMWI、健康優位種/健康不足種の相対的アバンダンス、およびα多様性を計算するユーザーフレンドリーなブラウザアプリケーション「GMWI-webtool」を紹介する。インタラクティブなオンラインツールでは、結果を可視化してメタゲノム統合リファレンスデータセットの結果と並べて比較できるほか、.csv形式のデータや高解像度図をエクスポートできる。GMWI-webtoolはhttps://gmwi-webtool.github.io/で無料利用できる。
https://gmwi-webtool.github.io/にアクセスする。
メタゲノムに対してMetaPhlAn2を実行した後、その出力ファイル(.txt)をブラウザにアップロードするか、出力データをペーストする。ここではexampleを使用。1検体や複数検体どちらにも対応している。

複数検体のデータをアップロードした場合、どのサンプルを可視化するか選択する。ここで指定したサンプルだけが、GMWI や α多様性(Richness, Evenness, Shannon, Inverse Simpson)などの計算に使われる。下の画像のように、入力テキストの1行目の名前が選択肢に表示されている。

次に、アップロードしたサンプルを比較するリファレンス集団(比較対象データセット)を指定する。GMWIではHealthyな集団とNonhealthyな集団を加えてデータベースを作っているので、比較対象を選ぶ。デフォルトはAllになっている。

最初の2つの図にのみ適用される。
出力例
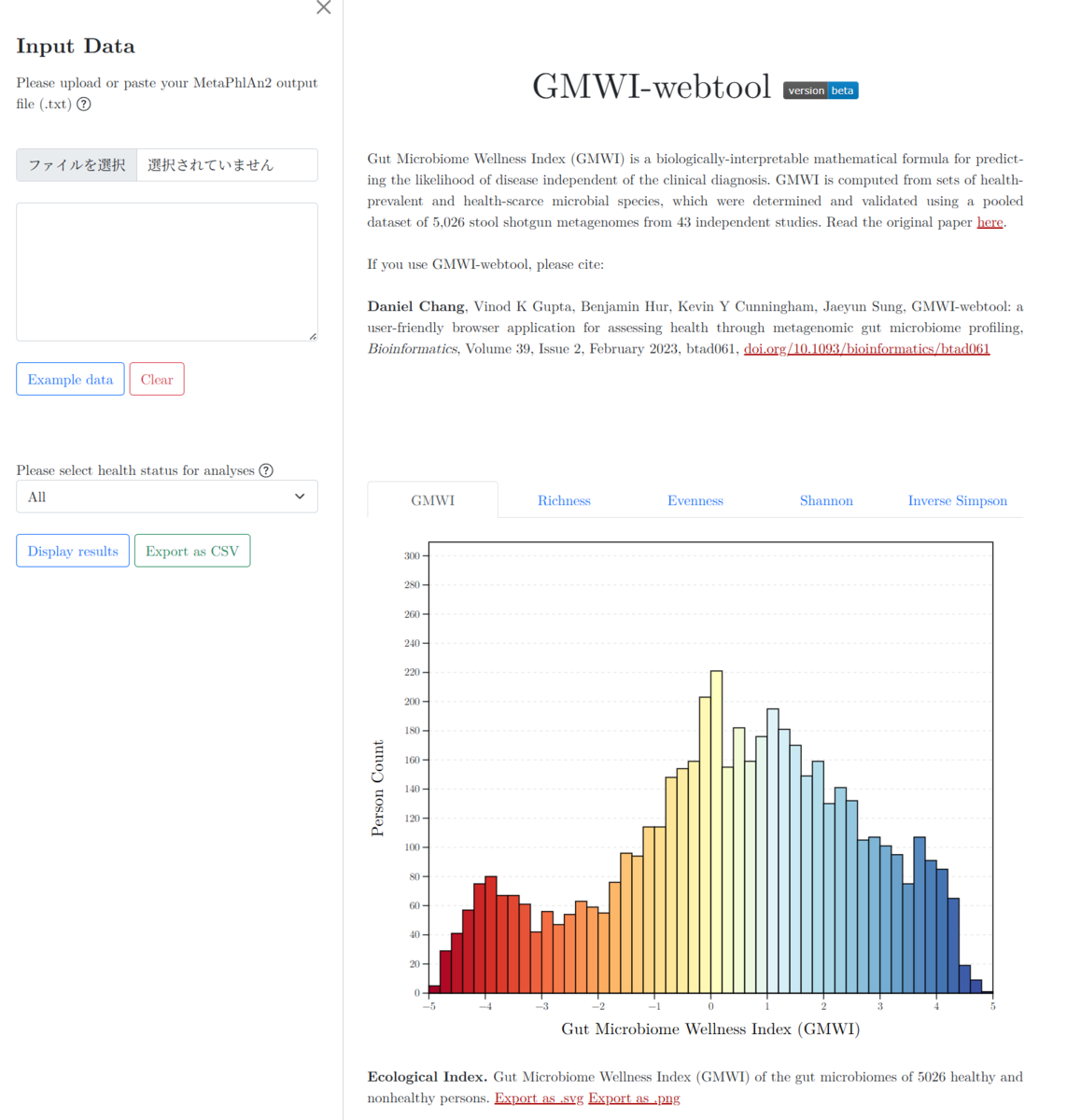
以下の可視化結果を得ることができる:(i)GMWI、(ii)健康優勢/稀少種の相対存在量、(iii)α多様性指標(種多様度、均等度、Shannon指数、逆Simpson指数)、(iv)各分類群の相対存在量。それぞれのグフにおいて、5026名の健康な人と健康でない人の腸内微生物叢の分布の上で、入力サンプルの位置がハイライトされる。
GMWI

Richness

Evenness

Shannon Diversity Index

Inverse Simpson Diversity

論文より
- 糞便メタゲノムに基づく健康モニタリング指標である Gut Microbiome Wellness Index(GMWI)[以前は Gut Microbiome Health Index(GMHI)]は。簡潔に言えば、臨床診断とは独立して疾患の可能性を予測するための生物学的に解釈可能な数理式である。GMWIは、健康および疾患状態に関連する2つの微生物種集合(それぞれ「健康優勢種(health-prevalent)」および「健康稀少種(health-scarce)」)に基づいて算出される。これらは、34件の独立した既報研究から得られた合計4347件の糞便ショットガンメタゲノムサンプルの統合データセットを用いて決定された。概念実証として、外部検証セット(679人の糞便メタゲノム)において、臨床的に疾患が診断されているか否かを予測する際に73.7%のバランス精度を達成し、ランダムフォレスト分類器や生態学的指標に基づく方法を上回っている。
- 2020年の初登場以降、GMWIは環境要因(Gacesa et al., 2022)や遺伝的/社会経済的要因(Xu et al., 2022)がヒト腸内マイクロバイオームに及ぼす影響を調べる研究において利用されてきた。家庭用ペットへの興味深い応用として、このGMWIの枠組みはネコの腸内健康評価にも適用された(Sung et al., 2022)
-
GMWIの計算にはRプログラミングおよび外部ライブラリの操作に習熟している必要があり、計算パイプラインの扱いに不慣れな研究者にとっては障壁となる可能性があった。さらに、GMWIスコアのデータベースが現時点で存在しないため、新たに得られたスコアを基準集団と比較して文脈化することが現状では困難である。
GMWI-webtoolは誰でも糞便ショットガンメタゲノムの分類プロファイルからGMWIおよびその他の生態学的指標を計算できる。
- 特定の被験者の腸内マイクロバイオームが大規模な症例または対照集団と比べてどのような位置にあるかを理解することは、有意義な知見をもたらす可能性がある。しかし、解析済み糞便メタゲノムデータセットのデータベースが不足しているため、現状では腸内マイクロバイオームに対してそれを行うのは容易ではない。GMWI-webtoolを用いれば、ユーザーは自らの結果を、統合リファレンス腸内マイクロバイオームデータセット(計5026件のメタゲノムサンプル)に基づく健康群(自己申告により疾患または疾患関連症状のない者)および非健康群(12種類の異なる疾患または異常体重状態のいずれかを有する患者)と比較できる。
コメント
少し深堀りしてみると、健康な腸内マイクロバイオームを定義すること自体が難しく、スコアが高い=健康とは言い切れないという意見があるようです。リンク先のGMWI2の記事では、個人差により腸内構成が大きく異なるため、ひとつのモデルがすべての人に当てはまるわけではないこと、また腸内マイクロバイオームは日々変動するため、スコアの時点評価よりも変化を追うことが重要と説明しています。取扱には注意が必要かと思います。
引用
GMWI-webtool: a user-friendly browser application for assessing health through metagenomic gut microbiome profiling
Daniel Chang, Vinod K Gupta, Benjamin Hur, Kevin Y Cunningham, Jaeyun Sung
Bioinformatics, Volume 39, Issue 2, February 2023
GMWI 2
Gut Microbiome Wellness Index 2 enhances health status prediction from gut microbiome taxonomic profiles
Daniel Chang, Vinod K Gupta, Benjamin Hur, Sergio Cobo-López, Kevin Y Cunningham, Nam Soo Han, Insuk Lee, Vanessa L Kronzer, Levi M Teigen, Lioudmila V Karnatovskaia, Erin E Longbrake, John M Davis 3rd, Heidi Nelson, Jaeyun Sung
Nat Commun. 2024 Aug 28;15(1):7447. doi: 10.1038/s41467-024-51651-9.
GMWI 1: previously called GMHI
A predictive index for health status using species-level gut microbiome profiling
Vinod K Gupta, Minsuk Kim, Utpal Bakshi, Kevin Y Cunningham, John M Davis III, Konstantinos N Lazaridis, Heidi Nelson, Nicholas Chia, Jaeyun Sung
Nat Commun. 2020 Sep 15;11:4635. doi: 10.1038/s41467-020-18476-8
関連