2021 8/22 説明を修正
MNIONのシーケンスデータはFAST5というフォーマットで出力される。FAST5はHDF5(wiki)という時系列データ関係でよく使われる形式に乗っ取っている。塩基配列になっていないバイナリーなデータである。このデータから塩基配列に変換するツールがいくつか発表されている。導入して検証してみる。
Poretools
公式ページ https://poretools.readthedocs.io/en/latest/index.html
brewでインストールできる。
brew install poretools
Bioconda環境なら"conda install poretools"で入る。
fastqの抽出。
poretools fastq <directory> > output.fq
--type bestの指定で2Dリードのみ出力する。他にtemplateかcomplementなどがある。デフォルトではallになっている。ディレクトリはnanopore/fast5/0/にあるなら、~0/まで記載しないとエラーになる。
fastaの抽出。
poretools fasta <directory> > output.fq
シーケンスのイールド。電圧の変更でポアが急激に減っていっていないかチェックするのに使える。
poretools yield_plot --plot-type reads <directory> --saveas output.pdf
--saveas: PDFかpngで保存。オプション無しだと画面出力される。
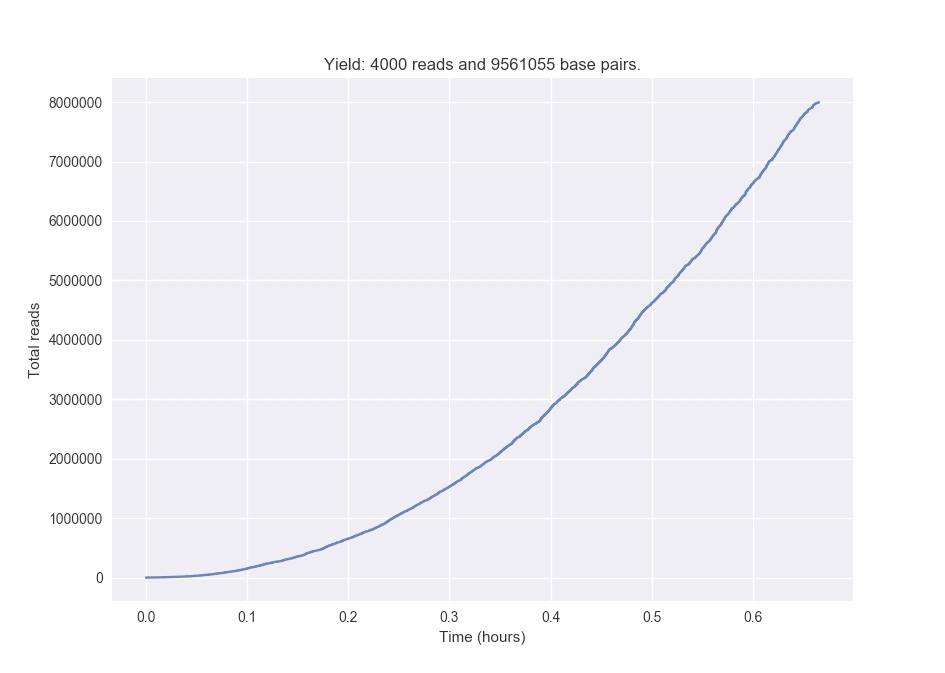
リード情報。
poretools stats <directory>
例えば以下のような画面が出力される。
total reads 4000
total base pairs 9561055
mean 2390.26
median 1182
min 11
max 148048
N25 10196
N50 4566
N75 1919
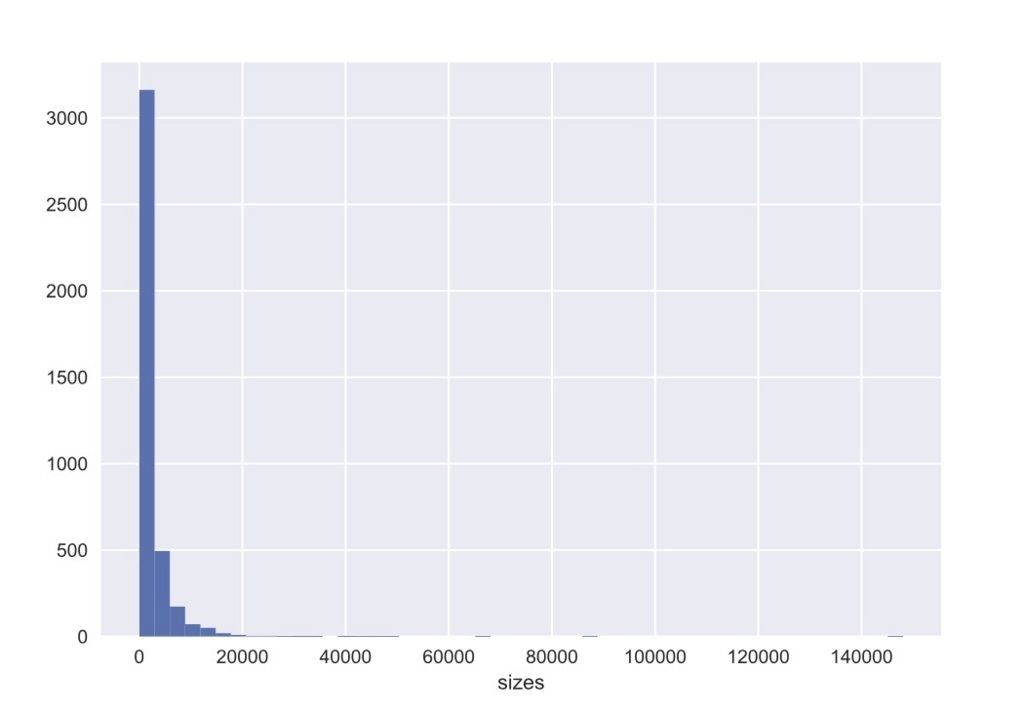
リード長のヒストグラム。
poretools hist <directory> --saveas output.pdf
--num-bins、 --max-length、 --min-lengthで間隔や下限、上限を指定することもできる。
塩基組成。
poretools nucdist <directory>
例えば以下のような画面が出力される。
A 2242285 9561055 0.234522759256
C 2455787 9561055 0.256853140161
T 2375819 9561055 0.248489209611
G 2487164 9561055 0.260134890972
クオリティスコア分布。
poretools qualdist <directory>
以下のような画面が出力される。
" 1 19774 9561055 0.0020681818063
# 2 47996 9561055 0.00501994811242
$ 3 118476 9561055 0.0123915195551
.
.
.
H 39 1074 9561055 0.000112330699907
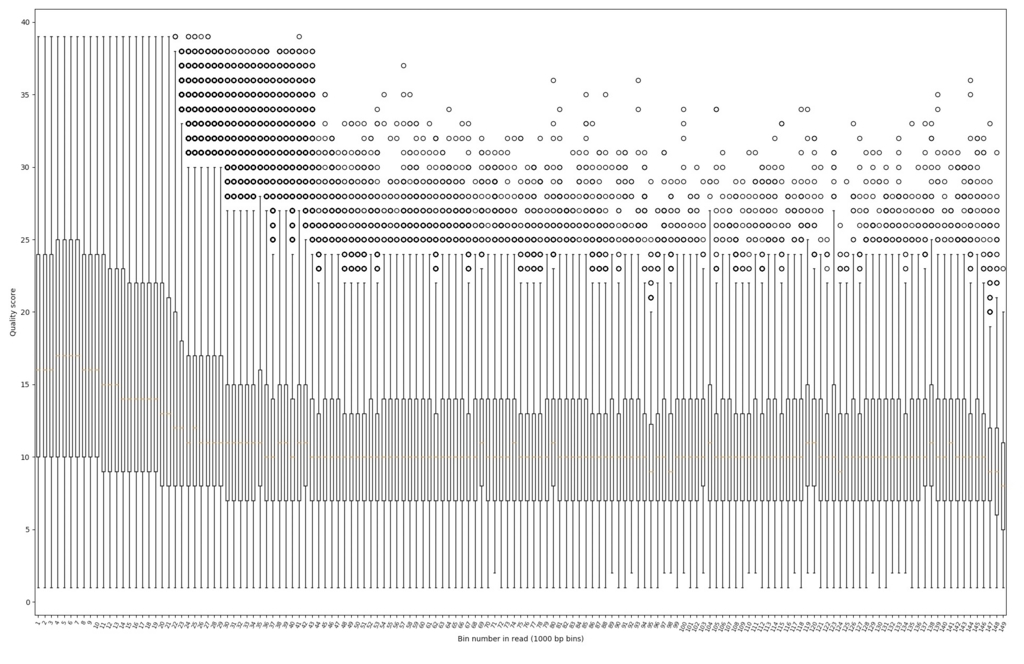
クオリティスコア分布のbox plot。
poretools qualpos <directory> --saveas output.pdf
フローセルの各ポアの総合パフォーマンス。
poretools occupancy <directory>

他にも、塩基変換前の生データやsquiggle plot を出力する機能などを持っている。公式サイトで確認してください。
Nanopolish
こちらでインストールは紹介している。
PoreSeq
Githubページ
GitHub - tszalay/poreseq: Error correction and variant calling algorithm for nanopore sequencing
githubからダウンロードして、PoreSeqのルートでsudo pip install -e .でインストールできる。
mac OSXで動作はするが、内部でLASTとsamtoolsを動かしているらしく、samtoolsのsort付近でエラーになりファイルが消える。現在のところ未修正。