次世代シーケンス(NGS)データは生命メカニズムへの前例のないアクセスを提供する。特に、これらのデータは染色体、個体または種間の遺伝的差異を評価することを可能にする。そのような多型は、農学、環境または医学における多数の用途を有する生物学の多くの局面における基本的な情報源を表す。
NGS技術によるシーケンシングの民主化に伴い、SNPまたはインデルなどの遺伝的差異を決定することは今や日常的な作業となっている。そのような多型を予測するために設計された多数のソフトウェアが存在する。ほとんどの場合、これらの方法はGATK [ref.6]やSamTools [ref.14]の場合のようにシーケンスリードをマッピングすることによって、あるいはDISCOVAR [ref.22](紹介)やFERMI [ref.12](紹介)のように部分アセンブリをマッピングすることによるリファレンスゲノムの使用に基づいている。基本的に、それらは最初にシーケンスをリファレンスゲノムにマッピングし、そして第二段階でそれらはリファレンス遺伝子座を走査して各遺伝子座についてリファレンス配列とマッピングされた配列との間の差異を分析する。
これらの方法は広く受け入れられ広く使用されている。しかしながら、それらは2つの重大な欠点を提示する。最初にそれらは非常に敏感である。リファレンスゲノムのrepetitive regionは、十分な信頼度でマッピングすることが困難である。これらのリピート領域から検出された多型は、マッピングされたリードの定量が誤っており、さらにリピートの出現間の差異が誤って多型として解釈される可能性があるため、誤っている可能性がある。第二に、それらは高品質のリファレンスゲノムを必要とするという事実に苦しんでいる。この明白で強い条件は、十分に研究された種への適用を制限する。(一部略)
うまくアセンブリされたリファレンス配列を必要とせずに、シーケンシングリードから直接SNPおよびindelを検出するリファレンスフリーの方法に対する重要かつ絶え間ない要求が依然としてある。 [ref.23]のように、代替的な方法は最初にリードをアセンブルしてリファレンスにその配列をマッピングする。しかしながら、そのような方法は、アセンブリおよびマッピングの両方の問題を累積する。ここでは、このような方法をハイブリッド戦略と呼ぶ。
いくつかの方法[ref.19、8、11、18、15、21]が多型の新規検出のために提案された。これらすべての方法は、de Bruijnグラフ、すなわち頂点の集合がリードに含まれる長さk(k-mers)のワードの集合に対応し、2つのkの間に辺がある有向グラフの使用に基づいている。それらがk - 1ヌクレオチド上で完全に重複するならばこのデータ構造では、多相は「バブル」と呼ばれる認識可能なパターンを生成する。これらのツールは、その起源を解読するためにそのようなバブルを検出し分析する(シーケンシングエラー、不正確なリピートによる多型、実際のSNPまたはindel)。
ここでは、DiscoSnp ++を提示する。これは、スキル、計算上のニーズ、および結果のクオリティの点で、他のリファレンスフリーの方法よりも優れた手法である。その主な特徴は、1、予測される変異型の範囲: 分離されているかどうかにかかわらずSNP、small indel。2、数十億のリードを6 GB以下のRAMメモリで処理できる非常に少ないメモリ使用量、3、その高い実行速度、4、優れたprecisionと recall、5、予測された各バリアントに割り当てられたfaithful score、6、出力ファイルはfastaおよびVCFフォーマットで、簡単にダウンストリーム分析に使用可能、7、1つだけも含み、任意の数のリードセットに適用できる、である。
DiscoSnp ++はDiscoSnpの全く新しいバージョンである[ref.21]。このツールはGATBライブラリ[ref.7](Github)を使用して最初から再実装されたため、実行時間が大幅に短縮され、メモリ使用量が削減された。シーケンシングエラーをよりよく排除し、新しい種類のバリアントを検出するために、アルゴリズムモデルが再検討された。DiscoSnp ++は、必要に応じてその予測をリファレンスゲノムにマッピングした後に、一般的に使用されているVCFフォーマットで予測結果を出力する。この最後の点は、リファレンスのないアプローチに対して直感に反するように見えるかもしれないが、de novo予測したバリアントの位置を決めるためにはリファレンスゲノムが特に役立つ。これは、リファレンスがあまりにもひどくアセンブリされているか、またはシーケンシングされた種から遠すぎる場合には特に当てはまる。さらに、優れたリファレンスゲノムの場合でさえ、リファレンスフリーアプローチを用いたバリアント予測および遺伝子型決定は、リファレンス対立遺伝子によって決してバイアスされない。
Our paper "Discovering Millions of Plankton Genomic Markers from the Atlantic Ocean and the Mediterranean Sea" is just published. https://t.co/CDSodQTMTX
— Pierre Peterlongo (@p_peterlongo) December 27, 2018
See how DiscoSnp can be used for discovering variants from seawater metagenomic raw data.@Genoscope pic.twitter.com/98dLViBXHw
DiscoSnpに関するツイート
インストール
ubuntu18.04でテストした。
依存
- CMake 2.6+; see http://www.cmake.org/cmake/resources/software.html
- c++ compiler; compilation was tested with gcc and g++ version>=4.5 (Linux) and clang version>=4.1 (Mac OSX).
本体 Github
git clone --recursive https://github.com/GATB/DiscoSnp.git
cd DiscoSnp
sh INSTALL
#bioconda(mac osにも対応)
conda install -c bioconda -y discosnp
> ./run_discoSnp++.sh
$ ./run_discoSnp++.sh
You must provide at least one read set (-r|--fof)
************
*** HELP ***
************
run_discoSnp++.sh, a pipelining kissnp2 and kissreads for calling SNPs and small indels from NGS reads without the need of a reference genome
Version 2.3.X
Usage: ./run_discoSnp++.sh -r read_file_of_files [OPTIONS]
MANDATORY
-r|--fof <file name of a file of file(s)>
The input read files indicated in a file of file(s)
Example: -r bank.fof with bank.fof containing the two lines
data_sample/reads_sequence1.fasta
data_sample/reads_sequence2.fasta.gz
OPTIONS
-k | --k_size value <int value>
Set the length of used kmers. Must fit the compiled value.
Default=31
-c | --min_coverage value <int value>
Set the minimal coverage per read set: Used by kissnp2 (don't use kmers with lower coverage) and kissreads (read coherency threshold).
This coverage can be automatically detected per read set (in this case use "auto" or specified per read set, see the documentation.
Default=3
-C | --max_coverage value <int value in 0, 1 or 2>
Set the maximal coverage for each read set: Used by kissnp2 (don't use kmers with higher coverage).
Default=2^31-1
-b | --branching value.
0: forbid variants for which any of the two paths is branching (high precision, lowers the recall in complex genomes).
Default value
1: (smart branching) forbid SNPs for which the two paths are branching (e.g. the two paths can be created either with a 'A' or a 'C' at the same position
2: No limitation on branching (lowers the precision, high recall)
-s | --symmetrical value <int value>
In -b 2 mode only: maximal number of symmetrical crossroads traversed while trying to close a bubble. Default: no limit
-g | --graph <file name>
Reuse a previously created graph (.h5 file) with same prefix and same k and c parameters.
-X Stop discoSnp++ right after variant calling - the output is only a fasta file with no coverage information.
-D | --deletion_max_size <int>
discoSnp++ will search for deletions of size from 1 to D included. Default=100
-a | --ambiguity_max_size <int>
Maximal size of ambiguity of INDELs. INDELS whose ambiguity is higher than this value are not output [default '20']
-P | --max_snp_per_bubble <int>
discoSnp++ will search up to P SNPs in a unique bubble. Default=3
--fof_mapping <file name of a file of file(s)>
If this option is used this fof is used when mapping back reads on the predicted variants instead of the original fof file provided by -r|--fof option
-p | --prefix <string>
All out files will start with this prefix. Default="discoRes"
-l | --no_low_complexity
Remove low complexity bubbles
-T | --contigs
Extend found polymorphisms with contigs (default: extend with unitigs)
-d | --max_substitutions <int>
Set the number of authorized substitutions used while mapping reads on found SNPs (kissreads). Default=1
-n | --no_genotype
Do not compute the genotypes
-u | --max_threads <int>
Max number of used threads. 0 means all threads
default 0
REFERENCE GENOME AND/OR VCF CREATION OPTIONS
-G | --reference_genome <file name>
Reference genome file (fasta, fastq, gzipped or nor). In absence of this file the VCF created by VCF_creator won't contain mapping related results.
-R
Use the reference file also in the variant calling, not only for mapping results
-B | --bwa_path <directory name>
bwa path. e.g. /home/me/my_programs/bwa-0.7.12/ (note that bwa must be pre-compiled)
Optional unless option -G used and bwa is not in the binary path.
-e Map variant predictions on reference genome with their unitig or contig extensions.
Useless unless mapping on reference genome is required (option -G).
-w Wraith mode: only show all discoSnp++ commands without running them
-v <0 or 1>
Verbose 0 (avoids progress output) or 1 (enables progress output) -- default=1.
-h | --help
Prints this message and exist
Any further question: read the readme file or contact us via the Biostar forum: https://www.biostars.org/t/discosnp/
> run_discoSnpRad.sh -help
$ run_discoSnpRad.sh -help
run_discoSnpRad.sh, pipelining kissnp2 and kissreads and clustering per locus for calling SNPs and small indels from RAD-seq data without the need of a reference genome
Version 2.3.X
Usage: ./run_discoSnpRad.sh -r read_file_of_files [OPTIONS]
MANDATORY:
-r read_file_of_files
Example: -r bank.fof with bank.fof containing the two lines
data_sample/reads_sequence1.fasta
data_sample/reads_sequence2.fasta.gz
-S **absolute** path to short_read_connector directory, containing the short_read_connector.sh'' file.
-Note1: short read connector must be compiled.
-Note2: if this option is missing, discoSnpRad will still however provide a fasta file containing SNPs and INDELS, that won't be clustered by locus
DISCOSNPRAD OPTIONS:
-g: reuse a previously created graph (.h5 file) with same prefix and same k and c parameters.
-b value.
1: (smart branching) forbid SNPs for which the two paths are branching (e.g. the two paths can be created either with a 'A' or a 'C' at the same position Default value
2: No limitation on branching (lowers the precision, high recall)
-s value. In b2 mode only: maximal number of symmetrical croasroads traversed while trying to close a bubble. Default: no limit
-D value. discoSnpRad will search for deletions of size from 1 to D included. Default=100
-a value. Maximal size of ambiguity of INDELs. INDELS whose ambiguity is higher than this value are not output [default '20']
-P value. discoSnpRad will search up to P SNPs in a unique bubble. Default=5
-p prefix. All out files will start with this prefix. Default="discoRad"
-l: remove low complexity bubbles
-k value. Set the length of used kmers. Must fit the compiled value. Default=31
-c value. Set the minimal coverage per read set: Used by kissnp2 (don't use kmers with lower coverage) and kissreads (read coherency threshold). This coverage can be automatically detected per read set or specified per read set, see the documentation. Default=auto
-C value. Set the maximal coverage for each read set: Used by kissnp2 (don't use kmers with higher coverage). Default=2^31-1
-d value. Set the number of authorized substitutions used while mapping reads on found SNPs (kissreads). Default=1
-u: max number of used threads
-v: verbose 0 (avoids progress output) or 1 (enables progress output) -- default=1.
-h: Prints this message and exist
Any further question: read the readme file or contact us via the Biostar forum: https://www.biostars.org/t/discosnp/
テストラン
例えば1セットのリードを調べ、不完全SNVとsmall indelを検出する。fastq/fastaのファイル名をテキストで指定する。
git clone https://github.com/GATB/DiscoSnp.git
cd DiscoSnp/test/
run_discoSnp++.sh -r fof.txt -T
- -r read_file_of_files. Example: -r bank.fof with bank.fof containing the two lines
data_sample/reads_sequence1.fasta
data_sample/reads_sequence2.fasta.gz - -T extend found polymorphisms with contigs
リードに加えリファレンスを指定する。さらにこのリファレンスを使ってバリアントコールを行う。bwaが必要。
run_discoSnp++.sh -r test/fof.txt -T -G test/reference_genome.fa -R
- -G reference genome file (fasta, fastq, gzipped or nor). In absence of this file the VCF created by VCF_creator won't contain mapping related results.
- -R use the reference file also in the variant calling, not only for mapping results
リファレンスを与えた場合でも、与えなかった場合でも、このデータセットでコールされるのは不完全なSNV三箇所になる。
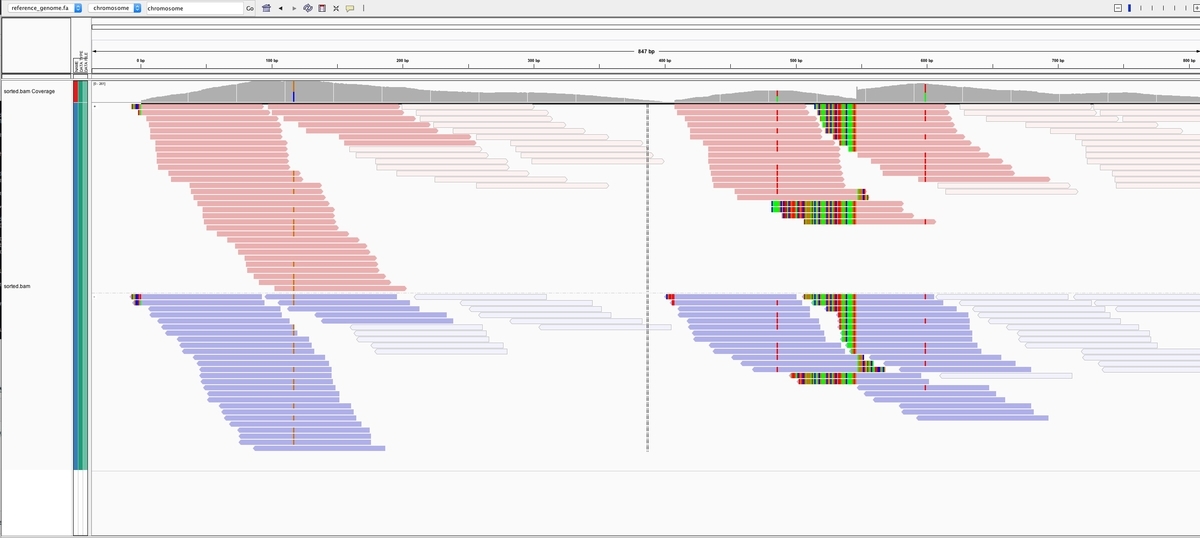
リファレンスを与えると、リファレンスにmappingしてリファレンスでのポジションを報告してくれる。
引用
DiscoSnp ++ : de novo detection of small variants from raw unassembled read set(s)
Peterlongo, P., Riou, C., Drezen, E., Lemaitre, C.
bioRxiv preprint first posted online Oct. 27, 2017
Reference-free detection of isolated SNPs
Uricaru R., Rizk G., Lacroix V., Quillery E., Plantard O., Chikhi R., Lemaitre C., Peterlongo P. (2014).
Nucleic Acids Research 43(2):e11