2018 9/19 コマンド修正
2018 11/17 文章修正
2018 12/15 インストール追記
2019 2/28 追記
2019 3/19 scafofldsのコマンドのミス修正
2019 5/15リンク追加
2020 3/8動画追加
2022/06/02 ツイート追加
bandageはde novo assemblerのfastgファイルを入力として、graphパスを描画してくれるツール。2015年に論文が発表された。
インストール
公式サイト
公式サイトからdmgファイルをダウンロード。指示にしたがってインストールする。
https://github.com/rrwick/Bandage/wiki
linuxなら、condaで導入して、コマンドラインから作図したりできる。
conda install -c bioconda -y bandage
>Bandage -h
$ Bandage -h
____ _
| _ \ | |
| |_) | __ _ _ __ __| | __ _ __ _ ___
| _ < / _` | '_ \ / _` |/ _` |/ _` |/ _ \
| |_) | (_| | | | | (_| | (_| | (_| | __/
|____/ \__,_|_| |_|\__,_|\__,_|\__, |\___|
__/ |
|___/
Version: 0.8.1
Usage: Bandage <command> [options]
Commands: <blank> Launch the Bandage GUI
load Launch the Bandage GUI and load a graph file
info Display information about a graph
image Generate an image file of a graph
querypaths Output graph paths for BLAST queries
reduce Save a subgraph of a larger graph
Options: --help View this help message
--helpall View all command line settings
--version View Bandage version number
> ./spades_contig_graph.py -h
$ ./spades_contig_graph.py -h
usage: spades_contig_graph.py [-h] [-p PATHS_OUT] (-c | -l)
graph contigs paths output
SPAdes Contig Graph: a tool for creating FASTG contig graphs from SPAdes
assemblies
positional arguments:
graph The assembly_graph.fastg file made by SPAdes
contigs A contigs or scaffolds fasta file made by SPAdes
paths The paths file which corresponds to the contigs or
scaffolds file
output The graph file made by this program
optional arguments:
-h, --help show this help message and exit
-p PATHS_OUT, --paths_out PATHS_OUT
Save the paths to this file (default: do not save
paths)
-c, --connection_priority
Prioritise graph connections over segment length
-l, --length_priority
Prioritise segment length over graph connections
——
プログラムをダウンロードしたら、passの通ったディレクトリにコピーしておく。
sudo cp Bandage /usr/local/bin/
#起動
Bandage
#起動したらblastが使えるか確認する
>B
spadesのグラフデータを使う場合、spadesのスクリプトspades_contig_graph.pyを使いフォーマットを少し変える必要がある。brewでspadesをインストールしているなら以下のように打つ。
#length
./spades_contig_graph.py -l assembly_graph.fastg contigs.fasta contigs.paths output_length.fastq
#coverage
./spades_contig_graph.py -c assembly_graph.fastg contigs.fasta contigs.paths output_coverage.fastq
#scaffoldsに対して行うなら
#coverage
./spades_contig_graph.py -c assembly_graph.fastg scaffolds.fasta scaffolds.paths output_coverage.fastq
contigs.fasta、contigs.path、assembly_graph.fastgは、spadesのアセンブル結果出力ディレクトリに存在する。それを指定している。"-p prefix" でpathファイルも別途保存できる。
またはscaffoldsに対して実行する。出力されるfastgをbandageに入力する。
実行方法
inputよりfastgファイルを選択。

Draw Graphをクリックするとアセンブルのgraphが描画される。

-基本操作-
- 拡大縮小:Trackpadでピンチイン/アウトかマウスホイールを回す。
- 画面のスクロール:Trackpadで2本指のドラッグかCtrl+画面のドラッグ。
- ノードの移動:ドラッグでノードを引っ張る。
- 複数ノードの移動:ノードをマウスで囲い選択してから引っ張る。
- edgeの形の変更:controlキーを押しながらedgeの変更したい部分をドラッグ。
- 画面の回転:Trackpadで2本指の回転か、Ctrl+画面の右クリックドラッグ。
- ノードの形を局所的に変更: Control +クリック
指定した情報をnodeの上に表示できる。

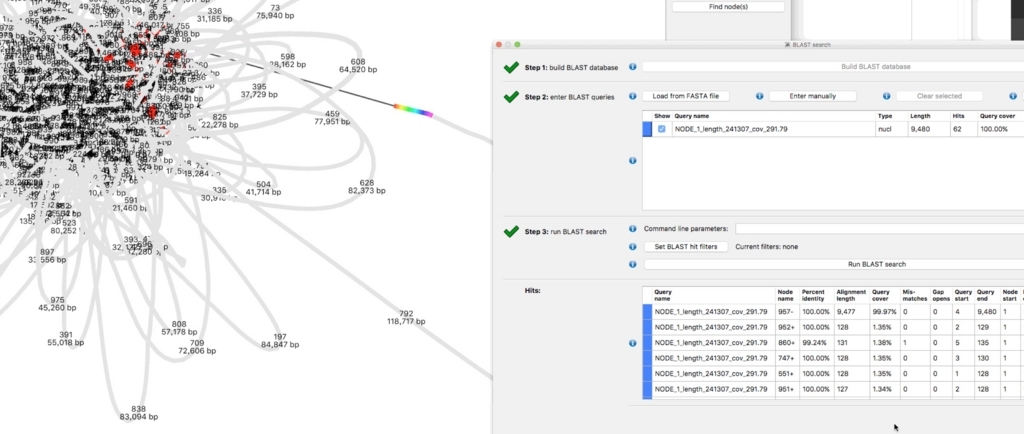
マーカー遺伝子がアセンブルされたcontigのどこに存在するか確認したいことがある。そういう時はbandage内部でBLAST解析すれば良い。Create/view BLAST searchを選択。

Build BLAST databaseをクリックし、contig配列からBLASTデータベースを作成する。

左上の方のLoad from FASTA fileを選択し、探したい配列のfastaファイルを選択する。終わったら中央下のRun BLAST searchを選択する。
解析が終わるとヒットしたノード名と領域が表示され、されにノードのヒットした領域がカラー表示される。
アノテーションをかけてコード領域のfnaファイルがあれば、遺伝子を可視化することも可能。
ドラッグしてnodeを囲めば、 選択されたnodeの合計サイズと平均depthを出せる。

contigを探すなら、右上の小窓にcontig名を打つ。

Bandageを使えば、アセンブルが途切れている可能性がある耐性カセットの位置を見極めるのも簡単である。例えば以下の記事では、bandageを使ったアセンブルのグラフからカセット配列を見つける流れが書かれている。
追記
この方の使い方がとても参考になります。動画を貼っておきます。
追記
引用
Bandage: interactive visualization of de novo genome assemblies
Wick RR, Schultz MB, Zobel J, Holt KE
Bioinformatics. 2015 Oct 15;31(20):3350-2
追記
2021 3/16
他のノードと最もエッジを共有しているノードを探す
Has anyone looked into what the most shared nodes in assembly graphs are? 1.4 Gbp assembly graph monster. #metagenomics pic.twitter.com/J4ei8WCU7D
— Rasmus Kirkegaard (@kirk3gaard) 2021年3月15日
Bandage: error while loading shared libraries: libGL.so.1: cannot open shared object file: No such file or directoryが出た時。
apt-get install libglu1
で解決(ubuntu)。
2022/06/02
Bandageのfork
Bandage-NG
Dear Bandage users and friends! As you might know, original Bandage by @rrwick entered maintenance stage. Together with our student @wafemand from @spbifmo_en we are presenting a pre-release of improved Bandage 0.9.5 (https://t.co/0BhTp0vX73) which features:
— Center for Algorithmic Biotechnology (@bioinf_spbu) June 2, 2022
1/3
