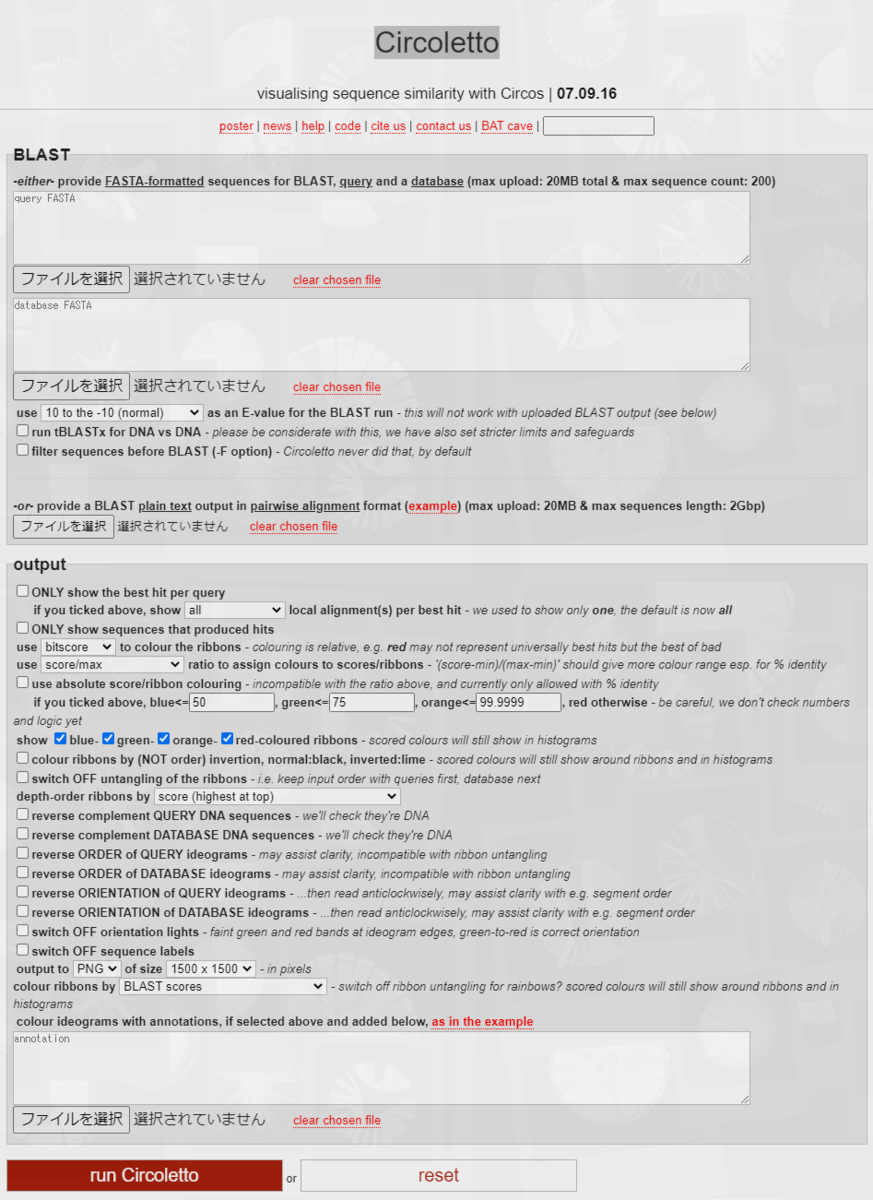
circosは多様な機能を備えたゲノムのビジュアル化ツールである。類似ソフトにDNA plotter、genome diagram、snap geneなどがあるが、機能の豊富さではcircosが抜きん出てる。circos独自の機能として、遺伝子の相関や相同性を線で表現してlinkageを表す機能がある。この表現を使い、染色体間の相同性や転移、発現調節などを表現した様々な図が作成されている。例えばキーワード"circos"で画像検索するとわかる (search)。
circosは環状表現でなんでも描けるが、 難点として、機能が豊富な分使いこなすにはかなり勉強が必要であることが挙げられる。
インストール(mac OS Sierra 10.12.2で検証した)
brew install circos
ランするには、circos.confファイルを作成してターミナルで下のように打つ。
> circos -conf circos.conf
この.confファイル中に図を書くのに必要なパラメータを記載する。circosはその情報に従ってランしているわけである。
confファイルには非常に多様な情報を記載することができるが、プログラムに慣れていないといきなり理解するのは難しい。circos公式サイトのチュートリアルを真似て、1つずつ理解していくのが一番である。一日頑張れば、欲しい図は大体かけると思う。
下にチュートリアルのリンクを載せておく。http://circos.ca/documentation/tutorials/
トレーニング1
circos.confの中身は、トレーニングページに公開されている。例えば練習1の
http://circos.ca/documentation/tutorials/quick_start/ticks_and_labels/configuration のページの # 1.1 MINIMUM CIRCOS CONFIGURATION ... ... <<include etc/housekeeping.conf>>
までを全てプレーンテキストエディタ(mi とか)に 貼り付ける。circos.confとして保存して、
circos -conf circos.conf
と実行する。 正常に終わるとpngとsvgファイルができる。DNA plotterならマウスオーバーで 情報を表示したり編集したりできるが、他ソフトと異なりcircosの結果は画像で出力される。後から編集することはできない。練習ページ1の出力結果が下になる。

上の図のパラメータは以下の通り
default = 0.005r #box間のスペース
radius = 0.90r #サークルのサイズ
thickness = 20p # boxの厚み
fill = yes #box内部のカラー
stroke_color = dgrey
stroke_thickness = 2p #box輪郭線の厚み
上のパラメータを
default = 0.05r
fill = no
stroke_thickness = 10p
に変更すると、下のようになる。

circos.confの中の
# Chromosome name, size and color definition
の次の行に
karyotype = /Users/user/karyotype.human.txt
という行がある。このkaryotype.human.txtはあくまで
例だが、中身は以下のような構造をしている。
chr - hs1 1 0 249250621 chr1
chr - hs2 2 0 243199373 chr2
chr - hs3 3 0 198022430 chr3
.
.中略
.
band hs1 p36.33 p36.33 0 2300000 gneg
band hs1 p36.32 p36.32 2300000 5400000 gpos25
band hs1 p36.31 p36.31 5400000 7200000 gneg
band hs1 p36.23 p36.23 7200000 9200000 gpos25
.
.中略
.
band hsY q11.223 q11.223 22100000 26200000 gpos50
band hsY q11.23 q11.23 26200000 28800000 gneg
band hsY q12 q12 28800000 59373566 gvar
886行目まである(karyotype.human.txtはcircosのexampleファイルの中に入っている)。
circos.confのkaryotype = /Users/user/karyotype.human.txt
の行は、karyotype.human.txtのパスを指定している。karyotype.human.txtを保存したパスに修正しておく。パスの修正はその都度行う必要がある。はじめのうちはミスしやすいところなので注意する。
karyotype.human.txtはhttps://github.com/vigsterkr/circos/tree/master/data
からダウンロードできる。文字をコピペしてkaryotype.human.txtとして保存。
トレーニング2
各クロモソームにアノテーション情報や目盛りをつける。
http://circos.ca/documentation/tutorials/quick_start/ticks_and_labels/configuration
の3つのコードを
CIRCOS.CONF
IDEOGRAM.CONF
TICKS.CONF
それぞれ上の名称で保存する。実行はcircos -conf circos.conf
下の画像が出力されるはず。

CIRCOS.CONF の中の
<<include ideogram.conf>>
<<include ticks.conf>>
の2行でIDEOGRAM.CONFとTICKS.CONFを読み込んでいる。
boxの情報はideogram.confファイルの中の
radius = 0.90r
thickness = 20p
fill = yes
stroke_color = dgrey
stroke_thickness = 2p
に書かれている。
アノテーション情報は
show_label = yes
# see etc/fonts.conf for list of font names
label_font = default
label_radius = dims(image,radius) - 60p
label_size = 30
label_parallel = yes
に書かれている。
目盛りはticks.confファイルの中の
show_ticks = yes
show_tick_labels = yes
<ticks>
radius = 1r
color = black
thickness = 2p
でメモリの厚みなどを規定。さらに、下の方の
<tick> #1種類目
spacing = 5u
size = 10p
</tick>
<tick>#2種類目
spacing = 25u
size = 15p
show_label = yes
label_size = 20p
label_offset = 10p
format = %d
</tick>
の2つの<tick> </tick>の中で、2種類の目盛りを指定している。
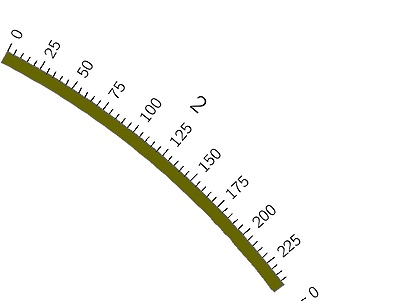
上の図のように、5ポイントごとの目盛りと、25ポイントごとの目盛りが描画される。1つ目の目盛りは文字なしなので、
show_label = yes
label_size = 20p
label_offset = 10p
format = %d
の行は書かれていない。
肝心のクロモソーム情報はkaryotype.human.txtから読み込んでいる。karyotype.human.txtファイル冒頭の
chr - hs1 1 0 249250621 chr1
chr - hs2 2 0 243199373 chr2
chr - hs3 3 0 198022430 chr3
.
.中略
.
chr - hs22 22 0 51304566 chr22
chr - hsX x 0 155270560 chrx
chr - hsY y 0 59373566 chry
を元にboxの名前とサイズが決められている。
トレーニング3
各クロモソームのサイズや向き、場所を変える。
http://circos.ca/documentation/tutorials/quick_start/selection_and_scale/configuration
から
の3つのコード
CIRCOS.CONF
IDEOGRAM.CONF
TICKS.CONF
を保存する。実行はcircos -conf circos.conf
下の画像が出力されるはず。

CIRCOS.CONFファイルの中の
chromosomes_scale = hs1=0.5r,/hs[234]/=0.5rn
でクロモソームのサイズを規定している。
chromosomes_reverse = /hs[234]/
でクロモソーム2、3、4の向きを逆転させている。
chromosomes_color = hs1=red,hs2=orange,hs3=green,hs4=blue
でクロモソームの色を指定している。
chromosomes_radius = hs4:0.9r
でクロモソーム4の位置を他より内側に指定している。
トレーニング4
各クロモソーム間のリンクを表示する練習。
http://circos.ca/documentation/tutorials/quick_start/links_and_rules/configuration
から
の3つのコード
CIRCOS.CONF
IDEOGRAM.CONF
TICKS.CONF
を保存する。
https://github.com/vigsterkr/circos/blob/master/data/5/segdup.txt
の文字をsegdup.txtとして保存する。
CIRCOS.CONFの58行目を
file = segdup.txt
にパスを変更する。
実行はcircos -conf circos.conf
下の画像が出力されるはず。

トレーニング2に続く。
web上でcircosのほぼ全機能を利用できるツールもあります。数時間で目的の絵を出せるよいツールです。
2021 7/23
http://tools.bat.infspire.org/circoletto/