2022/06/25追記
eggNOG 5.0のペーパーより
分化イベント後に共通の祖先から分岐したオルソログを同定することは、分子生物学および進化生物学における基本的な課題である。 duplication eventの後に分岐した配列であるパラログと比較して、オルソログは長い進化的タイムスケールでもその先祖の機能を保持する傾向がある(ref.3)。したがって、相同性関係のこれら2つのサブタイプを区別することは、正確な機能予測を作成するためにきわめて重要になる(ref.2,4,5)。それはまた、例えば系統学および比較ゲノミクス(ref.6)またはセルタイプ進化の研究(ref.7)における適切な分析にも不可欠である。そのため、さまざまなアプローチと運用上の定義を使用して事前計算されたオルソログ予測を提供するデータベースが長年にわたって開発されてきた(ref.8–13)。 eggNOGを含むこれらのリソースの大部分は、international consortium Quest for Orthologs(ref.14)の一部であり、標準化されたベンチマークアプローチ(ref.15)であり、リファレンスデータセットは開発され共有されている。
eggNOG(evolutionary genealogy of genes: Non-supervised Orthologous Groups)は、数千のゲノムが一度に分析されてそれらの全遺伝子間のオルソログ関係を確立している公共のリソースである。同種のデータベースと比較して、eggNOGは以下を提供することに焦点を合わせている。(i)推論されたオルソログに対する包括的な機能的アノテーション、(ii) three domains of lifeをカバーする何千ものゲノムにわたる予測、およびiii)オルソログ割り当てとphylogenetic analysisに基づく細かい粒度の階層的関係(すなわち、in-paralogies))。そのために、最善の相互ヒットの三角測量の概念に基づく種を意識したクラスタリングアルゴリズム(ref.16)を適用して、直交グループ(OG):同一のスペシエーションイベントから分岐し始めた相同配列のセットを識別する。 2008年に開始されて以来(ref.17)、生命のツリーの先端の解像度が高まるにつれオルソログ関係は想定されるリファレンスの分岐イベント(アウトグループ)に応じて変わってきているので、eggNOGは異なる分類レベルでオルソログ予測を計算する。すべての分類レベルからのすべてのOGs(orthologous groups)は、系統的方法を用いて機能的にアノテーション付けられ、分析される。これにより、各OG内のスペシエーションおよび重複イベントの履歴を詳細に調べることができる。
ここでは、eggNOG v5.0について説明する。以前のバージョンと比較して以下の点が改善されている。(i)原核生物、真核生物、ウイルスゲノムの中で最も包括的な選択肢の1つを含む、基礎データベースのメジャーアップグレード。 (ii)カスタム(メタ)ゲノムアノテーションのオンラインサービス更新。現在は高速オルソログ予測とクラウドコンピューティングによる計算能力の向上のオプション、および(iii)OGとそれに関連する機能データのより良い視覚化オプションが含まれる。
Methods
README (v4.5)
http://eggnogdb.embl.de/download/eggnog_4.5/README.txt
https://en.wikipedia.org/wiki/EggNOG_(database)
nice use of @eggnogdb and eggnog-mapper for functional annotation and orthology prediction in single-cell transcriptomics of non-model species. https://t.co/FYwcCTgcX3
— EggNOG database (@eggnogdb) May 8, 2018
EggNOG provides visualization of domain structure & alignment gappynes for all groups. Plus evolutionary context pic.twitter.com/CxpGTz7J5H
— EggNOG database (@eggnogdb) January 22, 2016
使い方
http://eggnog5.embl.de/#/app/homeにアクセスする。
2019年10月現在利用できるのは、5090のcellular organismsと2502のvirusesからの4.4Mのオロソロググループ情報となる。中央の下の方に以前のバージョンへのリンクもある。
上のサーチウィンドウからCitrate synthaseと検索してみた。複数候補表示された。
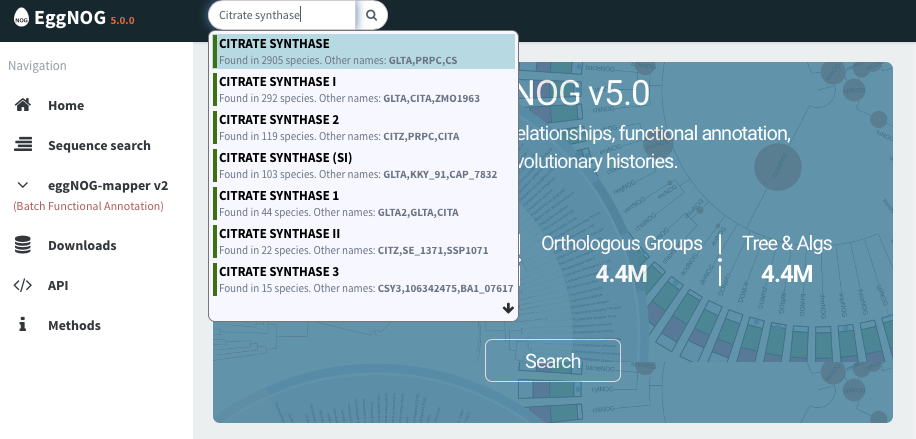
ここではCitrate synthase 1を選択。
そのキーワードでヒットしたオロソロググループが複数表示された。
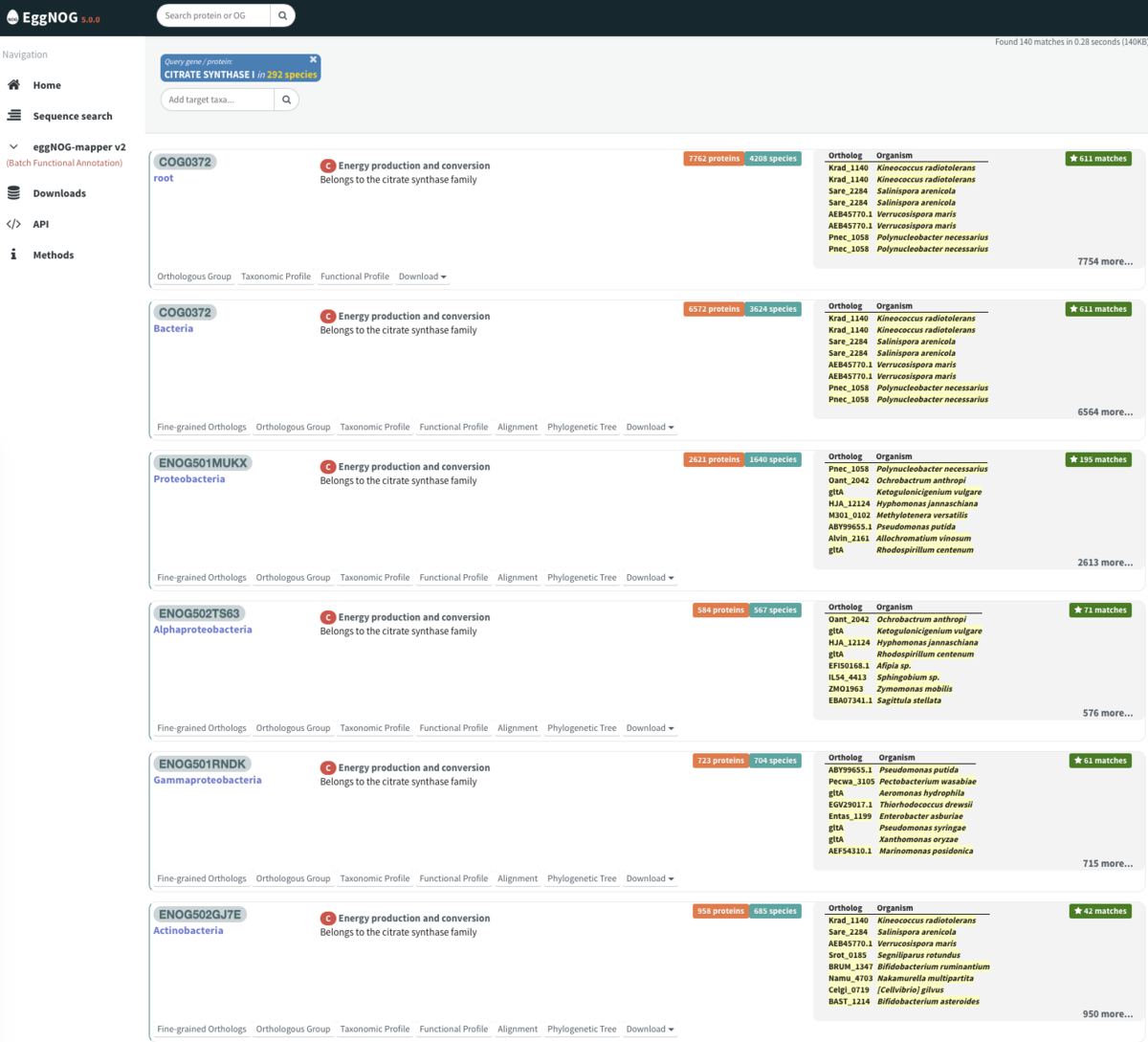
青がtaxonomy情報などを表している(rootは系統樹のrootのこと)。オレンジ部分が含まれるprotein数を表している。
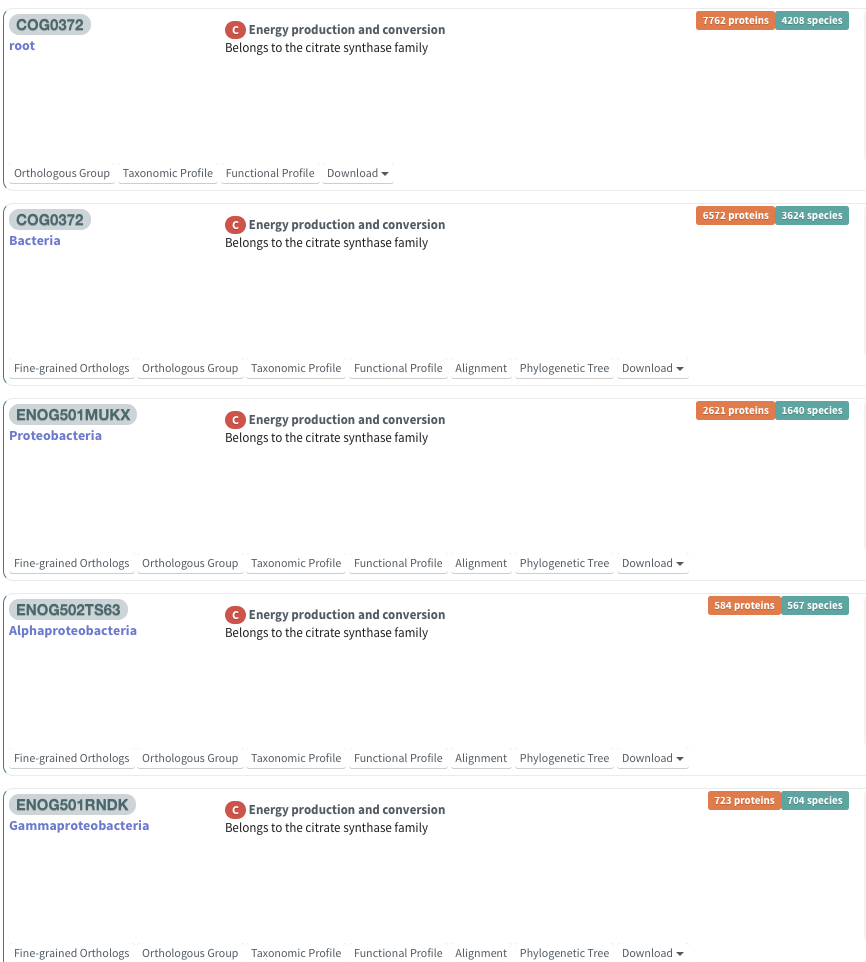
下の方はバクテリアの特定のクレードのヒットだった。
ここでは全生物を含めたいのでrootのCOG0372を選択した。改めてrootとbacteriaが表示された。ここではrootのオロソロググループを調べてみる。
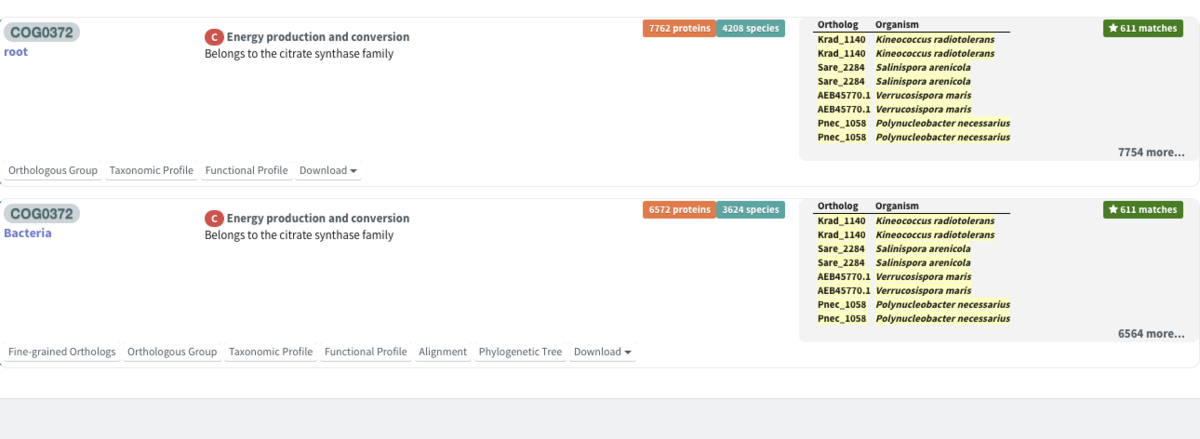
それぞれのヒットの下にある文字をクリックする。

Orthologous Groupをクリック。含まれるタンパク質リストが表示される。
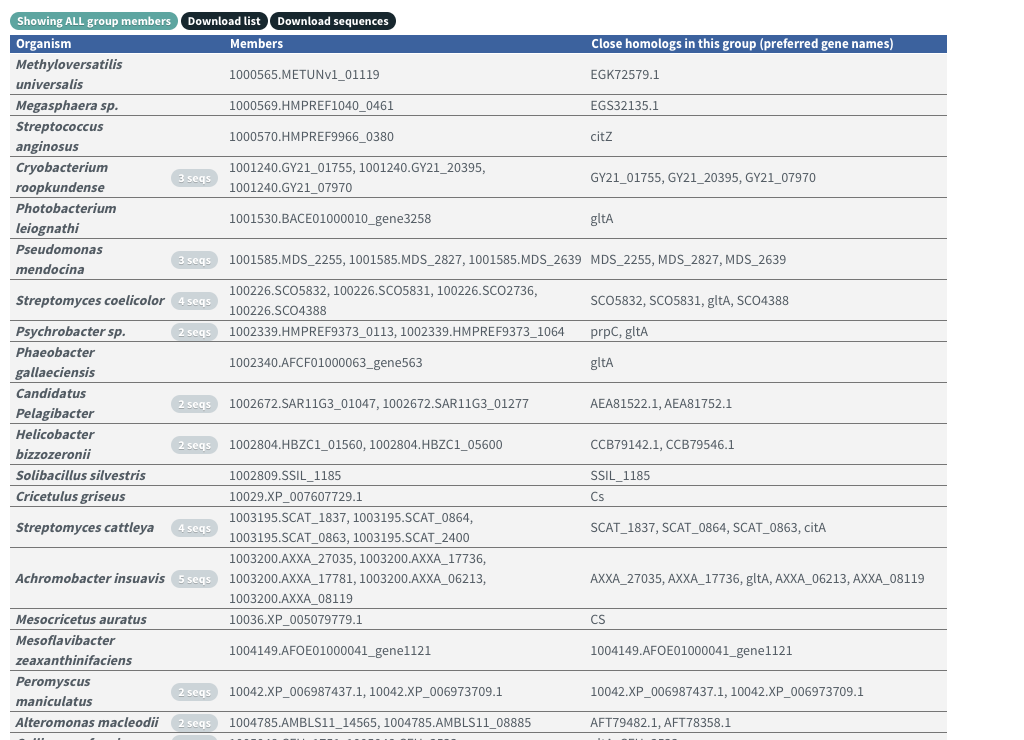
上のボタンから全リストと全配列をダウンロードできる。
左から2つ目のTaxonomic Profileをクリック。
Eukaryota(下の方の水色部分)は11.2%含まれる。
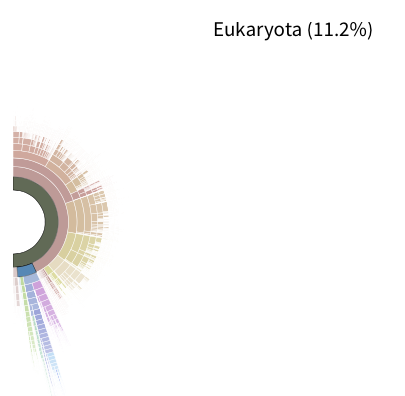
左から3つ目のFunctional Profileをクリック。3種類のアノテーション情報が利用できる。


Domains(アサイン頻度の高いもの)
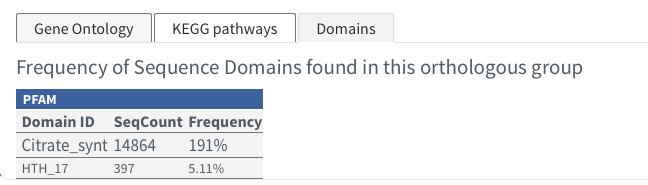
右端のDownloadから全データをダウンロードできる。
rootより下の階層ではより多くの比較を実行できる。
RubiscoのRibulose 1,5-bisphosphate carboxylase/oxygenase large subunit検索。下に表示されるメニューがrootより多いのが分かる。

ここでは143ヒットするシアノバクテリアを選択した。
alignment (raw /trimmed)
Phylogenetic Tree + PFAM domains
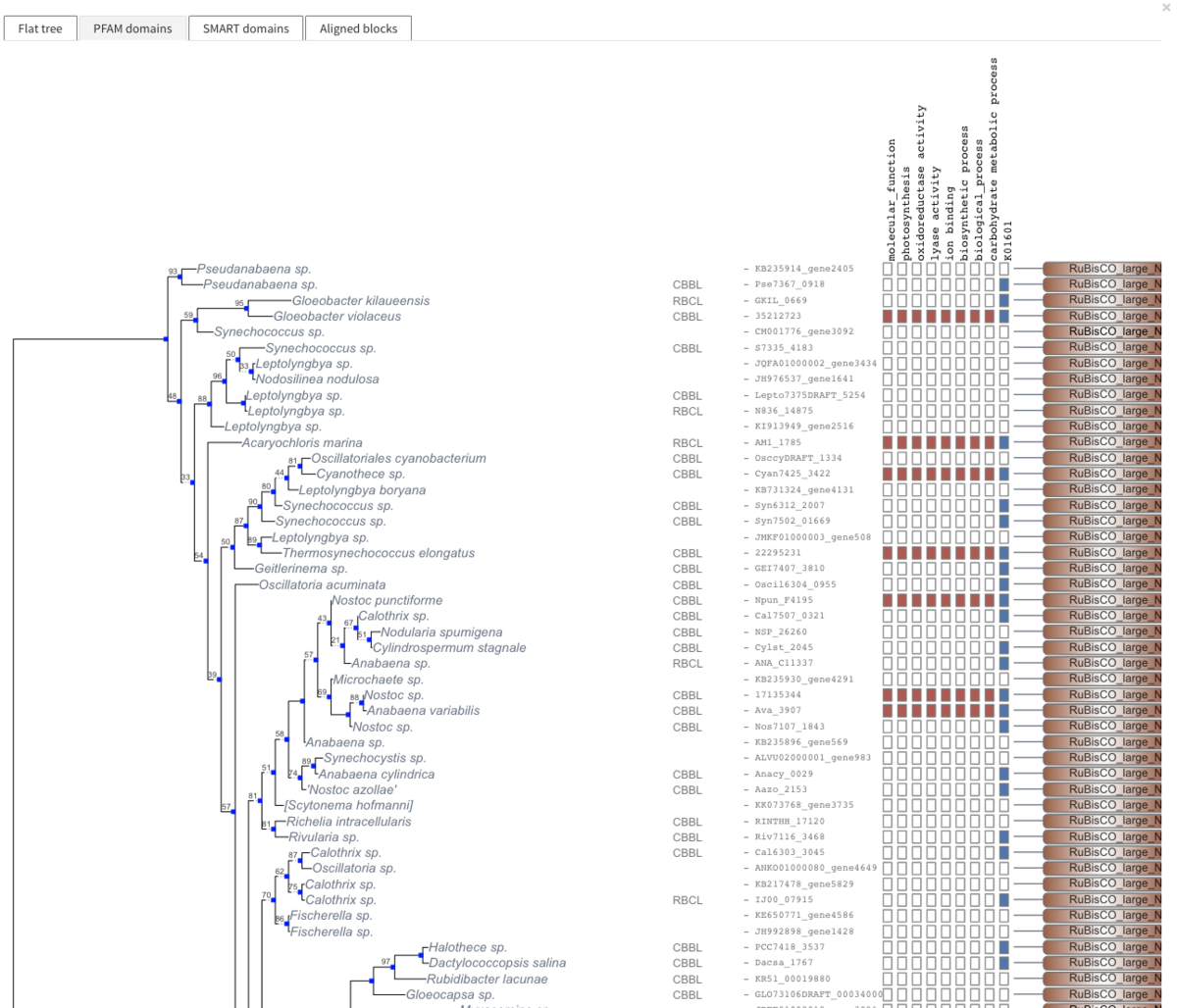
膜タンパク質は推定膜貫通領域のも表示される。

Phylogenetic Tree + aligned blocks

ダウンロードからはHMM modelファイルなどもダウンロードできるように変化している。
その他のサービスとして、Find orthologs by sequence similarityでは配列を入力してオロソロググループをサーチできます。
http://eggnog5.embl.de/#/app/seqscan
2022/06/25追記
MSAファイルやHMMプロファイルは幅広い分類群のタンパク質配列セットから作成されており、ダウンロードして利用できる。見るかぎりNCBI COGとeggNOG COGのIDは合致している。

引用
eggNOG 5.0: a hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses
Jaime Huerta-Cepas, Damian Szklarczyk, Davide Heller, Ana Hernández-Plaza, Sofia K Forslund, Helen Cook, Daniel R Mende, Ivica Letunic, Thomas Rattei, Lars J Jensen, Christian von Mering, Peer Bork
Nucleic Acids Res. 2019 Jan 8; 47
eggNOG 4.5: a hierarchical orthology framework with improved functional annotations for eukaryotic, prokaryotic and viral sequences
Huerta-Cepas J1, Szklarczyk D2, Forslund K1, Cook H3, Heller D2, Walter MC4, Rattei T5, Mende DR6, Sunagawa S1, Kuhn M7, Jensen LJ3, von Mering C8, Bork P
Nucleic Acids Res. 2016 Jan 4;44
eggNOG v4.0: nested orthology inference across 3686 organisms
Sean Powell,1 Kristoffer Forslund,1 Damian Szklarczyk,2 Kalliopi Trachana,3 Alexander Roth,2 Jaime Huerta-Cepas,4,5 Toni Gabaldón,4,5 Thomas Rattei,6 Chris Creevey,7 Michael Kuhn,8 Lars J. Jensen,9,* Christian von Mering, Peer Bork
Nucleic Acids Res. 2014 Jan 1; 42
eggNOG v3.0: orthologous groups covering 1133 organisms at 41 different taxonomic ranges
Sean Powell,1 Damian Szklarczyk,2 Kalliopi Trachana,1 Alexander Roth,3 Michael Kuhn,4 Jean Muller,5,6 Roland Arnold,7 Thomas Rattei,8 Ivica Letunic,1 Tobias Doerks,1 Lars J. Jensen,2,* Christian von Mering, Peer Bork
Nucleic Acids Res. 2012 Jan; 40
eggNOG v2.0: extending the evolutionary genealogy of genes with enhanced non-supervised orthologous groups, species and functional annotations
J. Muller,1 D. Szklarczyk,1,2 P. Julien,3 I. Letunic,1 A. Roth,4 M. Kuhn,1 S. Powell,1 C. von Mering,4 T. Doerks,1 L. J. Jensen, P. Bork
Nucleic Acids Res. 2010 Jan; 38
eggNOG: automated construction and annotation of orthologous groups of genes
Jensen LJ1, Julien P, Kuhn M, von Mering C, Muller J, Doerks T, Bork P.
Nucleic Acids Res. 2008 Jan;36
関連
参考
Question: What is the difference between eggNOG, COG and KEGG?