次世代シークエンシング(NGS)は、日常的な遺伝子検査のための強力かつ効率的で費用効果の高い臨床ツールとなっている。それは、遺伝子的に多くの遺伝病を解読することによって有用性が証明されており、これが最も顕著なのは癌である(Ardeshirdavani et al、2014)。ここ数年、マルチジェネパネル試験(論文より Hagemann et al、2014; Rehm、2013; Renkema、Stokman、Giles、&Knoers、2014)、whole-exome sequencing(Bamshad et al、 (Baird&Caldas、2013; Ellis et al、2012; Van Dijk、Auger、Jaszczyszyn、&Thermes、2014)。 1つのNGS実験で数百から数千もの変異が検出されているが(Rehm、2013; Renkema et al、2014)、現在の課題は病原性突然変異とその適切な解釈を特定することである。特定の情報を収集するためのデータベースは、煩雑で時間のかかるプロセスであり、これらのデータベースからのデータを有意義に解釈することができる。したがって、関連するデータベースを単一のインタフェース内にシームレスに統合できるワンストップのソフトウェアソリューションは、さまざまな注釈と解釈のための大きな助けになる。未処理のリードの処理と変異解析のために利用可能なソフトウェアツールがいくつかあるが、参照、視覚化ツールがまだ不足している(論文 表1)。効果予測とアノテーションツールには ANNOVAR(Yang&Wang、2015)、SnpEff(Cingolani et al、2012a)、VCF-Miner(Hart et al。、2016)、VAAST(Huat et al。、2010)、 WHOTIF(Ye et al、2016)、SnpSift(Cingolaniet al、2012b)、GEMINI(Paila、Chapman、Kirchner、&Quinlan、2013)などが挙げられる。 ANNOVAR、SnpEff、およびVAASTはグラフィカルユーザーインターフェイスを使用しないでゲノム効果の予測と機能割り当てを行うためのコマンドラインツールであり、生物学者やnonex-pertユーザーには使いにくいものである。これらのツールのどれも、サンガーの変異およびホットスポット突然変異分析をサポートしていない。さらに、これらのツールの各々は、最初の使用前にリファレンスゲノムアノテーションファイルを準備する必要とする。
VCF-Minerは、バリアントのアノテーションとフィルタリングのためのWebベースのアプリケーションであるが、公に利用可能なデータベース情報でバリアントのクロスリンクを提供するものではなく、ゲノムブラウザのバリアントを視覚化する可能性は持っていない。 wANNOVAR(Chang&Wang、2012)は、ANNOVARのような機能を提供するが関連するデータベースとのクロスリンクなどのいくつかの機能が欠けているWebベースのツールである。 wANNOVARでは、複数のファイルを一度にアップロードすることはできない。またローカルWebサーバーにはインストールできない。さらに、UCSCゲノムブラウザへのリンクは、バリアント座標ではなく、遺伝子レベルで行われる。これは、ヒト配列決定のための変異分析のみをサポートする。 VAASTは、変異型の分類と損傷遺伝子とその病原性の変異体の同定に有用なもう1つのコマンドラインツールであるが、データの解釈や決定支援に重要なpublicデータベースからの情報とクロスリンクする機能はない。WHATIFは、EnememblとClinVarのアノテーションを提供し、VCF形式の入力のみを受け入れるWindows専用ツールである。GEMINIは、VCF形式の入力を受け入れるバリアント注釈用のデータベースツールで、SangerおよびHotspotvariant解析のアノテーションは提供していない。要約すると、利用可能なツールの多くは、(1)さまざまなリソースから容易に入手可能な関連情報の統合、(2)経験の少ないユーザーにとって特に重要な直感的なグラフィカルなWebインターフェイス、(3)単一の入力ファイルを分析する能力(4)BED入力ファイルのサポート、(5)サンガー配列から得られた突然変異を分析するための適切なガイドライン、および(6)新しく同定された変異体および所定のホットスポットを共分析する能力、に欠けている。
アノテーションの解釈での上記の制限に対処するため、この論文ではDaMoldという新しいバイオインフォマティクスソリューションを紹介している。これは、NGS、サンガー、ホットスポットの亜種をフィルタリング、アノテーション、クロスリンク、可視化するための、強力で統合されたWebベースのツールである。DaMoldは、各変異体の効果、例えばコドンおよびアミノ酸変化を予測することができる。関連するゲノム、プロテオーム、臨床情報(論文 表2)とともに、以前の実験から既に報告された変異を含む37の臨床的に関連する公開データベースと各変異体とをクロスリンクする。 DaMoldは、4つのデータベースのUCSCゲノムブラウザ(Kent et al、2002)、Ensemblゲノムブラウザ(Kerseyet al、2010)、1000 Genomesブラウザ(1000 Genomes Project Con -sortium et al、2003)、NCBIバリエーションビューア(NCBI ResourceCoordinators、2015)のバリアントをシームレスに解釈することができる。DaMoldは、targeted resequencing、 gene panel sequencing、exome sequencing、およびwhole genome sequencingによって生成されたデータから変異体を分析、解明および解釈するために使用できる。
マニュアル
https://sourceforge.net/p/damold/wiki/DaMold_Manual/
組み込まれたデータベースについてもまとめられている。
DaMoldサーバー
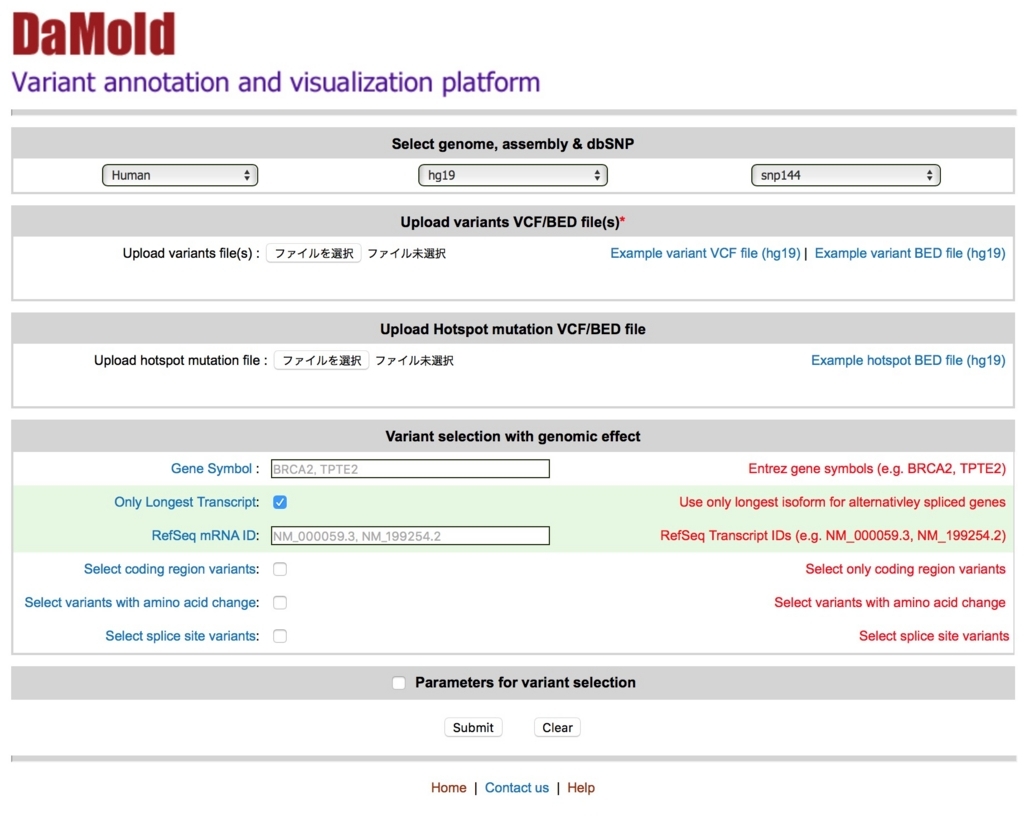
--Run--
exampleデータ"damold_variant.vcf"をアップしてみる。このデータは写真(↓)のExample variant VCF file (hg19) を押すとダウンロードできる。

サンガーなどで解析したHotspot変異のデータもあるなら、uploadする。VCFとBEDフォーマットに対応している(リンク)。サンプルデータをアップロードしてみる。
Gene SymbolにEnsemblスタイルで遺伝子名を記載すると、特定の遺伝子のコールのみ出力できる。
VCFにはカバレッジ情報も記載されているので(VCF紹介)、それに従いlow coverageのコールなどを排除したければ、Parameterの数値を変更する。デフォルトではフィルタリングは無しになっている。
- Minimum coverage of variant allele. Default 2
- Minimum total coverage. Default 4.
- Maximum total coverage. Default 300.
- Minimum minor allele frequency. Default 0.10.
--出力--
exampleなら10秒程度で結果が表示される。コール上の遺伝子名、mRNA名、プロテイン名、変異の場所、種類、各データベースへのリンクが表示される。

リストは、上のボタンからダウンロードできる。
右端のDatabaseより、Variant DBs、Disease DBs、 Genomic DBs、Protein DBs、 Pathways DBs、Genome Browsers、 Literature & GO DBs、Other DBsなどへジャンプできる。
ローカル環境へのインストール
local版はdockerで導入できる。
mac向けDockerコンテナのダウンロード (直リンク)
unzip DaMold_v1.0.zip
cd DaMold/
docker build -t damold .
#apache webserver を停止する(起動している場合)。
sudo /usr/sbin/apachectl stop
#damoldをランする。
docker run --name damold --restart always -p 80:80 -d damold
#動作状況を調べる
docker ps
virtual machine版も準備されています。
https://sourceforge.net/p/damold/wiki/DaMold_VM/
引用
DaMold: A data-mining platform for variant annotation and visualization in molecular diagnostics research.
Pandey RV, Pabinger S, Kriegner A, Weinhäusel A
Hum Mutat. 2017 Jul;38(7):778-787