2020 5/14 フィーチャー => 観測値に変更
全ゲノムの非環状プロットは、全染色体に沿って配列されたゲノムデータを自然に表現したものである。現在のところ、非環状の全ゲノム図を作成するために設計された専用のグラフィカル・ユーザー・インターフェース(GUI)は存在せず、既存のツールを使用するにはユーザーのコーディング作業が必要となる。また、このようなツールには新機能の追加などの改良が必要である。これらの課題を解決するために、非環状全ゲノム図をインタラクティブに作成するためのGUIとして、R/Shinyアプリケーション「shinyChromosome」を開発した。shinyChromosomeは、http://150.109.59.144:3838/shinyChromosome/, http://shinyChromosome.ncpgr.cn, https://yimingyu.shinyapps.io/shinyChromosome で展開されており、オンラインでの利用が可能になっている。shinyChromosomeのソースコードとマニュアルは https://github.com/venyao/shinyChromosome で自由に入手できる。
http://shinychromosome.ncpgr.cn
gallary(ファイルをダウンロードして使用することも可能)
http://shinychromosome.ncpgr.cn
インストール
依存
- R and RStudio installed (tested with R 3.5.0 and RStudio 1.1.419).
インストール手順はhttp://shinychromosome.ncpgr.cn/#tab-1306-1 に書かれています。ローカルにインストールするなら、コンフリクトを避けるためにrockerプロジェクトのrstudio等を使って下さい(紹介)。
データの準備
視覚化するには2つのファイルが必要になる。1つ目は染色体番号とサイズを示したテキストファイルで、もう1つはグラフにプロットされる観測値の値を示したファイルになる。順番に説明する。
1、染色体番号と染色体のサイズを示したテキストファイル
染色体をサイズを示したファイルを準備する。1列目には染色体ID、2列目には染色体の長さが記載されていなければならない。
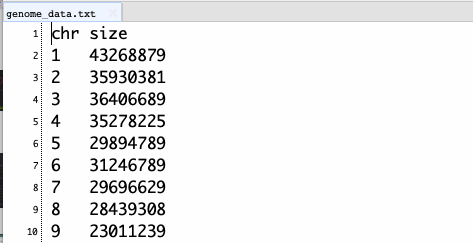
1行目はあっても無くてもよい。samtools faidxを使うと簡単に取り出せる。
samtools faidx Arabidopsis_thaliana.TAIR10.dna.toplevel.fa && \
cut -f 1-2 Arabidopsis_thaliana.TAIR10.dna.toplevel.fa.fai > genome.txt
2、観測値の値を示したテキストファイル
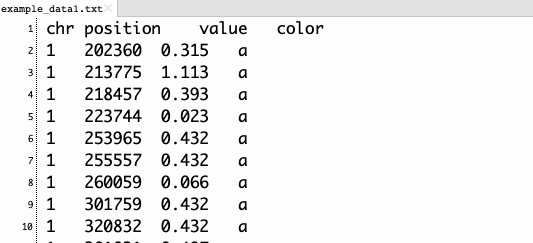
プロットしたい観測値の値を示したファイルを準備する。ラインデータ、ヒートマップデータなどを描画したい場合は、それぞれ指定のフォーマットで準備しなければならない。ここではプロットデータを例に挙げる。1列目にはフィーチャの染色体ID、2列目には観測値のポジション、3列目にはフィーチャの値が記載されていなければならない。また、任意で4列目にグループを指定する。color列の先頭行の名前は'color'でなければならない。
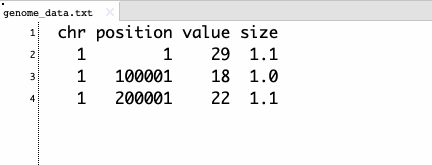
プロット図形を丸から変更するにはshapeのカラムを追加し、数値で指定する(R Plot PCH Symbols Chart)。先頭行の名前は'shape'でなければならない。

プロットサイズ調整にはsizeカラムを追加する。数値が大きいほどプロットサイズは小さくなる。
同時に複数指定する場合、4列目以降に順に記載する。
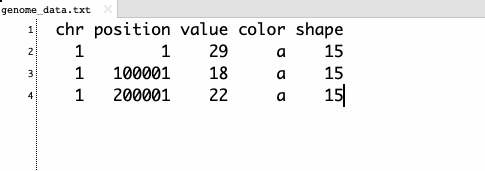
これらの設定はアプリ上でも変更できる。最小設定のままでも問題はない。プロット以外のファイルのフォーマットはhelpを確認して下さい。
http://shinychromosome.ncpgr.cn/
interactive creation of non-circular whole genome diagram にアクセスする。
single genome plotとtwo genome plotに分かれている。single genome plotから見ていく。

まずは染色体番号と染色体のサイズを示したテキストファイルを読み込ませる。
次に観測値のテキストファイルを読み込ませる。data1-10とあるのは複数の観測値を同時に指定してプロットするため。
ここでは1種類の観測値のみプロットする。Data1のみ選択し、DisableからUpload input dataに切り替える。

切り替えた。ここではtrack1にpoint ファイルとして観測値ファイルをロードする。
ここではExampleデータをクリックして、Exampleテキストをダウンロードした。それからBrowseボタンでロードした。
一番下のGoボタンをクリックすると作図される。
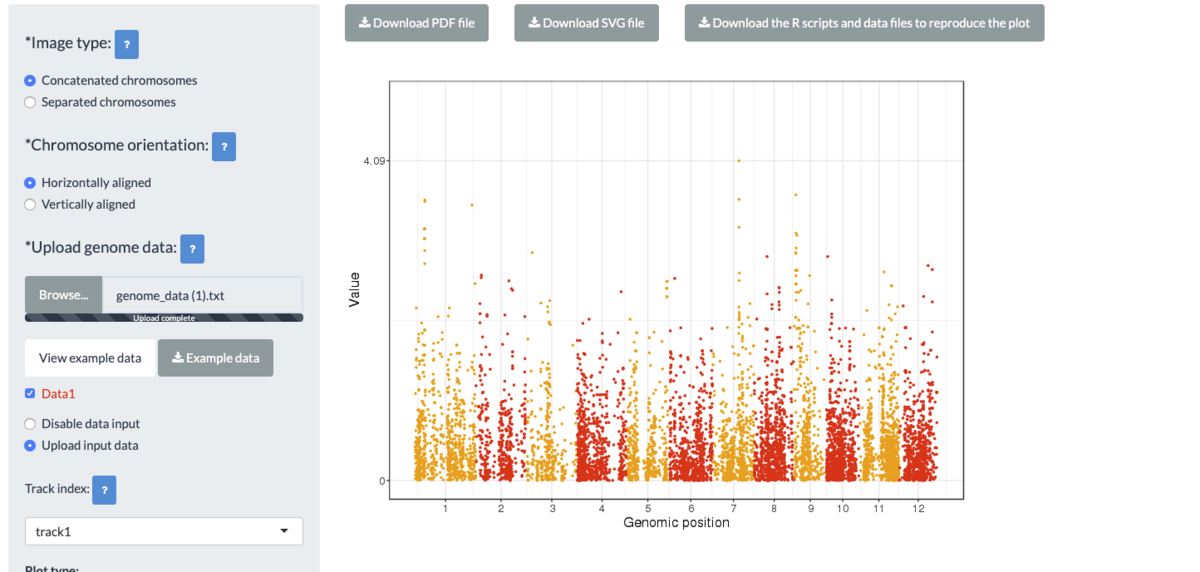
メニューの一番上のimages sizeをSeparated chromosomesに切り替えてみる。

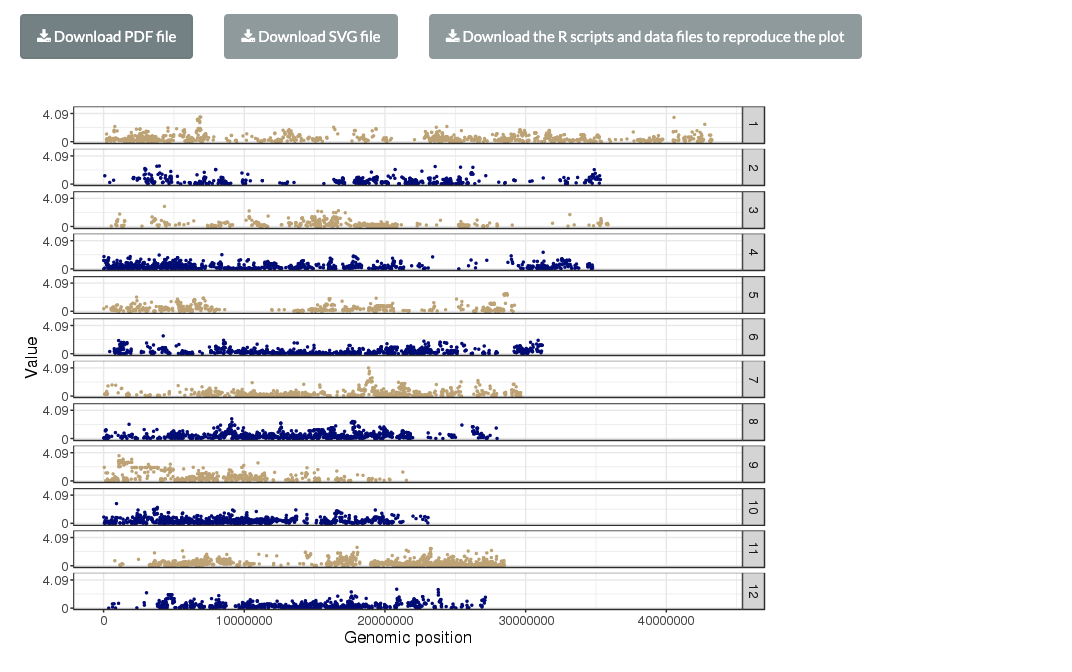
GoGoボタンをクリックすると再描画される。染色体ごとに分けて作図された。
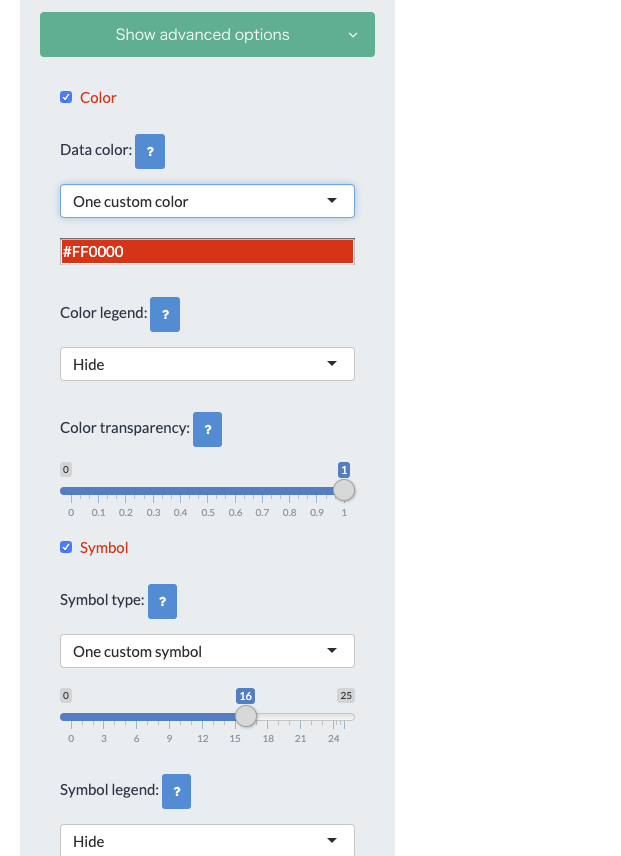
Advanced optionから プロットのパラメータを変更できる。
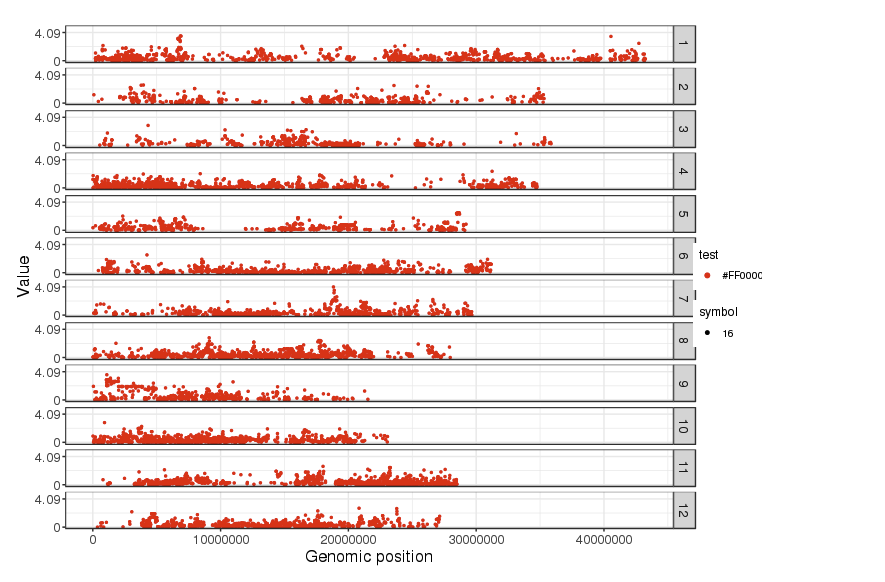
プロットの色をランダムから赤に変更し、legendも追加した。
data1と2にチェックを入れて、それぞれtrack1とtrack2にした。このtrack1とか2というのはデータの順番を表す。
(data2はplotからlineに変更)
それからプロットした。指定した通りdata2は線で表現された。
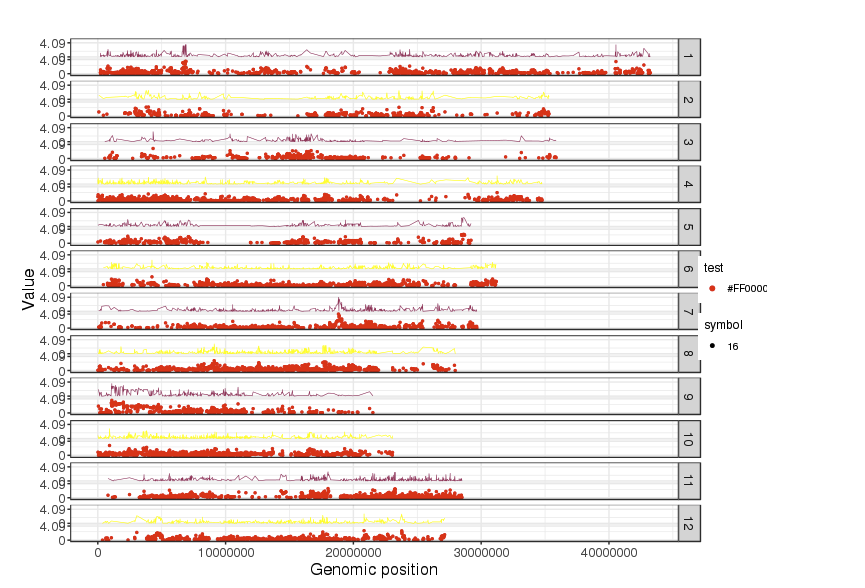
向きを垂直にして

3つのデータをプロットした。data3のみlineにしている。
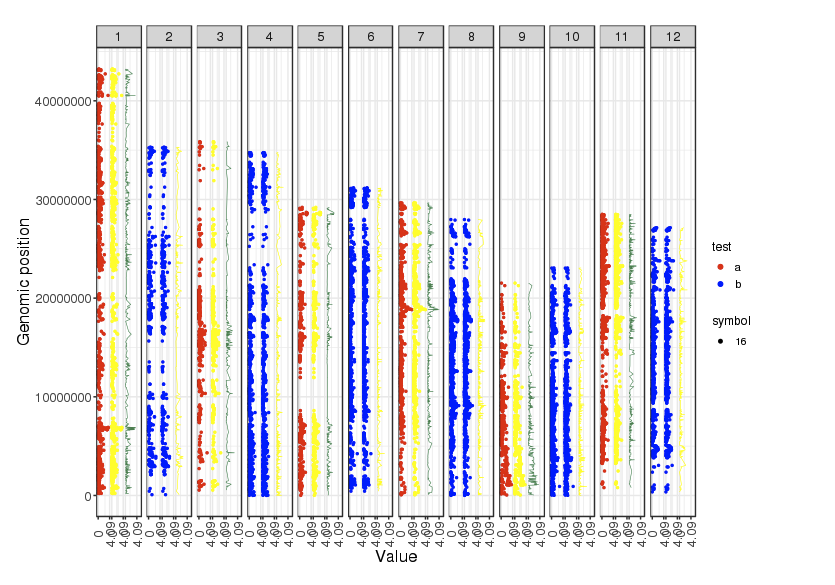
向きを元に戻し、Concatenated chromosomesスタイルで3つのデータをプロットした。
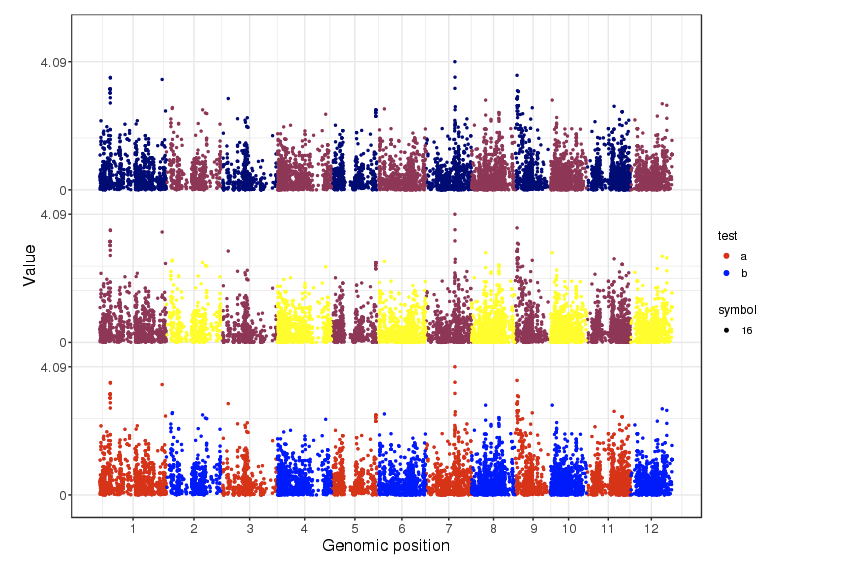
Vertically alignedで4つのデータをプロットした。

もう1つのモードはtwo genome plotになる。
2genome間で比較する。2つのゲノムそれぞれの染色体のサイズを示したテキストファイルと、フィーチャファイルの合計3ファイルが必要。

読み込ませる。ここではexample fileをダウンロードして指定した。
出力

18種類のテーマが用意されている。
theme11
theme 18
gallaryから好みの作図設定を探して下さい。
http://shinychromosome.ncpgr.cn
引用
shinyChromosome: An R/Shiny Application for Interactive Creation of Non-circular Plots of Whole Genomes
Yiming Yu, Wen Yao, Yuping Wang, Fangfang Huang
Genomics Proteomics Bioinformatics. 2019 Oct; 17(5): 535–539

