2020 5/15 snpEffのデータベース追加方法を追記, step2とlogの写真差し替え、dockerのコマンド追加、補足修正
2020 5/16 出力の写真差し替え、レポート追加
2020 10/11,10/12 インストールコマンド修正
2021 9/21 タイトル修正
ニューヨーク大 - Center for Genomics and Systems Biology (CGSB)のMohammed Khalfanさんの記事より(記事一覧)
2016年に公開されたバリアントコールパイプラインの投稿の更新版を公開する。この更新版はGATKを採用しており、コンテナ化されたNextflowスクリプトとしてGitHubで公開している。
次世代シーケンシングデータから、一塩基多型(SNP)やDNAの挿入・欠失(indel)を含むゲノムのバリアントを特定することは、科学的発見の重要な部分である。ニューヨーク大学ゲノム・システム生物学センター(CGSB)では、この作業が多くの研究プログラムの中心となっている。例えば、カールトン研究室では、SNPデータを解析してマラリア寄生虫Plasmodium falciparumとPlasmodium vivaxの集団遺伝学を研究している。グレシャム研究室ではSNPとindelデータを用いて酵母の適応進化を研究し、化学科のルポリ研究室ではバリアント解析を用いて大腸菌の抗生物質耐性を研究している。
この研究を促進するために、研究者が正確かつ迅速にシーケンスバリアントを同定し、アノテーションを行うことができるバイオインフォマティクスパイプラインが開発された。このパイプラインは、Genome Analysis Toolkit 4(GATK4)を使用してバリアントコールを行う。これはBroad Instituteによって概説されたバリアント発見解析のベストプラクティスに基づいている。SNPが同定されると、SnpEffを使用してアノテーションを行い、バリアント効果を予測する。
このパイプラインは、クローン性のサンプル、すなわち単一の個人のサンプルのバリアントをコールすることを目的としている。 これらのサンプルにおけるバリアントの頻度は、1(ハプロイドまたはホモ接合体二倍体の場合)または0.5(ヘテロ接合体二倍体の場合)であると予想される。 ヒト腫瘍や混合微生物集団など、対立遺伝子頻度が0から1の間で連続的に変化するような不均一なサンプルのバリアントをコールするには、研究者はサブクローナルイベントを同定するために設計されたGATK4 Mutect2を使用する必要がある(ワークフローは近日中に公開予定)。
Base Quality Score Recalibration (BQSR)は、ベースクオリティスコア(Phredスコアとして測定)に対するテクニカルなばらつきの影響を最小化することを目的とした、正確なバリアント検出のための重要なステップである。オリジナルのパイプライン(リンク)と同様に、このパイプラインでは「ゴールドスタンダード」と呼ばれる SNPS とindelのセットが BQSR のために利用できないことを前提としている。 ゴールドスタンダードがない場合、パイプラインは BQSR を実行せずにバリアントを検出する最初のステップを実行し、識別された SNP を BQSR の入力として使用してから再びバリアントをコールする。このプロセスはブートストラップと呼ばれ、ゴールドスタンダードが利用できない場合のバリアント発見解析のためのBroad Instituteのベストプラクティスで推奨されている手順である。
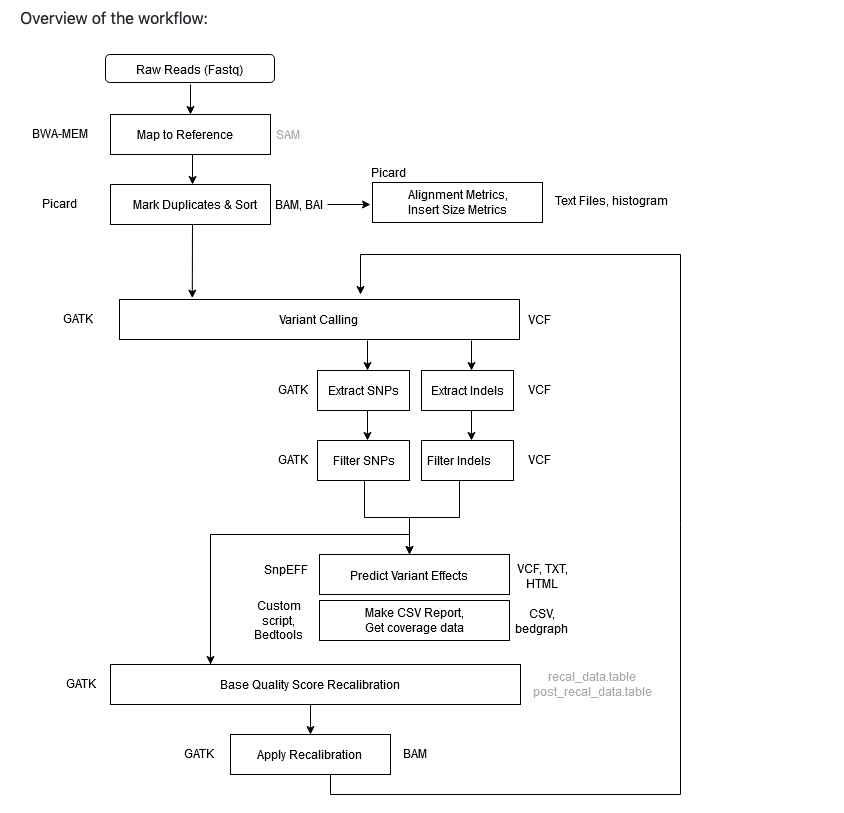
下のフローチャート図のようなパイプラインとなる。パイプラインの内容自体は以前紹介したv3から変化していない。
githubより転載
インストール
docker imageをビルドしてmac mini2018でテストした。
本体 Github
git clone https://github.com/gencorefacility/variant-calling-pipeline-gatk4.git
cd variant-calling-pipeline-gatk4/
#docker imageのビルド
docker build ./ -t cgsbgatk4
一番下に補足として説明を追加した。
実行方法
1、bwaのindex、fasta.fai、fasta.dictファイルをリファレンスのFASTAファイルと同じパスに用意しておく。
# #genome index
samtools faidx ref.fa
#ref.dict
picard CreateSequenceDictionary R=ref.fa O=ref.dict
#bwa index
bwa index -a is ref.fa
#snpeff database
#arabidopsis thalianaの場合
#利用可能なデータベースをサーチ
java -jar /data/snpEff_latest_core/snpEff/snpEff.jar databases | grep -i Arabidopsis|cut -f 1
#追加する
java -jar /data/snpEff_latest_core/snpEff/snpEff.jar download \
-v Arabidopsis_thaliana \
-c /data/snpEff_latest_core/snpEff/snpEff.config
- -a <STR> BWT construction algorithm: bwtsw, is or rb2 [auto]
2、nextflow.configを書き換える。/dataで作業するとして以下のように直した。

fastqは/data/fastqに、レファレンス配列とindexは/data/reference/に置く。
3、run
#1 main.nfとスクリプトを作業ディレクトリにコピー。
cp variant-calling-pipeline-gatk4/main.nf .
cp variant-calling-pipeline-gatk4/bin/* .
#nextflow.configもコピーしておく
#2作業ディレクトリでdockerを立ち上げる
docker run -itv $PWD:/data/ -w /data cgsbgatk4
cp calling-pipeline-gatk4/bin/* /usr/local/bin/
#3 run
nextflow run main.nf -c nextflow.config
#3 run and export the report
nextflow run main.nf -with-report
#resume from previous run
nextflow run main.nf -resume
#configファイルを使わずラン時にパラメータを全指定する。
nextflow run main.nf \
--reads "/path/to/reads/*_n0{1,2}_*.fastq.gz" \
--ref "/path/to/ref.fa" \
--outdir "/scratch/user/gatk4/" \
--snpeff_db "athalianaTair10" \
--pl "illumina" \
--pm "illumina"
進捗が表示される。
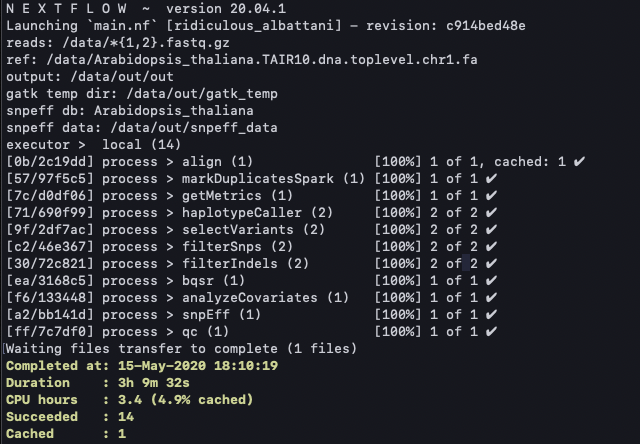
無事完了した。1度目はsnpEffのランに失敗したが、フルパス指定に変更することでエラー回避した(補足参照)。
出力

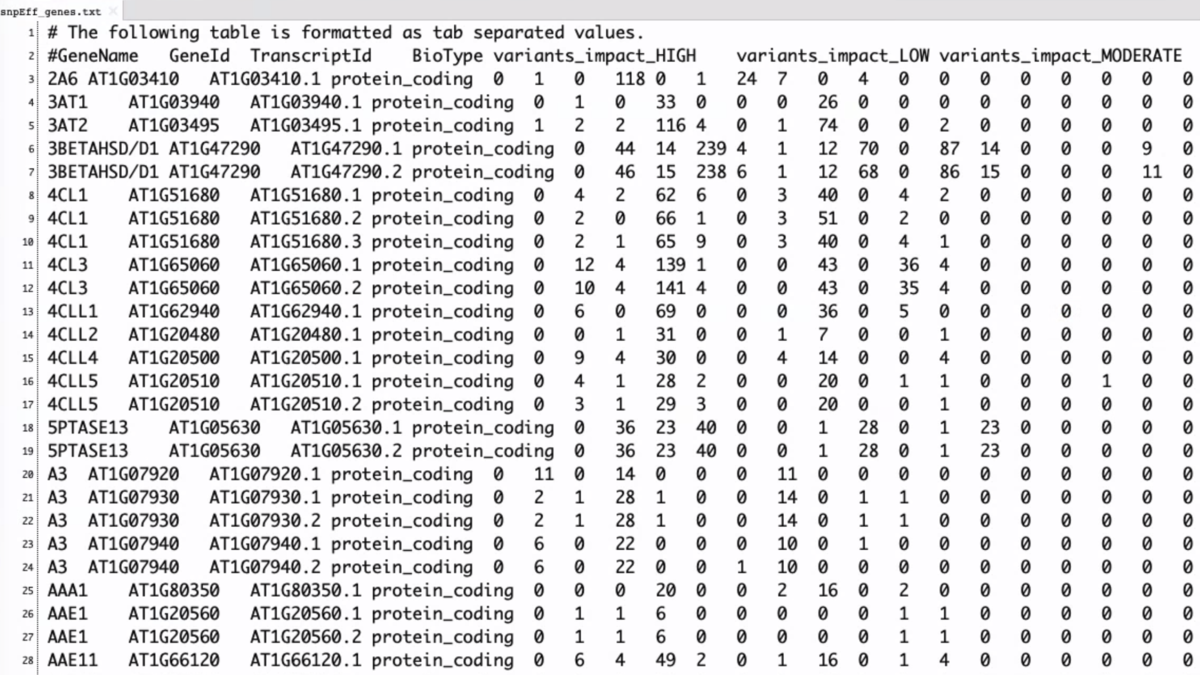
snpEff_genes.txt
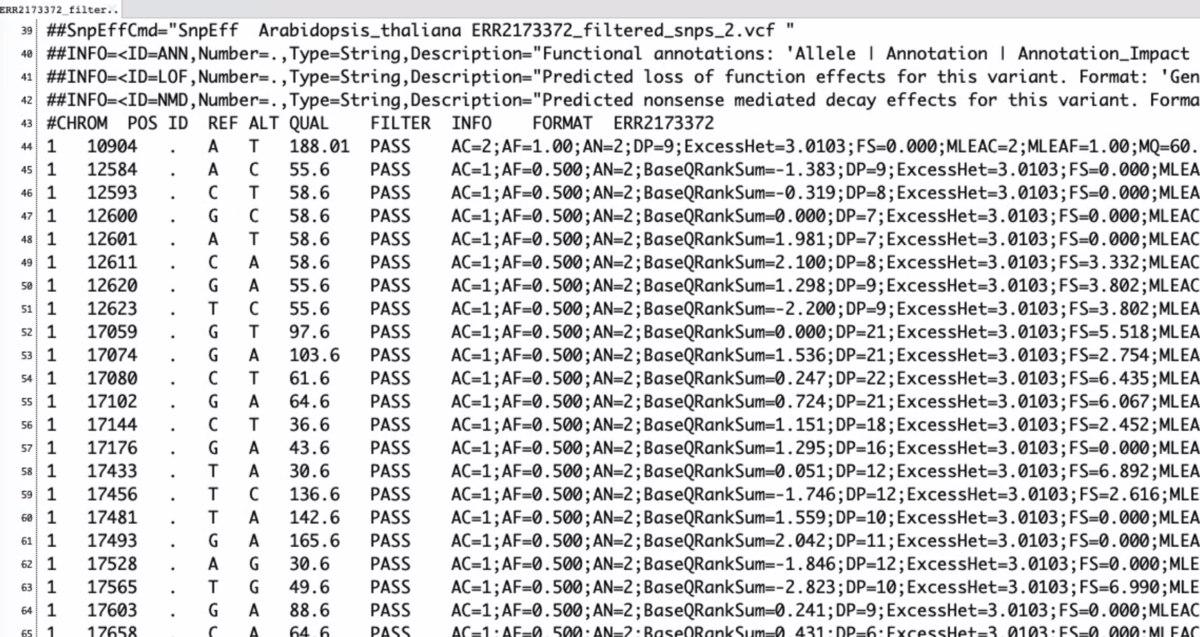
ERR2173372_filtered_snps.ann.vcf
snpEffのサマリーレポート(いくつか端折ってます)
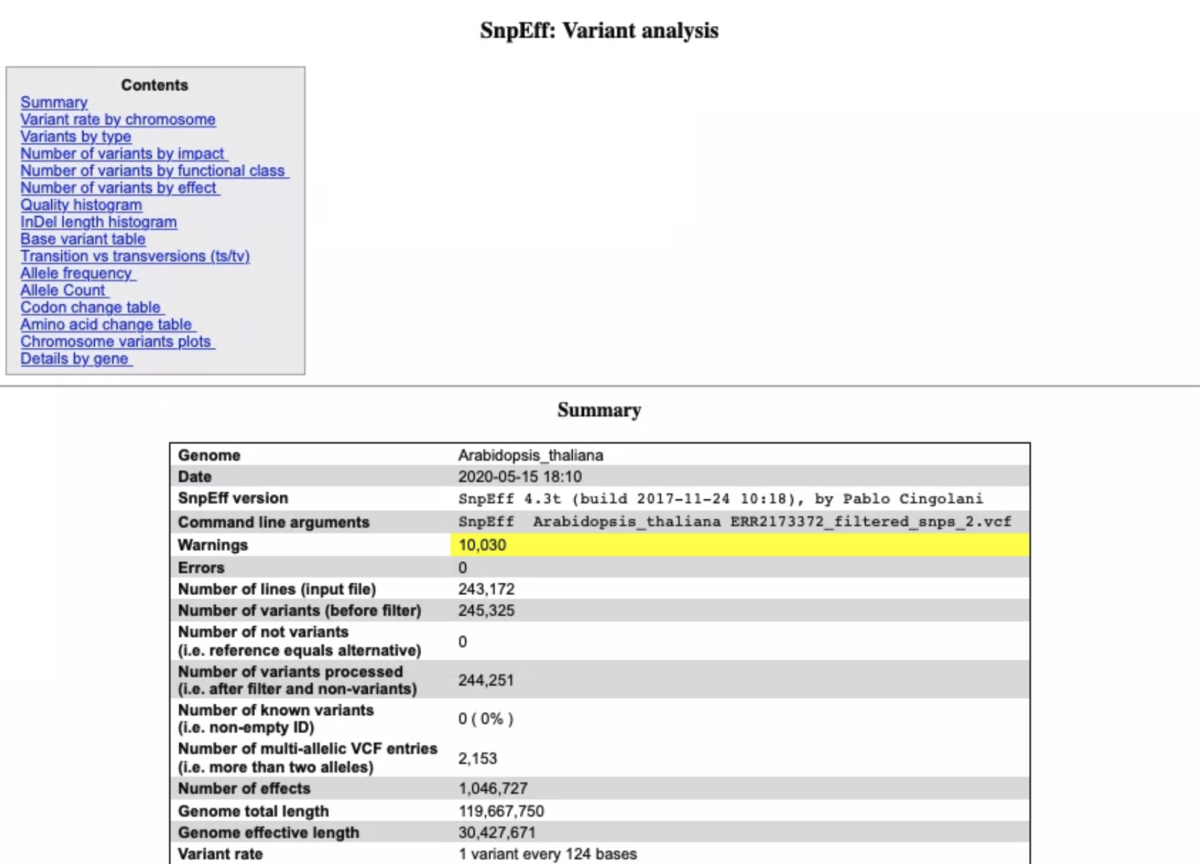
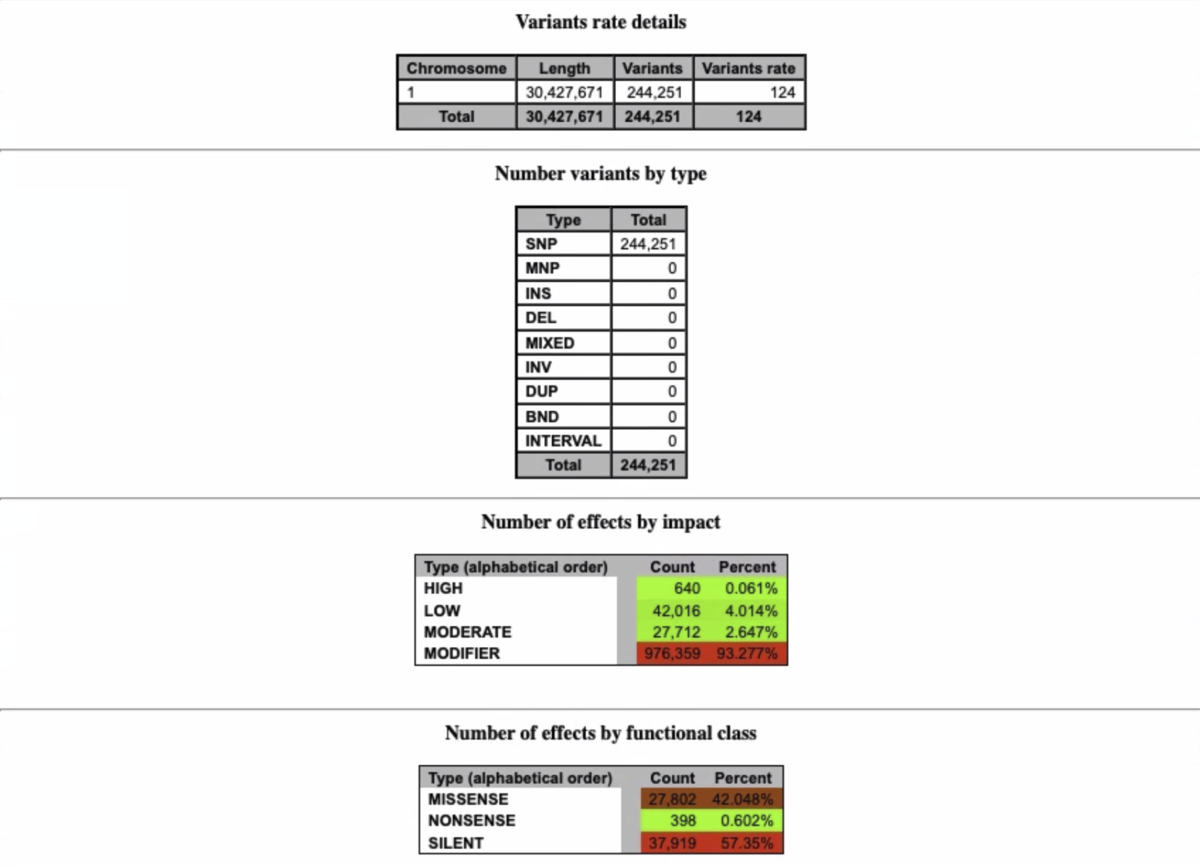

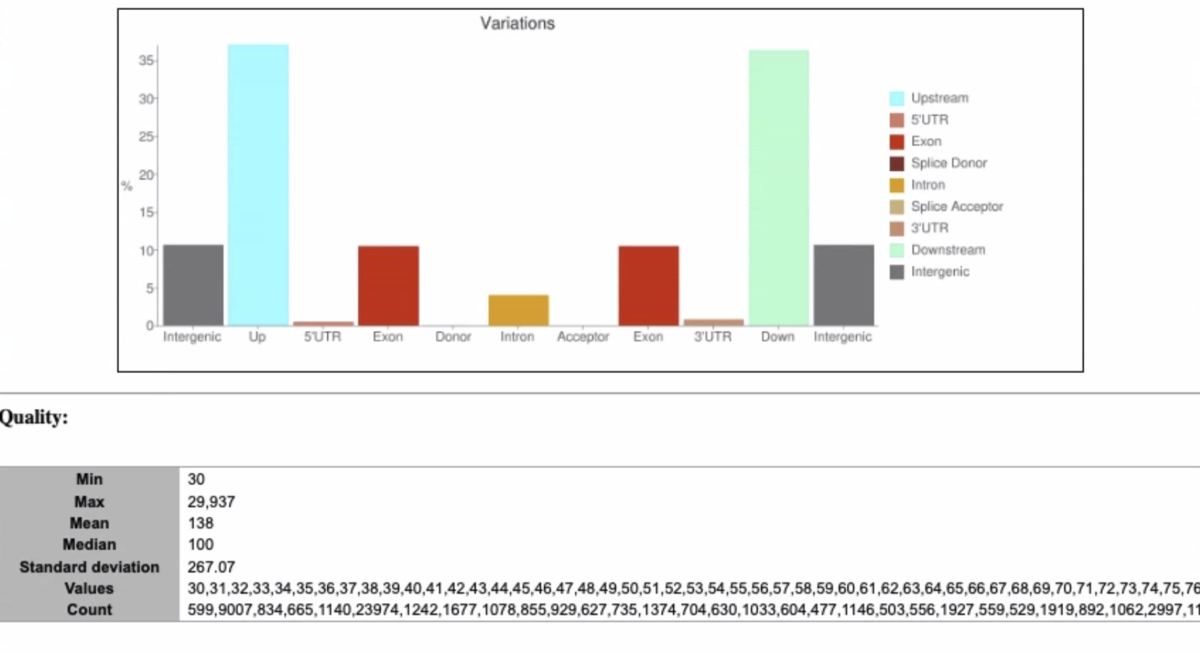

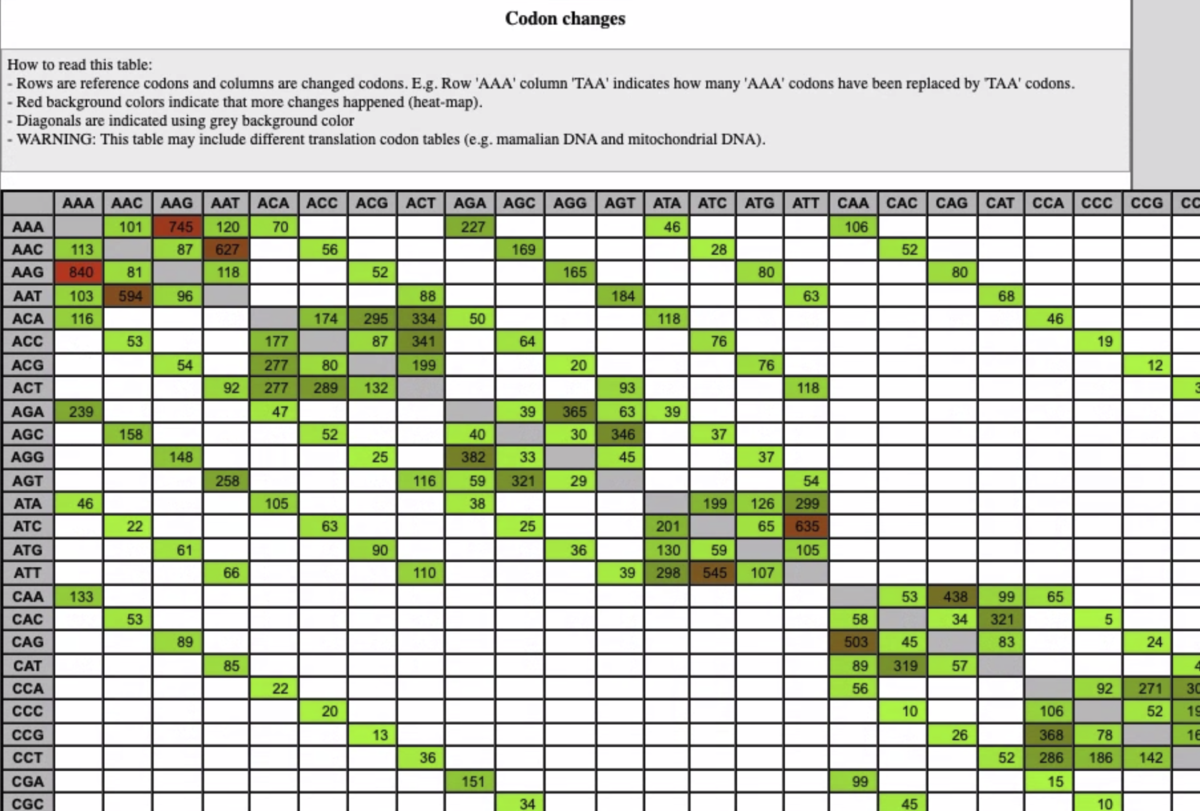


gatk3.8やgatk4についてベンチマークしたペーパーも出ています。
https://bmcbioinformatics.biomedcentral.com/articles/10.1186/s12859-019-3169-7
チャンクに分割して、複数ノードで並列計算する。
https://bmcbioinformatics.biomedcentral.com/track/pdf/10.1186/s12859-019-3169-7
GPUを使う。
ホタペンさん教えていただきありがとうございます。
引用
Variant Calling Pipeline using GATK4 – Genomics Core at NYU CGSB
参考
Changing workflows around calling SNPs and indels
関連
補足
エラーが出たので以下のように対処しました。動作を保証するものではありませんが、参考までに残しておきます。
1、dockerfileで導入されたjavaを消す。
yum remove java -y
さらに
which java
= > 表示されるjavaを消す。
不要
2、condaを使ってnextflowを導入すると依存しているjavaも導入されるが、これのせいで話がややこしくなる。nextflowはcondaを使わず導入する(link)。
wget -qO- https://get.nextflow.io | bash
cp nextflow /usr/local/bin/
3、javaのJDSK1.8 のrpmをダウンロードしてインストールする。複数あるが古いものを選んだ。ダウンロードにはログインが必要
https://www.oracle.com/java/technologies/javase/javase8-archive-downloads.html
# ダウンロードしたパッケージをインストール
rpm -ivh jdk-8-linux-x64.rpm
rpm -ivh jre-8u11-linux-x64.rpm
#check
java
java -version
#間違えた時にアンインストールするなら
rpm -e jre
4、作業ディレクトリにsnpEff_latest_core/をダウンロードし、(すでに導入されているはず)
main.nfの352行目のsnpEff変数指定を、snpEff のバイナリ(実行ファイル)であるsnpEff.jar .jarのフルパスに変更。
java -jar /apps/snpeff/4_3i/snpEff.jar -v \
5、そのほか、気に入らないパラメータがあればmain.nfの該当部分を直接書き換えも良い。ここではbwaのスレッドを増やすため、main.nf52行目を
-t 12 \
に書き換えた。
6、小さなデータ、または小さな染色体1つでテストランして動作確認する。