ほとんどの環境微生物が難培養性であることを考えると、microbial ecologyの分野では、metagenomicsは全コミュニティの機能を調べる手段に由来していた(論文より Handelsman、2004; Kunin et al、2008; Teeling and Glockner、2012)。研究者は、微生物群全体からのDNAのショットガンシーケンシングを行い、得られたメタゲノムをコミュニティ全体の遺伝子プールからのサンプルとして扱い、それらの集合的機能潜在性の像を再構成するか、またはアセンブルして個々の微生物ゲノムを抽出する( "binning")。メタゲノムからのゲノムのビニングは、比較的低い多様性のサンプル(Tyson et al、2004; Woyke et al、2006)を用いて過去に行われてきたが、ハイスループットシークエンシングの最近の進歩により、より多様化したコミュニティから個々のゲノムを隔離することが現実的になった。
ビニングの戦略は、(i)配列の内部統計的性質、例えばk-mer頻度(Teeling et al、2004)、(ii) (Kembel et al、2011)または全ヌクレオチドデータベース(Kumar et al、2013)、または(iii)異なる配列間のカバレッジ/カバレッジの生物学的または技術的変動(Albertsen et al、2013; Imelfort et al、2014; Nielsen et al、2014)、self-organizing maps(Dick et al、2009)、補間マルコフモデル(Strous et al、2012)、 expectation maximization(Wu et al、 2014)、およびk-medioids clustering(Kang et al。、2015)がある。いずれも多数のサンプルからの微生物ゲノムを自動的かつ高スループットでビニングすることを最終目的とする。
ビジュアライゼーションは、通常、データ探査の第一歩であり、現在の多くの方法がunsupervisedのビニングに精通しているにもかかわらず、いまだメタゲノミクスツールキットの重要な部分となっている。host-symbiont systemsや微生物のコンソーシアムで発生するような、多様性の低い、カバレッジの高いサンプルでは、カバレッジGCプロットから手動でビンを定義することは可能かもしれない。リードのカバレッジを各コンティグのGC率に対してプロットする、または異なるライブラリ由来コンティグについてカバレッジ同士のプロットを行う。同じゲノムに由来するコンティグは、類似の配列組成(GC%で表される)および存在量(カバレッジで表される)を有すると予想されるので、これらのプロットにおいて一緒に集まるべきである。したがって、各クラスターは単一の推定ゲノムビンを表す。このようなヒューリスティックがAlbertsen et al(論文 紹介)によって用いられ、テトラヌクレオチド頻度の主成分分析およびプロット上に重ね合わされたマーカー遺伝子からのさらなる分類学的情報の助けを借りて、活性汚泥コミュニティから未培養バクテリアのほぼ完全なゲノム12個が抽出された。また、ビジュアライゼーションすることは、事後検証、不完全なビニングによる潜在的なアーティファクトの発見、自動化された方法の検証やトラブルシューティングに役立つ。
しかし、既存のビジュアライゼーションツールは、主に特定のビニング方法またはパイプラインに添付されている。したがって、その適用は比較的狭い(論文より 表1)。 Albertsen rt al(2013)はプロットと手作業によるビニングのためのスクリプト(統計コンピューティング言語Rで書かれている)を利用できるようにしたが、これらは新しいデータセットに対して大幅なカスタマイズが必要である。したがって、メタゲノムのビニングに関連するデータを統合し、エンドユーザが直感的な方法でデータの探索と可視化を実行できるようにするツールを提供することが著者らの動機だった。
マニュアル
https://github.com/kbseah/genome-bin-tools/wiki
インストール
mac osx 10.13に導入したが、一部ツールはcent osをつかった。
optional
https://github.com/kbseah/genome-bin-tools
git clone https://github.com/kbseah/genome-bin-tools.git
cd /genome-bin-tools/R_source_package/
R #Rを立ち上げる
> install.packages("sp")
> install.packages("plyr")
> install.packages("gbtools_2.4.5.tar.gz",repos=NULL,type="source") #2.4.5の場合
> library(gbtools) #読み込み
> help(gbtools)
gbtools package:gbtools R Documentation
Interactive visualization of metagenome assemblies in R
Description:
gbtools lets you visualize metagenome assemblies by GC-coverage or
differential coverage plots, and interactively bin them.
Details:
See website for more details.
Author(s):
Brandon Seah, <email: kbseah@mpi-bremen.de>
References:
<URL: https://github.com/kbseah/genome-bin-tools>
ラン
step1 カバレッジ計算
ここではすでに全シーケンスデータを混ぜてde novoアセンブリを完了しているものとする。このメタゲノムのアセンブリツールに指定はない。
アセンブリで得たFASTAファイルを基に、BBtoolsを使いカバレッジを計算する。BBtoolsがなければ"brew install bbtools"でインストールしておく。
ライブラリAのカバレッジ計算。出力はSampleG1.covstatsとする。
bbmap.sh ref=assembly.fasta nodisk in1=sample1_f.fq.gz in2=sample1_r.fq.gz covstats=SampleG1.covstats
ライブラリBのカバレッジ計算。出力はSampleA2.covstatsとする。
bbmap.sh ref=assembly.fasta nodisk in1=sample2_f.fq.gz in2=sample2_r.fq.gz covstats=SampleA2.covstats
ライブラリAとBは、例えば同じサンプルを異なる方法でDNA抽出したシーケンスデータ。AとBのシーケンスデータを両方使いde novoアセンブリを実行し、それを上記で使っている。
ランが終わると、カバレッジ、GC、lengthなどが出力される。

最近のバージョンのBBtoolsは、先頭行にコメントアウト"#"を使っているが、今回は邪魔になるので消しておく。右端にはMedian_foldとStd_Devのカラムがあるかもしれないが、この解析では使わない。
step2 マーカー遺伝子検出 (optional)
AMPHORA2でマーカー遺伝子を検出する。AMPHORA2がなければgithubからcloneする。
#AMPHORA2のインストール
git clone https://github.com/martinwu/AMPHORA2.git
cd AMPHORA2/
sudo perl preinstall.pl #依存でないものが導入される。
export AMPHORA2_home=/home/foo/AMPHORA2
source ~/.bash_profile #またはbashrc
chmod +x /home/foo/AMPHORA2/Scripts/*
AMPHORA2が準備できたら、はじめにmarker protein配列を検出する。生物種が想定できなければ"-DNA"、限定するなら"-Bacteria"か"-Archaeal"をフラグに立てる。AMPHORA2/Marker/にデータベースがある。
cd AMPHORA2/Scripts/
mkdir output && cd output/
perl ../MarkerScanner.pl -DNA ~/data/metagenome_assrmbly.fasta
#各marker proteinのpepファイルが出力される。続いてmarker protein配列のアライメントを行い、マスクしてトリミングする。
perl ../MarkerAlignTrim.pl -WithReference -OutputFormat phylip
各marker proteinのalnと maskファイルが出力される。maker proteinの結果からphylotypeをアサインする。
perl ../Phylotyping.pl -CPUs 12 > phylotype.result
ここでgbtoolsに戻る。accessory_scripts/parse_phylotype_result.plを使い、Phylotyping.plの結果をパースする。
cd ~/genome-bin-tools/accessory_scripts/
perl parse_phylotype_result.pl -p phylotype.result > phylotype.result.parsed
step3、rRNA検出 (optional)
phyloFlashのSSU rRNAデータベースを使い、rRNAを検出する。まずphyloFlashのデータベースを構築する。phyloFlashはBBtools、bowtie、barrnap(紹介)、vsearchを使う。なければbrewで導入しとく。"brew install bbtools vsearch barrnap bowtie"。
#phyloFlashのデータベース準備
wget https://github.com/HRGV/phyloFlash/archive/pf3.0b1.tar.gz
tar -xzf pf3.0b1.tar.gz
cd phyloFlash-pf3.0b1
./phyloFlash_makedb.pl --remote
番号のディレクトリ(テスト時は132/)の中にSILVAのデータベースfastaができる。このファイルを指定して、gbtoolsのaccessory_scripts/get_ssu_for_genome_bin_tools.plを走らせる。
perl get_ssu_for_genome_bin_tools.pl -dphyloFlash-pf3.0b1/132/SILVA_SSU.noLSU.masked.trimmed.fasta -c 10 -a metagenome_assrmbly.fasta -o output
ssuRNAが検出されたcontigのリストファイル <output_prefix>.ssu.tabができる。これをgbtoolsに使用する。
step4、tRNA検出 (optional)
tRNAscan-SEサーバーにアクセスする。
メタゲノムアセンブリのFASTAを指定して、tRNA探索を実行する。解析は10分前後かかる。ラン後のテキストファイルをダウンロードする。ファイル名はtrna.tabとする。
optional step
付属スクリプトでstep1-4のファイルのチェック(禁則文字がないか)
perl input_validator.pl --covstats FILE1,FILE2,FILE3 \ # Covstats files
--mark MARK1,MARK2 \ # Marker taxonomy files
--ssu SSU \ # SSU taxonomy file
--trna TRNA \ # tRNA gene prediction file
作業がやり直しになってしまうので、de novo アセンブリのFASTAを得たら、すぐヘッダーをチェックしたほうがよいかもしれない。
step5、gbtoolsによるVisualizationとbinning
準備がおわったら、gbtoolsで結果を可視化する。ここからR内で作業する。
R #Rに入る。
> library(gbtools)
> library(sp)
> library(plyr)
ここではテストデータで手順を確認する。読み込むのはAlbertsen et alと同じ、2種類の抽出法でシーケンスされたというシーケンスデータになる。アセンブリしてstep1-4の分析が終わっている。
# SSU gene annotations
> column -t example_data/Olavius_example/olavius_metagenome.ssu.tab |head
$ column -t example_data/Olavius_example/olavius_metagenome.ssu.tab |head
scaffold SSUid SSUgenbankid Superkingdom Phylum Class Order Family Genus Species pid alnlen eval
contig-100_1055 contig-100_1055:1134-2950(+) AF411870.1.1810 Eukaryota Opisthokonta Holozoa Metazoa Animalia Annelida Tubificidae 99.8 1809 -1
contig-100_1182 contig-100_1182:199-1249(+) AF328856.1.1497 Bacteria Proteobacteria Gammaproteobacteria Gammaproteobacteria Incertae Sedis Unknown Family Arenicella Olavius algarvensis sulfur-oxidizing endosymbiont 99.9 1015 -1
contig-100_2324 contig-100_2324:1-1275(-) AJ620497.1.1523 Bacteria Proteobacteria Deltaproteobacteria Desulfobacterales Desulfobacteraceae uncultured Olavius algarvensis Delta 4 endosymbiont 99.9 1269 -1
# Marker genes
> column -t example_data/Olavius_example/amphora2_results.tab |head
$ column -t example_data/Olavius_example/amphora2_results.tab |head
scaffold markerid gene Superkingdom Phylum Class Order Family Genus Species
contig-100_1020 contig-100_1020_29 dnaG Bacteria Proteobacteria Deltaproteobacteria Desulfobacterales Desulfobacteraceae Desulfobacterium Desulfobacterium autotrophicum
contig-100_505 contig-100_505_94 dnaG Bacteria Tenericutes Mollicutes Mycoplasmatales Mycoplasmataceae Mycoplasma Mycoplasma synoviae
contig-100_91717 contig-100_91717_10 dnaG Bacteria Fibrobacteres Fibrobacteria Fibrobacterales Fibrobacteraceae Fibrobacter Fibrobacter succinogenes
contig-100_328 contig-100_328_161 dnaG Bacteria Proteobacteria Gammaproteobacteria Legionellales Coxiellaceae Coxiella Coxiella burnetii
contig-100_3045 contig-100_3045_43 dnaG Bacteria Proteobacteria Gammaproteobacteria Chromatiales Chromatiaceae Allochromatium Allochromatium vinosum
contig-100_88109 contig-100_88109_5 dnaG Bacteria Spirochaetes Spirochaetia Spirochaetales Spirochaetaceae Spirochaeta Spirochaeta smaragdinae
contig-100_220 contig-100_220_33 dnaG Bacteria Proteobacteria Deltaproteobacteria Desulfobacterales Desulfobacteraceae Desulfococcus Desulfococcus oleovorans
contig-100_92 contig-100_92_330 frr Bacteria Proteobacteria Gammaproteobacteria Chromatiales Halothiobacillaceae Halothiobacillus Halothiobacillus neapolitanus
contig-100_91097 contig-100_91097_8 frr Bacteria Bacteroidetes Bacteroidetes Order II. Incertae sedis Rhodothermaceae Salinibacter Salinibacter ruber (Salinibacter ruber)
uesaka-no-Air-2:example_data kazumaxneo$
step1-4で得たファイルをインポートする。contigのカバレッジファイルは必須で、step2−4は任意。(詳細)。
> d <- gbt (covstats=c("SampleA2.covstats","SampleG1.covstats"),
ssu="olavius_metagenome.ssu.tab",
mark=c("amphora2_results.tab","blobology_results.tab"),
marksource=c("amphora2","blob"),
tRNA="trna.tab")
ssu = "SSU rRNA data"
mark = "Taxonomic marker data" (phylotype.result.parsed)
tRNA="tRNA data"
gbtのstats
> d #またはsummary (d)
> d
*** Object of class gbt ***
*** Scaffolds ***
Total Scaffolds Max Min Median N50
1 107852810 92415 106400 500 884 1110
*** Marker counts by source ***
amphora2 blob
143 3072
*** SSU markers ***
[1] 3
*** tRNA markers ***
[1] NA
*** User-supplied variables ***
[1] ""
*** Function call history ***
gbt.default(covstats = c("SampleA2.covstats", "SampleG1.covstats"),
mark = c("amphora2_results.tab", "blobology_results.tab"),
marksource = c("amphora2", "blob"), ssu = "olavius_metagenome.ssu.tab")
plot (d, ssu=TRUE, textlabels=TRUE,legend=TRUE)
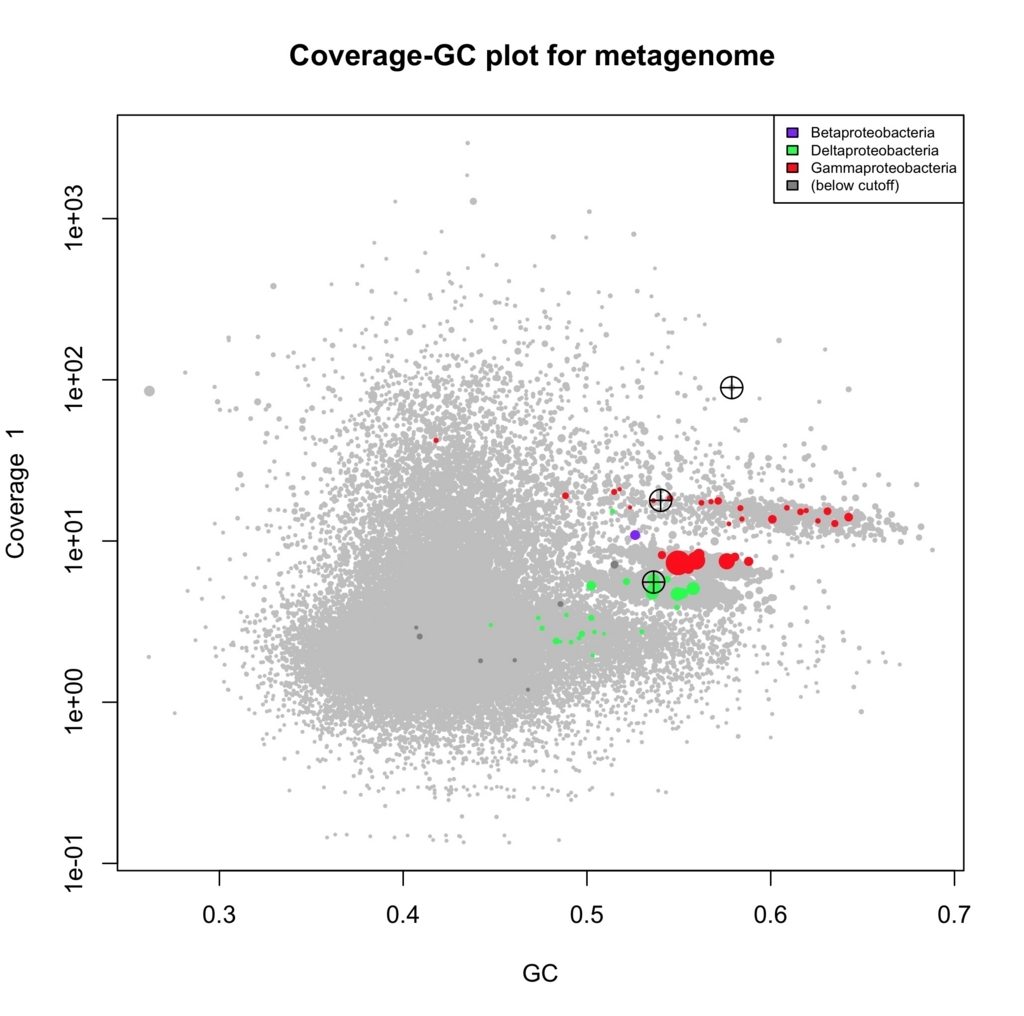
taxonがアサインされたcontigは色が付いている(step2出力情報を使用)。
両軸カバレッジ(異なるライブラリ)。
plot (d, slice=c(1,2))

任意のクラスターをbinning。
plot (d,slice=1,marker=FALSE) # Turn off color overlays
d.bin1 <- choosebin (d, slice=1) # Click on the plot to define the region you want
グラフを直接クリックしてbinningを行う。

囲んだ領域を検出する。
summary(d.bin1) # Summarize the newly-created bin
points(d.bin1, slice=1) # Overlay the new bin on your plot
検出された。

binningされたクラスターを保存する。
write.gbtbin(d.bin1,file="list.txt")
faSomeRecordsを使いFASTAに変換する。faSomeRecordsコマンドがなければkentutilsを導入する。
#kentutilsの導入。
git clone https://github.com/ENCODE-DCC/kentUtils.git
cd kentUtils/
./configure
make
cd bin/
#bin/にパスを通すなら(bash_profileの場合)
echo 'export PATH=$PATH:`pwd`' >> ~/.bash_profile && source ~/.bash_profile
listファイルからFASTAに変換。
faSomeRecords original_assembly.fasta list.txt list.fasta
lista.fastaが出力される。
BBtoolsを使い、binningされたcontigにマッピングされるリードを抽出する(outmにはペアリードの両方がアライメントされたものだけ出力される)。
bbmap.sh ref=shortlist.fasta outputunmapped=f outm1=mapped1.fq.gz outm2=mapped2.fq.gz in1=pair1.fq.gz in2=pair2.fq.gz
spades.py -k auto -t 20 --careful -1 mapped1.fq.gz -2 mapped2.fq.gz -o reassembly/
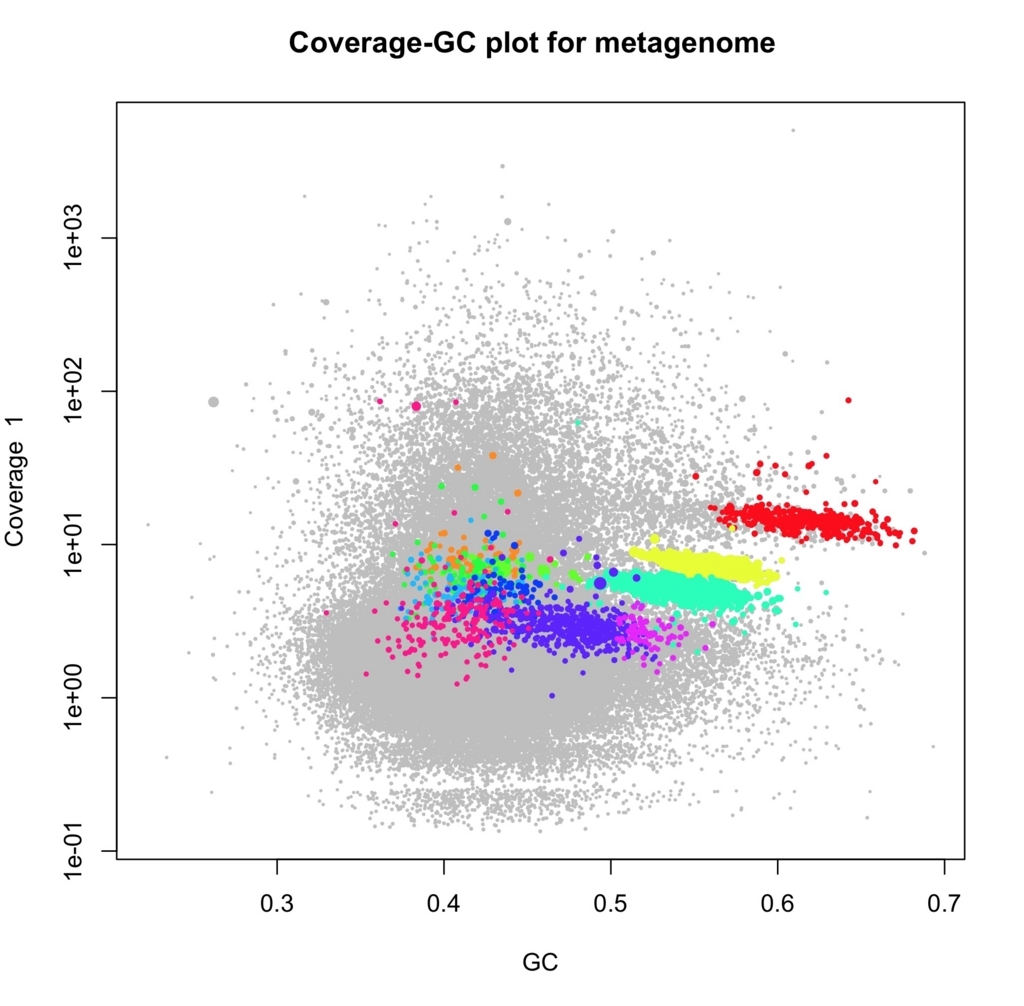
3-rd パーティのbinning結果を取り込むこともできる。ここではMetabatの結果を取り込む。
> head -n 5 Olavius_example/metabat_bins
$ head -n 5 Olavius_example/metabat_bins
bin09 contig-100_896
bin09 contig-100_3692
bin09 contig-100_3624
bin09 contig-100_3166
bin09 contig-100_3161
d.metabat_bins <- importBins (d, file="metabat_bins")
multiBinPlot (d, bins=d.metabat_bins)
検出された。
画像のパラメータ調整等についてはマニュアルを参照してください。
https://github.com/kbseah/genome-bin-tools/wiki
入力フォーマット
https://github.com/kbseah/genome-bin-tools/wiki/Appendix.-Input-file-formats
引用
gbtools: Interactive Visualization of Metagenome Bins in R.
Seah BK, Gruber-Vodicka HR
Front Microbiol. 2015 Dec 18;6:1451.
関連
