メタゲノム解析でコンティグを探索し、ビニングすることは、微生物群集の機能的、進化的なギルドを明らかにするために不可欠である。しかし、自動化されたビニングがあるにもかかわらず、研究者はしばしば、代表的な結果を得るために人間の関与が必要であると感じている。BinaRenaは、カスタマイズ可能な視覚化によってコンティグを探索し、配列指標、カバレッジプロファイル、分類学的割り当て、機能注釈など様々なデータタイプに基づいてビンと関連付けるために、人間の操作を支援する対話的グラフィックインターフェースである。ビニングプランは、視覚的またはアルゴリズムを用いて編集、検査、比較することができる。ユーザーが選択したコンティグの完全性と冗長性をリアルタイムに計算することができる。BinaRenaは、熱帯泥炭地のメタゲノムにおける生物学的パターンの発見、仮説の生成、ビンの絞り込みを容易にしたことを示す。また、ヒトの腸管サンプルから、密接に関連した集団内の病原性ゲノムを分離することができた。海洋のシミュレーションデータセットを用いたキュレーションにより、ビニングの質を大幅に向上させた。BinaRenaは、あらゆるレベルの研究者のための、インストール不要のクライアントエンドWebアプリケーションである。
Live demo
https://qiyunlab.github.io/binarena/demo.html
依存
- BinaRena runs in most modern web browsers, including Chrome, Firefox, Edge, and Safari (see screenshots below).
使い方
プログラムをダウンロードして解凍後、BinaRena.htmlをダブルクリックして立ち上げる。データセットを読み込むには、TSV形式のファイルを空白のプログラムウィンドウにドラッグ&ドロップする。

(レポジトリより)BinaRenaには依存がない。このプログラムは、ローカルでもリモートでも、Node.jsどころかサードパーティのライブラリも一切使っていない(つまり、<script>セクションにリモートのWebフレームワークやアイコンパックをsrcしていない)。プログラム全体が手作りで作られており、デプロイや利用のための労力を最小限に抑えている。
分析に必要なのは、タブ区切りのTSVファイル。個々のコンティグのプロパティを示したファイルを準備する。プロパティとして、数値、カテゴリー、特徴セット、記述の4つのデータ型を受け付ける。どのようなプロパティを提供するかはユーザーに委ねられている。

各行は1つのコンティグを表す。最初の列は一意のコンティグ識別子でなければならない。残りの列は任意のプロパティである。空のセルは欠損値として扱われる。
デモデータを見てみる(binarena-master/examples/input.tsv)。ファイルをウィンドウにドラッグ&ドロップするとどのプロパティをどのようなデータ型として読み込むのか選択できる。O.Kなら左上のimportをクリックする。

- numeric(n): 長さ、カバレッジ、GC%など。整数または浮動小数点数のいずれかを指定可能。
- categorical(c): 分類群、外部ビニング結果など。
- 特徴セット(f): 遺伝子など、各コンティグに関連する特徴量。dnaK,rpoB,ftsZ "のようにカンマで区切られた識別子で記述される。
- descriptive(d): コメントなどの任意のフリーテキスト。
-
BinaRenaは、コンティグ長(bp)とカバー率(x)の2つのプロパティを特別に扱う。これは、binアバンダンスを計算し、binのサマリーメトリックスを正規化するために使用するため。これらは、データインポートウィンドウの「Sp」欄で指定できる(「Len」「Cov」)。BinaRenaは、数値列の名前からこの2つの列を「推測」しようとするが、それでも必要であれば修正する必要がある。この2つの列のどちらか、あるいは両方がデータセットになくても問題ない。
BinaRenaは、データに基づいて各フィールドの最も適切なデータ型を「推測」する。しかし、やはり見直しをして、プログラムが十分に賢くないところは修正する必要があちる。同じデータセットを開くたびに手作業で見直すのを避けるには、生のTSVファイルの中に、「遺伝子|n」「プラスミド|c」のように、パイプ(|)の後にフィールド名を付けて型コードを付ける方法がある(マニュアルより)。
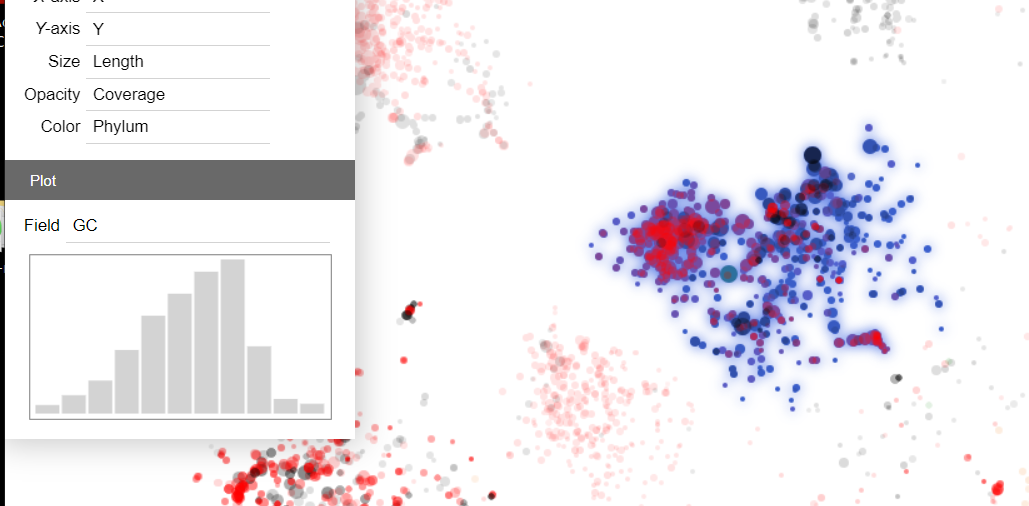
読み込まれた。表示項目は、X軸、Y軸、サイズ(コンティグの半径)、不透明度(アルファ値)、カラー。ドラッグや矢印キーで移動、マウスホイール(または = と -)で拡大・縮小が可能。

マーカーのサイズはコンティグ長の立方根スケールで(球体の体積は半径の3乗に比例するため)、カバレッジはマーカーの不透明度の平方根スケールで、highest taxonomic groupをマーカーの色で描画している。x軸および/またはy軸が見つからない場合、最初の2つの数値列がx軸および/またはy軸として使用される。
開いているデータセットにさらにデータを追加するには、さらにファイルをドラッグ&ドロップする。これらのファイルは、現在のコンティグID(最初の列)と一致するようにフィルタリングされ、各々のコンティグに情報が追加される。
複数のデータセットを同時に操作するには、複数のブラウザタブを開く。
プロットをクリックすると右側に情報が表示される(ユーザーが提供したプロパティの一覧)。Shift キーを押しながらクリックすると、複数のコンティグを選択できる。もう一度クリックすると、そのコンティグが非選択になる。

Search パネルでは、条件に一致するコンティグを検索してハイライト表示できる。

Plotパネルでは、選択されたコンティグについて、ユーザーが選択した変数のヒストグラムをインタラクティブに表示する。
コンティグの選択方法について説明する。
Enterキーを押すか左下の多角形マークをクリックすると、ポリゴン選択モードになる。コンティグを含む多角形をマウスでクリックしながら囲む。

もう一度Enterキーを押すと選択が完了する。Shiftキーを押しながら2回目のEnterキーを押すと、既存の選択範囲にコンティグを追加できる。

DeleteまたはBackspaceを押すと、選択したコンティグがマスクされる。Zキーを押すと元に戻る(50回まで戻れる)。この機能は、ビニングの際に不要なコンティグを隠すのに役立つ。

マスクされたコンティグは、その後の操作からも除外され、選択できなくなったり、メトリクスの計算に影響を与えたりする(データセットから削除されるわけではなく、マスクされているだけ)。
L を押すと選択した輪郭線がハイライトされる。

Fキーを押すと選択したコンティグ以外を隠す。

Spaceキーを押すと選択したコンティグから新しいビンが作成される(manual binning)。

ビンの名前をクリックすると、そのビンとメンバーであるすべてのコンティグが選択される。bin2(中央の一番下)。Shiftキーを押しながらクリックすると、複数のビンを選択できる。ビンの名前をもう一度クリックすると名前を編集できる。

binの左の格子状のボタンをクリックするとコンティグ一覧が表示される。

表示された。

binを選択し、さらにプロットをクリックして選択後>を押すと、選択したコンティグを現在のビンに追加し、<を押すと選択したコンティグを現在のビンから削除し、 /を押すと現在のビンを選択したコンティグで更新(つまり、その内容を置き換える)できる。0を押すと、プロットビューがリセットされる。
プロットのサイズは現在lengthとなっている。

lengfth の文字の右に表示されるアイコンから、表示されるデータの範囲を変更できる。

生物学的データは通常、大きく歪んでいる。それらを効果的に表示するためには、通常、適切な変換が必要になる。BinaRenaでは、様々な変換方法を用意している。変換するには、lengfth の文字の右に表示されるアイコンをクリックする。プロットのサイズをlogに変更。

注:特定の値は、特定の変換後に無効となる場合がある。例えば、0や負の数は対数変換できない。このような場合、コンティグはデフォルトの設定(例:色は灰色)で表示される(Githubより)。
カテゴリーデータの場合、BinaRenaはデータセット内で最も頻度の高いカテゴリーを自動的に識別して着色し、残りのすべてのカテゴリーを黒で残す。色付けするカテゴリの数は、フローティングの+ボタンと-ボタンで増減できる(Githubより)。

右上のビニングプランパネルでは、読み込んだプロパティから、どんなbinning結果になるか表示できる。
プランからMetaBATを選択。

MetaBATではこれだけのグループにビニングされることが分かる(ユーザーが用意した情報)。

B5をクリックすると、2つの領域がハイライトされていた。MetaBATの結果を参考にコンティグをマニュアル選択することでより上手くbinnig出来そうである。

binnigプランを修正(ビンの追加・削除、ビンへのコンティグの追加・削除、ビンの名前変更、新しいプラン名の入力など)した場合、Saveボタンが表示される。これをクリックすると、binnigプランがカテゴリカルフィールドとして保存される(Githubより)。
BinaRenaは、シルエット係数を計算することで、ビニングの信頼度を評価することができる。シルエット係数とは、あるコンティグが同じビンに含まれる他のコンティグとどの程度似ているかを、他のビンに含まれるコンティグと対比して測定するものである。その範囲は-1(最悪)から1(最高)である。Evaluateをクリック。

"シルエット係数 "ウィンドウが表示される。計算に含める変数を選択する。

シルエット係数がMetaBATのbinについて計算された。

「export result to file」にチェックを入れると、結果(コンティグID、bin assignments、シルエット係数)をTSVファイルに保存することができる。
シルエット係数は、クラスター解析の評価指標として広く用いられている。しかし、ビニングの作業においては、他の生物学的要因も重要である場合があり、過大評価しないように注意する(Githubより)。
BinaRenaで描画したプロットは、画像ファイルとして書き出せる。右クリックして選ぶか下のメニューの✿ボタンをクリックする。

引用
BinaRena: a dedicated interactive platform for human-guided exploration and binning of metagenomes
Michael J. Pavia, Abhinav Chede, Zijun Wu, Hinsby Cadillo-Quiroz, Qiyun Zhu
bioRxiv, Posted July 02, 2022
関連