2019 4/22 誤字修正
2019 5/6 リンク追記
2019 6/23 ランのstep1,2 の説明修正
2019 9/25 step1のフラグの誤り修正
微生物は、人間を含む地球上のほとんどすべての生物に繋がることが知られており、そこでは微生物は健康、病気および農業に多大な影響を与えると考えられている(ref.1-3)。しかしながら、ほんのわずかな割合の環境微生物しか実験室で容易に培養できないことが長い間知られてきた(ref.4)。したがって、微生物系統樹の大部分は、培養に依存しないシークエンシング(「メタゲノミクス」)を介してのみまだアクセス可能である。初期のメタゲノム解析では、異なる環境内の個々のバクテリア種の相対的存在量を調べることによって群集の系統発生プロファイルに焦点を当てていたが(16S rRNA遺伝子シークエンシングを通じて定量)、これらの環境を形成する機能的寄与および生物レベルの相互作用に関する情報は限られていた(ref.5) 。全ゲノム「ショットガン」シーケンシングは、標準的なプライマーでは検出できない非標準のリボソームRNA遺伝子に関する問題や固有の低分解能など、ハイスループット16S rRNAアンプリコンシーケンシングが直面するいくつかの課題を克服することができる。しかし、メタゲノムコンティグを個々のゲノムを表すクラスターに分類すること(「ビニング」)は、困難な計算問題であり、現在も活発な研究分野である(ref.7,8)。ビニングは、コミュニティ全体の代謝能力に対する個々の微生物の代謝、機能的寄与を理解するための必要なステップである。言い換えれば、メタゲノムのゲノムレベルの解明により、研究者は、代謝機能の解釈を超えて、その場で集合的に複雑な系内の個々の生物の役割を理解することが可能になる。
ほとんどの環境が主に特徴付けられていない微生物で構成されていることを考え、リファレンスフリーのビニングを達成するためにさまざまなアプローチが取られてきた。例えば、ヌクレオチド組成は、emergent self-organizing maps(ESOM)(ref.9)またはBarnes-Hut stochastic Neighbor Embedding(BH-tSNE)(ref.10、11)でコンティグをグループ化するために使用されてきた。これらのアプローチは、k-merスペクトラムの変動を2次元に減らし、高次元データの視覚化を可能にし、そしてヒト主導のクラスタリングを可能にする。他の取り組みでは、共有ゲノムのコンティグのカバレッジは明確な共分散を示すという仮定の下、複数のサンプルからの情報を活用することに焦点が当てられている。手動(ref.12、13)と自動パイプライン(ref.14、15)の両方がこのアプローチを使用している。ただし、この方法にはいくつかの欠点がある。多くのマルチサンプルプロトコルでは、すべてのサンプルからのリードのアセンブリが必要である。共有ゲノムがクローンでない場合、計算要件が高まり、潜在的にアセンブリ品質が低下する。この問題は「microdiversity」として知られている - Albertsen et alによって認められた問題であり(ref.13 link(有名な論文))、そして最近他でも実証された(ref.16 link)(紹介)。同時アセンブリのためにサンプルをプールすることによって、ユーザーはco-assemblyの効果を悪化させることもあり得る。Co-assembly結果は、1つのサンプルから採取された単一系統、生物または集団のゲノムの代わりに、広く集約されたコンセンサス配列を表している(ref.8,16,17) 。そのようなaggregationは、個々の株またはサンプルにのみ見られるpan-genome配列(ref.18)の存在をマスクでき、それは抗生物質耐性を付与する可動要素(ref.19,20)または水平伝播を通して得られる生合成遺伝子クラスターを考慮するとき重要な臨床的およびバイオテクノロジー的意味を有する 。システムの根本的な変動性または重複が未知であるさらなる状況があり、少数のパイロットデータセットから情報を抽出したいという要望がある。さらに、本質的により高いシークエンシングコストを招くマルチサンプル比較は、1つのサンプルに固有のゲノムのビニングに必ずしも役立つわけではない(ref.21)。
今日まで、bioactive small moleculeを作る海洋無脊椎共生生物のゲノムをシーケンシングするための我々の努力は半手動のビニング技術に頼っていた(ref.21、22)。しかし、海綿動物のマイクロバイオームは豊富なバイオテクノロジーの可能性を提供し(ref.23)、何百もの微生物種を含むことができ、海綿動物の組織体積の最大40%を占める(ref.24、25)。他のシステムも挑戦的である。例えば、カブトムシLagria villosaの卵はBurkholderia gladioliのいくつかclosely relatedな株の混合物と関連していることを発見したが、これらのうちのいくつかだけが培養可能で卵を感染から守る抗真菌化合物を生産する。これらのシステムは合理的な手動処理の限界を超え、そしてそのような宿主関連メタゲノムのための既存の自動ビニングパイプラインの不十分な性能が、本著者らの自動化されたスケーラブルなビニングアルゴリズム開発の動機となった。本手法Autometaは、個々のメタゲノムアセンブリからのメタゲノムの複雑さに従ってスケーリングを最大化するために、コンティグの単純化されたサブセット(分類学的にバクテリアまたはアーキアのいずれかに分類されるもの)でクラスタリングを実行する。初期クラスタは、教師付き機械学習アルゴリズムによるその後の分類のためのトレーニングセットとして機能する。我々(著者ら)は、既知の構成要素ゲノムを参照して性能を評価できるシミュレーションの合成メタゲノムを使い、ならびに以前にセミマニュアルビニングによって調べた実際の宿主関連メタゲノムを使用し、Autometaを評価した(ref.21、22)。特にメタゲノムの複雑さが高い場合やホスト関連のデータセットにおいて、AutometaがMaxBin(ref.14)、MetaBAT(ref.27)、MyCC(ref.28)、BusyBee Web(ref.29)と同程度または性能を上回っていることがわかった。さらに、lowest common ancestor(LCA)分析を使用したコンティグレベルの分類学上の分類により、Autometaのパフォーマンスや他のパイプラインのビニングパフォーマンスを向上させることができた。
Autometaは、コンティグを区別するために配列相同性、カバレッジおよびヌクレオチド組成を使用して、シングルショットガンメタゲノムから新規にゲノムを回収する。このタスクは、以前にバクテリアおよびアーキアにおいて同定されているシングルコピー存在することが知られているマーカー遺伝子の存在によってガイドされる。各マーカーはビンごとに1回しか検出されないため、マーカー遺伝子の存在は、ビンのゲノム完全性および汚染レベルを推定するために使用することができる。シングルコピーマーカーは、以前はMaxBin(ref.14)とMyCC(ref.28)で使用されていたが、ここでは異なるアプローチを取る。 MaxBinでは、期待値最大化(EM)アルゴリズムのためシングルコピーマーカーを使用してクラスター数、平均テトラヌクレオチド頻度、およびカバレッジを初期化する。ビン内の2以上のマーカーの中央値は、EMが収束したかどうかの大まかな尺度として使用される。 MyCCは、どのクラスターをマージまたは分割すべきかを決定するために、 affinity propagationによる1ラウンドのクラスター化の後にシングルコピーマーカーを利用する(ref.28)。これら両方のパイプラインは、真のゲノムが予想される数のマーカーを有するという仮定に悩まされており、そしてMyCCの場合には、この情報はクラスター化段階を導くために使用されない。これとは対照的に、Autometaはシングルコピーマーカーを使用してクラスタリングをガイドし、回復可能なゲノムが必ずしも「完全」であるとは想定していない。環境メタゲノムに見られる微生物は、以前にシーケンシングされたすべての生物とは非常に異なっている可能性があり、真核生物宿主に関連する微生物はしばしば独立した生活に不可欠な機能を失うようなゲノムの分解および減少の過程を経ることが知られている(ref.31,32)。例えば、本著者らは最近、NCBI NRデータベースでヒットした遺伝子はわずか20%で、予想されるバクテリアのシングルコピーマーカーのわずか20%しか検出できないという、既知の配列とは異なるゲノムが減少したバクテリアを同定した 。したがって、MaxBin(ref.14)のように、ビンが100%完全に近いことを想定したり、ビンの数を事前計算するためにシングルコピーマーカーを使用したりすることは想定していない。 Autometaで採用されているプロセス全体は、大きく3つの段階から構成されている(論文図1)。
コンティグをKingdomビンに広範に分離することにより、宿主由来または他の真核生物汚染を除去することが可能になり(たとえ宿主ゲノムがリファレンスデータベースに表されていなくても)、バクテリアおよびアーキア由来コンティグの分離が可能になる。遺伝子は、Prodigal(ref.33)を使用して指定された長さのカットオフよりも長いすべてのコンティグで識別される(デフォルトは10,000 bpだが、ここでテストされたすべてのデータセットは3,000 bpカットオフに基づいている)。次いで、翻訳されたコード配列は、DIAMOND(ref.34)を用いてNCBI NRデータベースに対してクエリされる。トップヒットの10%以内のビットスコアを有するヒットのLCAを用いて、NCBI taxonomyデータベース(https://www.ncbi.nlm.nih.gov/taxonomy)に従って各予測タンパク質にtaxonomy IDを割り当てる。遺伝子水平伝播の影響を減らすために、コンティグレベルの分類は、構成要素予測コード配列の修正多数決によって割り当てられる。分類は特異性の低い順に(種、次に属、科、目、綱、門および界)考慮され、タンパク質の大多数がより低い特異性で分類されてしまい答えが得られない場合、コンティグ内のすべてのタンパク質の最小公約数がコンティグ分類として使用される。
(以下略)

Autometa metagenome workflow. 論文より転載
Our automated metagenomic binning pipeline - Autometa - is now published in @NAR_Open! https://t.co/KbCVOxyigp
— Jason C Kwan (@kwan_lab) March 6, 2019
インストール
オーサーらが準備したdockerイメージを使いテストした。
依存
Third party programs
Databases (these will be automatically downloaded the first time you run make_taxonomy_table.py). NOTE: You will need ~150 GB (at time of writing) to download and process these.
- nr (Note: tested with newer NR versions without GI numbers)
- taxdump.tar.gz
- prot.accession2taxid.gz
Python packages (Note: this list assumes the use of Anaconda Python)
Additionally, if you want to calculate your own contig coverages (rather than trusting the coverage values given by the SPAdes assembler), you will need:
本体 Github
docker image
docker pull jasonkwan/autometa
docker run -itv $PWD:/data/ jasonkwan/autometa
> cd /autometa/pipeline/
> run_autometa.py -h
# run_autometa.py -h
usage: run_autometa.py [-h] -a <assembly.fasta> [-p <int>] [-l <int>]
[-c <float>] [-k <archaea|bacteria>]
[-t <taxonomy.tab>] [-o <dir>] [-r] [-m] [-db <dir>]
[-v <coverage.tab>]
Script to run the Autometa pipeline.
optional arguments:
-h, --help show this help message and exit
-a <assembly.fasta>, --assembly <assembly.fasta>
Path to metagenomic assembly fasta (default: None)
-p <int>, --processors <int>
Number of processors to use (default: 1)
-l <int>, --length_cutoff <int>
Contig length cutoff to consider for binning in bp
(default: 10000)
-c <float>, --completeness_cutoff <float>
Completeness cutoff (in percent) to use for accepting
clusters (default: 20.0)
-k <archaea|bacteria>, --kingdom <archaea|bacteria>
Kingdom to consider (default: bacteria)
-t <taxonomy.tab>, --taxonomy_table <taxonomy.tab>
Path to output of make_taxonomy_table.py (default:
None)
-o <dir>, --output_dir <dir>
Path to directory to store all output files (default:
.)
-r, --ML_recruitment Use ML to further recruit unclassified contigs
(default: False)
-m, --maketaxtable runs make_taxonomy_table.py before performing autometa
binning. Must specify databases directory (-db)
(default: False)
-db <dir>, --db_dir <dir>
Path to directory with taxdump files. If this doesn't
exist, the files will be automatically downloaded
(default: /autometa/databases)
-v <coverage.tab>, --cov_table <coverage.tab>
Path to coverage table made by
calculate_read_coverage.py. If this is not specified
then coverage information will be extracted from
contig names (SPAdes format) (default: None)
Please do not forget to cite us. Thank you for using Autometa!
> calculate_read_coverage.py -h
# calculate_read_coverage.py -h
usage: calculate_read_coverage.py [-h] -a <assembly.fasta> [-p <int>]
[-F [<reads.fastq|reads.fastq.gz> [<reads.fastq|reads.fastq.gz> ...]]]
[-R [<reads.fastq|reads.fastq.gz> [<reads.fastq|reads.fastq.gz> ...]]]
[-S [<reads.fastq|reads.fastq.gz> [<reads.fastq|reads.fastq.gz> ...]]]
[-o <dir>]
Script to tabulate paired-end or single read coverage using Samtools and
Bedtools. Note: it is recommended that you quality filter your *.fastq or
*fastq.gz files prior to calculating read coverage.
optional arguments:
-h, --help show this help message and exit
-a <assembly.fasta>, --assembly <assembly.fasta>
Path to input assembly file
-p <int>, --processors <int>
Number of processors
-F [<reads.fastq|reads.fastq.gz> [<reads.fastq|reads.fastq.gz> ...]], --forward_reads [<reads.fastq|reads.fastq.gz> [<reads.fastq|reads.fastq.gz> ...]]
Paired (*.fastq|*.fastq.gz) forward reads (must be in
same order as reverse list)
-R [<reads.fastq|reads.fastq.gz> [<reads.fastq|reads.fastq.gz> ...]], --reverse_reads [<reads.fastq|reads.fastq.gz> [<reads.fastq|reads.fastq.gz> ...]]
Paired (*.fastq|*.fastq.gz) reverse reads (must be in
same order as forward list)
-S [<reads.fastq|reads.fastq.gz> [<reads.fastq|reads.fastq.gz> ...]], --single_reads [<reads.fastq|reads.fastq.gz> [<reads.fastq|reads.fastq.gz> ...]]
Single (*.fastq|*.fastq.gz) reads
-o <dir>, --output_dir <dir>
Path to output directory
> make_taxonomy_table.py -h
# make_taxonomy_table.py -h
usage: make_taxonomy_table.py [-h] -a <assembly.fasta> [-p <int>] [-db <dir>]
[-udb <user_prot_db>] [-l <int>]
[-v <coverage.tab>] [-o <dir>] [-bgc <dir>] [-s]
[-u]
Script to generate the contig taxonomy table.
optional arguments:
-h, --help show this help message and exit
-a <assembly.fasta>, --assembly <assembly.fasta>
Path to metagenomic assembly fasta
-p <int>, --processors <int>
Number of processors to use.
-db <dir>, --db_dir <dir>
Path to directory with taxdump, protein accessions and
diamond (NR) protein files. If this path does not
exist, will create and download files.
-udb <user_prot_db>, --user_prot_db <user_prot_db>
Replaces the default diamond database (nr.dmnd)
-l <int>, --length_cutoff <int>
Contig length cutoff to consider for binning in bp
-v <coverage.tab>, --cov_table <coverage.tab>
Path to coverage table made by
calculate_read_coverage.py. If this is not specified
then coverage information will be extracted from
contig names (SPAdes format)
-o <dir>, --output_dir <dir>
Path to directory to store output files
-bgc <dir>, --bgcs_dir <dir>
Path to directory of biosynthetic gene clusters. Masks
BGCs
-s, --single_genome Specifies single genome mode
-u, --update Checks/Adds/Updates: nodes.dmp, names.dmp,
accession2taxid, nr.dmnd files within specified
directory.
Output will be directed to recursive_dbscan_output.tab
> ML_recruitment.py -h
# ML_recruitment.py -h
usage: ML_recruitment.py [-h] -t <contig.tab> [-c <column header>] [-p <int>]
[-r] [-C <int>] [-u <unclustered name>] [-n <int>]
[-m <k-mer.tab>] -o <output.tab>
[-k <archaea|bacteria>]
Recruit unclustered (or non-marker) sequences with Machine Learning
classification using clustered sequence as training data. Features to train
with include sequence coverage, composition, and homology. Confidence is
calculated using jackknife cross-validation by randomly subsetting the
training data n number of times.
optional arguments:
-h, --help show this help message and exit
-t <contig.tab>, --contig_tab <contig.tab>
Path to master contig table which includes initial
clusters
-c <column header>, --cluster_column <column header>
Name of column containing initial cluster information
-p <int>, --processors <int>
Number of processors to use
-r, --recursive If specified, will run classification iteratively and
refine traning data after each iteration.
-C <int>, --Confidence_cutoff <int>
Confidence cutoff value to use to keep ML-based
predictions.
-u <unclustered name>, --unclustered_name <unclustered name>
Name of unclustered group in cluster column
-n <int>, --num_iterations <int>
Number of iterations for jackknife cross-validation.
-m <k-mer.tab>, --k_mer_matrix <k-mer.tab>
Path to k-mer_matrix file.
-o <output.tab>, --out_table <output.tab>
Path to create output table with new column for ML-
recruited sequences.
-k <archaea|bacteria>, --kingdom <archaea|bacteria>
Kingdom to consider (archaea|bacteria)
> cluster_process.py -h
# cluster_process.py -h
usage: cluster_process.py [-h] -b <bin.tab> [-c <bin column name>] -f
<assembly.fasta> [-o <dir>] [-k <archaea|bacteria>]
[-t] [-db <dir>]
Script to summarize the assembly characteristics and taxonomy of binned
clusters, as well producing individual cluster fasta files
optional arguments:
-h, --help show this help message and exit
-b <bin.tab>, --bin_table <bin.tab>
path to the output from either run_autometa.py or
ML_recruitment.py
-c <bin column name>, --column <bin column name>
the name of the column to use for binning purposes
-f <assembly.fasta>, --fasta <assembly.fasta>
path to the assembly used to make the bin table
-o <dir>, --output_dir <dir>
path to the directory where output files will go
-k <archaea|bacteria>, --kingdom <archaea|bacteria>
kingdom to consider
-t, --do_taxonomy carry out taxonomic analysis on the clusters (you must
have already run make_taxonomy_table.py)
-db <dir>, --db_dir <dir>
Path to directory with taxdump files
実行方法
Step0 、de novo assembly
メタゲノムのアセンブラはmetaspadesが推奨されている。ラン後にできるscaffolds.fastaを使う。
この先はstep by stepで進めるか、または全ステップをワンライナーで自動実行する。
step by stepの流れ
optional step
Autometaは、metaspadesなどのアセンブルのmulti-fastaのヘッダにあるcoverage情報を使う。"metaspades"以外のメタゲノムアセンブラでアセンブルを行なった場合、まずこのマンドでcoverage情報を取得する。
calculate_read_coverage.py --assembly scaffolds.fasta -p 24 \
-F reads_R1.fastq.gz -R reads_R2.fastq.gz
- -F Paired (*.fastq|*.fastq.gz) forward reads (must be in same order as reverse list)
- -R Paired (*.fastq|*.fastq.gz) reverse reads (must be in same order as forward list)
- -S Single (*.fastq|*.fastq.gz) reads
coverage.tabが出力される。step1、2で使用する。
Step 1: Split data into kingdom bins [optional]
アセンブル配列のkingdomレベルでのbiinningを行い真核生物のコンタミネーションを除く(例えばホストと相互作用する系から取ったメタゲノムなど)。真核生物のコンタミネーションがないサンプルなら、このステップはスキップしても支障ない。このジョブにはデータベースとしてnrを使う。
実行する。初回ランでデータベースがない場合はまずダウンロードされる(/autometa/databases/)。150GBほどディスクスペースを使うので注意する(*1)。step1はDiamondなどを使ったリソース集約的なジョブなので時間がかかる。CPUも利用できるだけ指定する。optional stepでcoverage情報ファイルを計算している場合、"-v coverage.tab"で指定する。
#metaspades scaffolds
make_taxonomy_table.py -a autometa/test_data/scaffolds.fasta \
-p 24 -l 3000
#other assembler (step0出力の "--cov_table coverage.tabをつける")
make_taxonomy_table.py -a autometa/test_data/scaffolds.fasta \
-p 24 -l 3000 --cov_table coverage.tab
- -a Path to metagenomic assembly fasta (default: None)
- -l Contig length cutoff to consider for binning in bp
- -v Path to coverage table made by calculate_read_coverage.py. If this is not specified then coverage information will be extracted from contig names (SPAdes format)
- -p Number of processors to use (default: 1)
- -o Path to directory to store output files (default: .)
ジョブが終わるとBacteria.fastaとEukaryotes.fasta、Viruses.fasta、その他の関連ファイルが出力される。taxonomy.tabというcontigにtaxonomyがアサインされたファイルも出力される。
taxonomy.tabの中身

Step 2: Bin bacterial contigs with BH-tSNE and DBSCAN
binningを行うAutometaのコアジョブになる。先ほど作成したbacteria.fastaを使う。このコマンドはbacteriaのkingdomのみで正常動作する。これはautomataにはbacteriaのsingle-copy geneデータベースしか入っていないためで、他のkingdomのfastaファイルではうまく動作しないとされる。binning精度を上げるためstep1で作ったtaxonomy.tabも必要になる。"-v coverage.tab"で指定する。
#metaspades scaffolds
run_autometa.py -a Bacteria.fasta -p 24 -l 3000 -t taxonomy.tab -c 20
##other assembler
run_autometa.py -a Bacteria.fasta -p 24 -l 3000 -t taxonomy.tab -c 20 --cov_table coverage.tab
- -a Path to metagenomic assembly fasta (default: None)
- -p Number of processors to use (default: 1)
- -l Contig length cutoff to consider for binning in bp (default: 10000)
- -c Completeness cutoff (in percent) to use for accepting clusters (default: 20.0)
- -k <archaea|bacteria> Kingdom to consider (default: bacteria)
- -t Path to output of make_taxonomy_table.py (default: None)
- -v Path to coverage table made by calculate_read_coverage.py. If this is not specified then coverage information will be extracted from contig names (SPAdes format) (default: None).
- -o Path to directory to store all output files (default: .)
各contigに見つかったmarker geneをまとめたファイルBacteria_filtered_marker.tab、binning結果をまとめたtab仕分けファイルrecursive_dbscan_output.tab、各contigの5-mer頻度をまとめたk-mer_matrixなどが出力される。
Step 3: Recruit unclustered contigs to bins through supervised machine learning [optional]
step2のbinning結果をデータベースにsupervisedのmachine learningを行い、クラスタリングされなかったcontigの再binningを試みる。step2で作成したrecursive_dbscan_output.tabとk-mer_matrixを指定する。
ML_recruitment.py --contig_tab recursive_dbscan_output.tab --recursive \
--k_mer_matrix k-mer_matrix -o ML_recruitment_output.tab
- -t Path to master contig table which includes initial clusters
- --k_mer_matrix Path to k-mer_matrix file.
- -o Path to create output table with new column for ML-recruited sequences.
- --recursive If specified, will run classification iteratively and refine traning data after each iteration.
ML_recruitment_output.tabが出力される。
Running all steps in sequence
全ステップの自動実行。--ML_recruitment をつけるとstep3のML_recruitment.pyも含めて実行される。
run_autometa.py -a /autometa/test_data/scaffolds.fasta \
-p 16 -l 3000 -m --ML_recruitment
- -m runs make_taxonomy_table.py before performing autometa binning. Must specify databases directory (-db) (default: False)
- --ML_recruitment Use ML to further recruit unclassified contigs (default: False)
Analysis of results
最後に分離したfastaやtaxnomy情報を記載したファイルを出力する。step3のML_recruitment_output.tab、step1のBacteria.fasta、データベースのパスを指定する。
cluster_process.py --bin_table ML_recruitment_output.tab \
-c ML_expanded_clustering --fasta Bacteria.fasta \
--do_taxonomy -db /autometa/databases -o cluster_process_output
- -c the name of the column to use for binning purposes
- -db Path to directory with taxdump files. If this doesn't exist, the files will be automatically downloaded (default: /autometa/databases)
- --do_taxonomy carry out taxonomic analysis on the clusters (you must have already run make_taxonomy_table.py)
上記コマンドの実行でcluster_process_output/ができ、その中にbinning(クラスタリング)したfasta、summary table等が出力される。
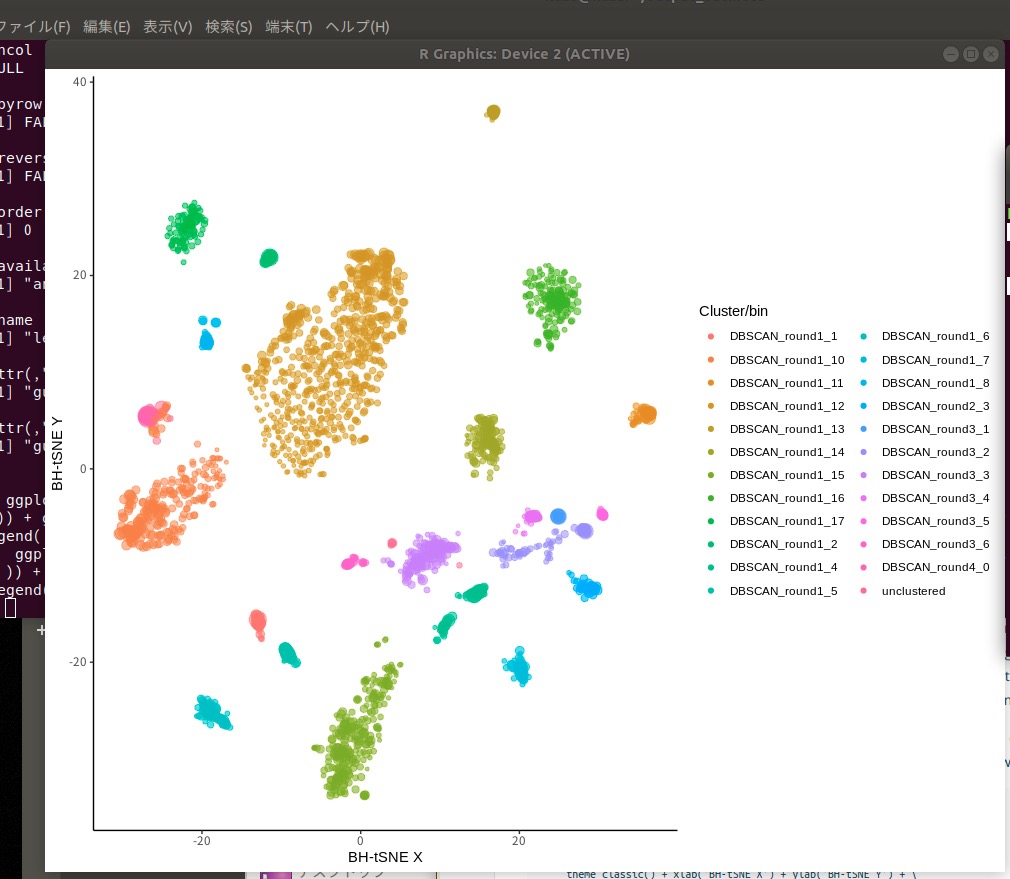
結果の視覚化 - ggplot2を使う。
library(ggplot2)
data = read.table('ML_recruitment_output.tab', header=TRUE, sep='\t')
ggplot( data, aes( x = bh_tsne_x, y = bh_tsne_y,
col = ML_expanded_clustering )) +
geom_point( aes( alpha = 0.5, size = sqrt(data$length ) / 100 )) +
guides( color = 'legend', size = 'none', alpha = 'none' ) +
theme_classic() + xlab('BH-tSNE X') +
ylab('BH-tSNE Y') + guides( color =
guide_legend( title = 'Cluster/bin' ))
引用
Autometa: automated extraction of microbial genomes from individual shotgun metagenomes
Ian J Miller Evan R Rees Jennifer Ross Izaak Miller Jared Baxa Juan Lopera Robert L Kerby Federico E Rey Jason C Kwan
Nucleic Acids Research, Published: 06 March 2019
関連ツール
*1
ダウンロードしてデータベースビルドするのに一晩かかった。