Gephiは大規模なネットワークグラフを可視化・分析するためのオープンソースソフトウェアである。Gephiは3Dレンダリングエンジンを使ってグラフをリアルタイムに表示し、探索を高速化する。あらゆる種類のグラフの探索、分析、空間化、フィルタリング、クルータライズ、操作、エクスポートが可能になっている。
https://ppanggolin.readthedocs.io/en/latest/user/PangenomeAnalyses/pangenomeGraphOut.html
ウェブ版gephi-liteも用意されている(小さなpangenomeグラフのみ扱える)
インストール
Download
Windows、Mac OS X(intel and apple silicon)、Linux版がある。
実行手順
ここではPPanGGOLiNで計算したパンゲノムグラフを使用します。
起動した。

ファイル=> 開くでpangenomeGraph.gexfを指定する(*1)。

ノード数12323、エッジ18007

開いた。中央のパネルがグラフビューアとなる。マウスホイールで拡大縮小、右クリックしてのドラッグでスクロールとなる。

PPanGGOLiNでは、レイアウトにForce Atlas 2 アルゴリズム(pubmed)を選択し、"Stronger Gravity: on "と "scaling: 4000 "でレイアウトを計算することを推奨している。レイアウトパネルでForce Atlas2を選択する。

Stronger Gravity: on、scaling: 4,000

実行をクリックするとForce Atlas 2 のレイアウトがリアルタイムで適用されていく。

ここからは個別の機能を見ていく。
短形選択で特定のノードだけ選択できる。ドラッグを繰り返すと追加で選択できる。
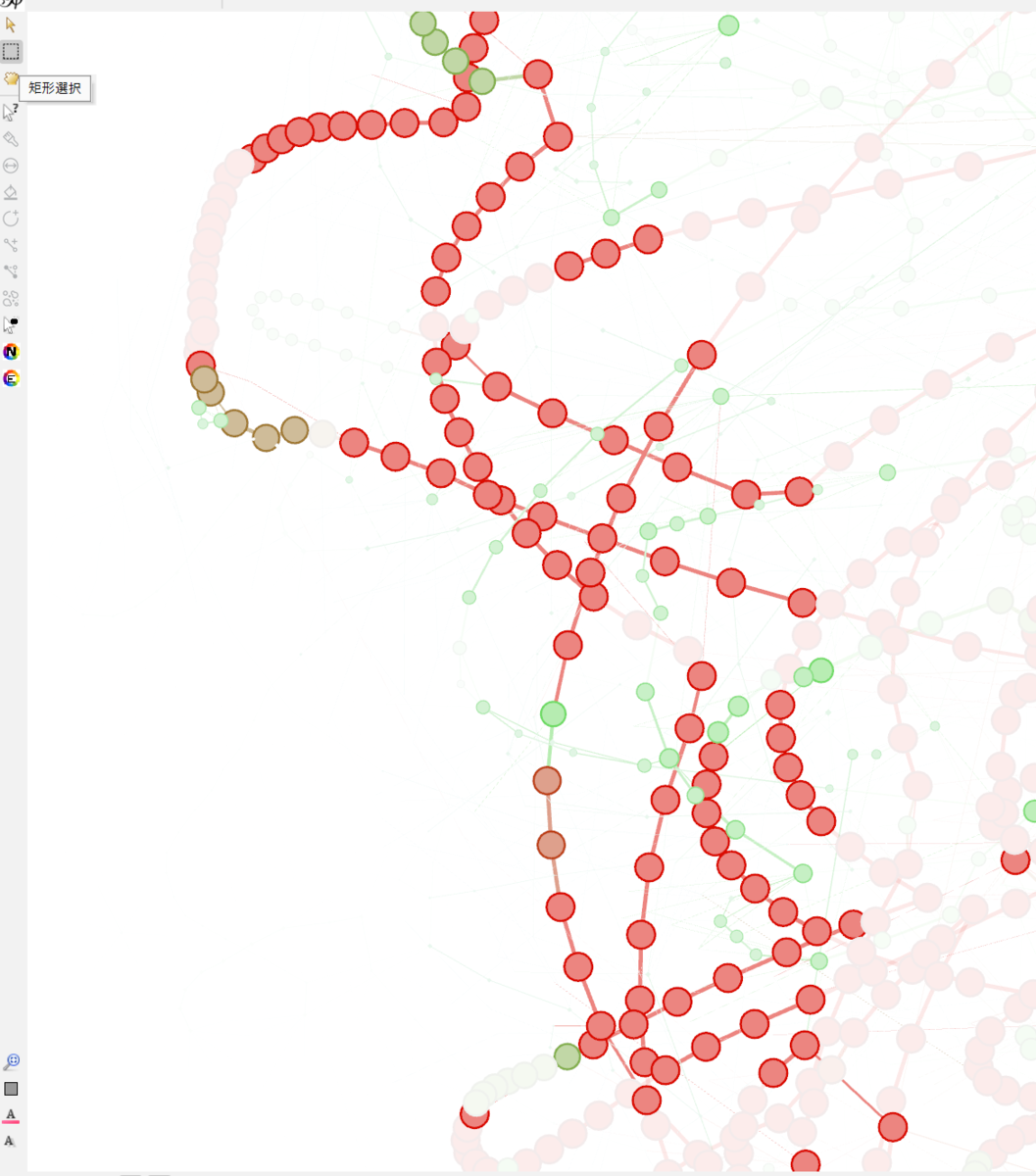
右クリックして新規ワークスペースにコピーする。

新規ワークスペースのタブに移動後、グラフを中心ボタンを押すとグラフの位置にスクロールする。

表示された。カラーリセットなどのボタンも用意されている。

exportでグラフフォーマットかSVG, PDF, PNGとして書き出せる。

選択できるグラフフォーマット

Appearanceパネルからは、ノードやエッジの外観をカスタマイズできる。
AppearanceのNodesを選択、

Uniqueタブ - 特定の色が適用される。
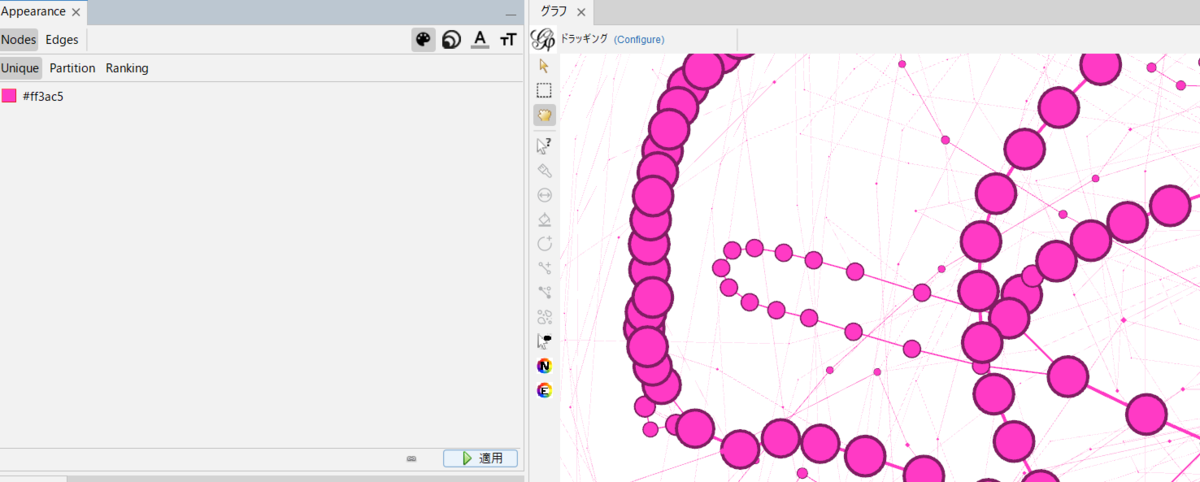
Partitionタブ- 例えば遺伝子ごとに色をアサインできるが、パンゲノムのノード全てを視覚化する時は適していないと思われる。

Partitionタブの下の方には、cloud、persistent、shellに事前分類されたpartitionも選べる。

PartitionのPartition_soft適用;soft core & soft accesoryの2色

Partitionのnb_genes:遺伝子コピー数も見たいときなど

Rankingタブ - ノード(遺伝子)のサイズ、共有されている遺伝子数の度合いでグラデーションカラーを適用する。

No of genomeで3色のグラデーションを適用した。全部のゲノムに共有されているノード(遺伝子)は赤とした。

(反映させるには右下の適用をクリックする)
同様にエッジの色も指定できる。

ノードの大きさを変えるには、右上の4つ並んだボタンの左から2つ目;”大きさ”ボタンを選ぶ。

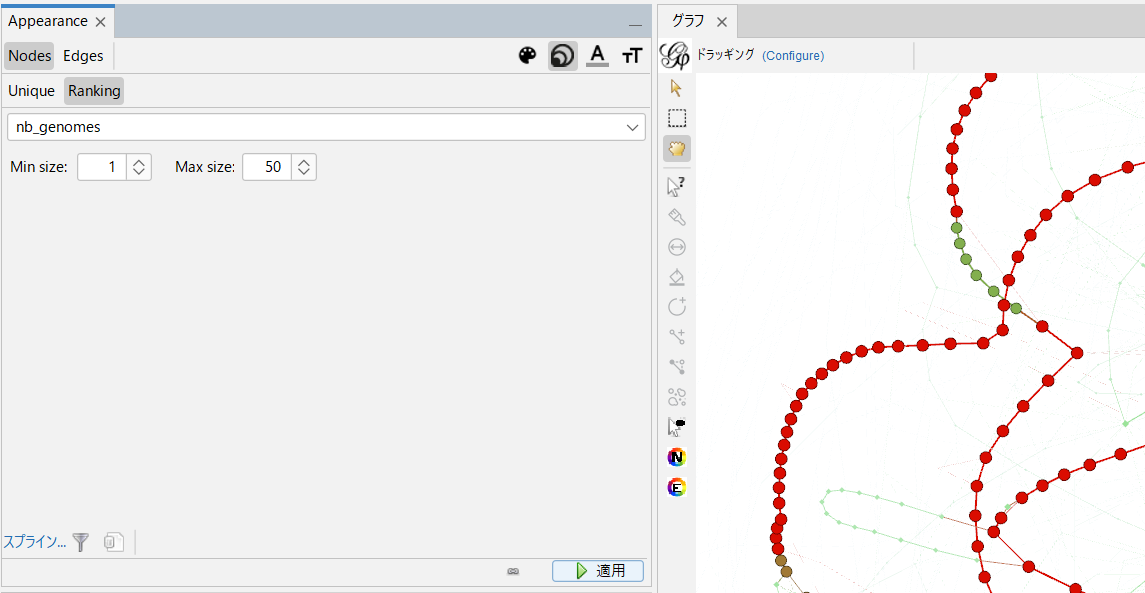
NodesのRankingタブを選択、そしてnb_genomesを選択した。

Min: 1、Max: 100とした。

Min: 1、Max: 200

Min: 1、Max: 50
ノードラベルを表示させるにはビューアパネルの下のTマークをクリックする。
(エッジのラベルを表示させるには黒いTボタン右の白いTボタンをクリックする)
グラフパネルの左下の電球マークをクリックすると背景が黒になる。

フォントの種類とサイズは中央下のボタンから指定できる。視認性に優れたMyriad Proを選択、

ラベルもAppearanceから細かく調整できる。label Colorを選択、

No_genomesでグラデーションカラーを適用した。コア遺伝子以外は色が薄くなった。
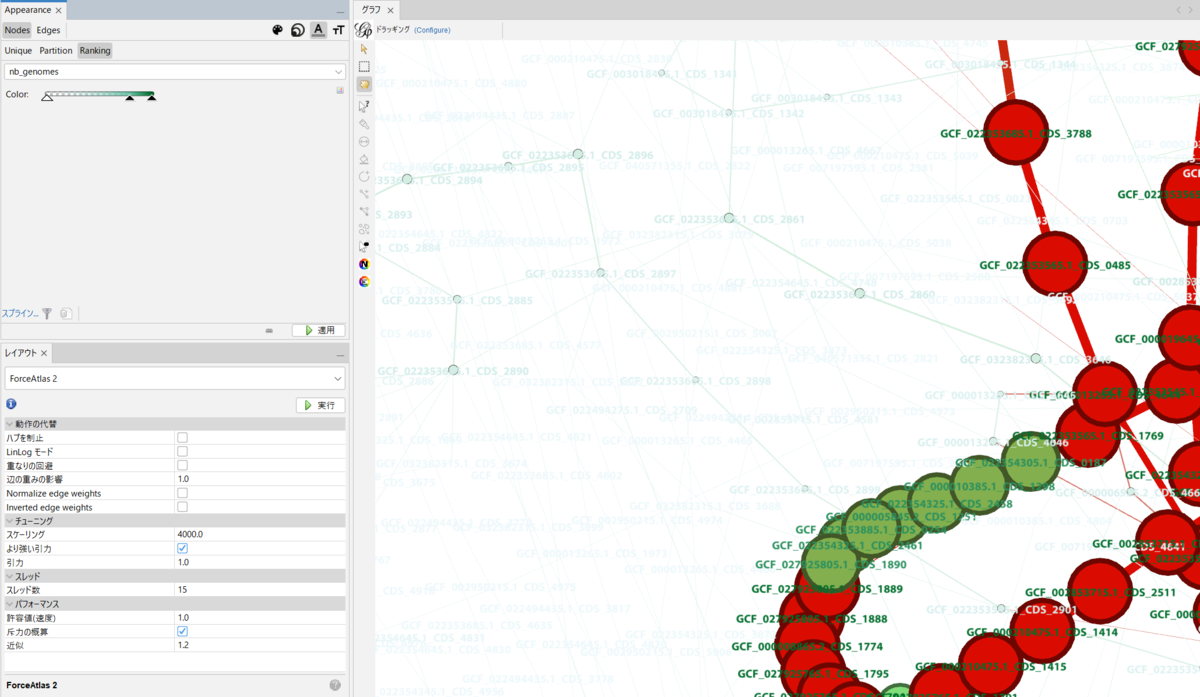
Label sizeではラベルの大きさを調整できる。

MIn: 0.1、Max: 1.5とした。
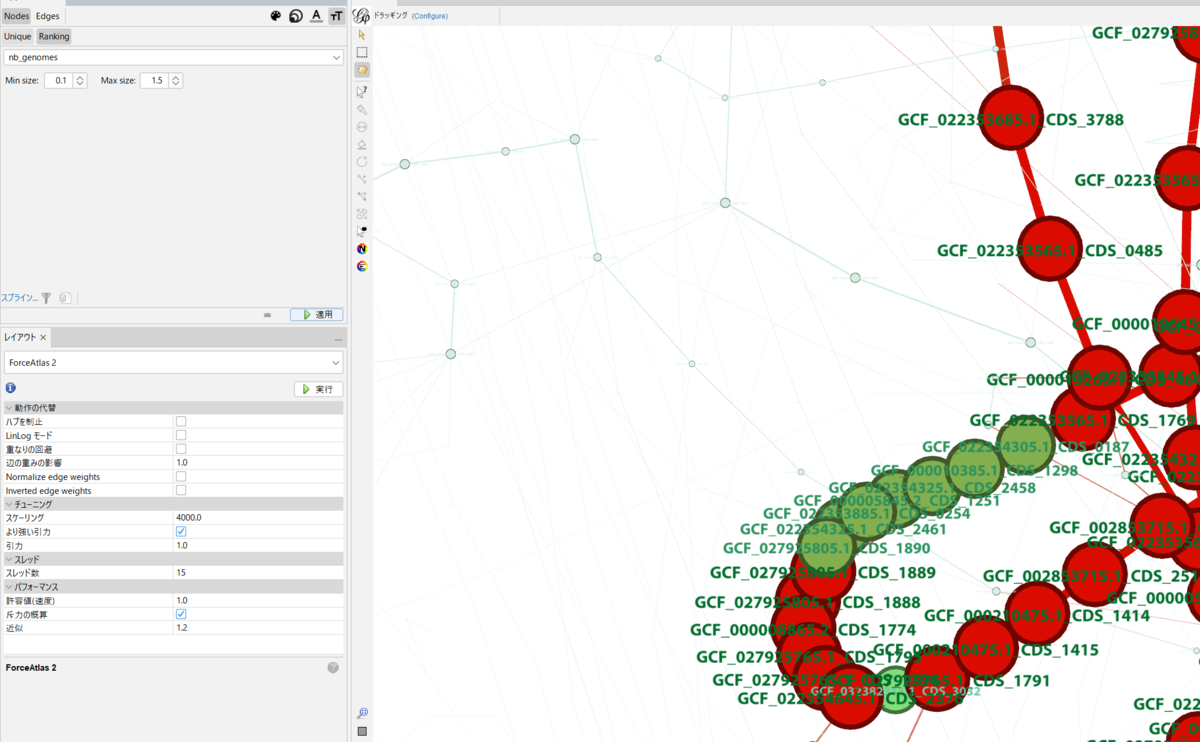
注;反映させるには下のボタンでTextを選択しておく必要がある。

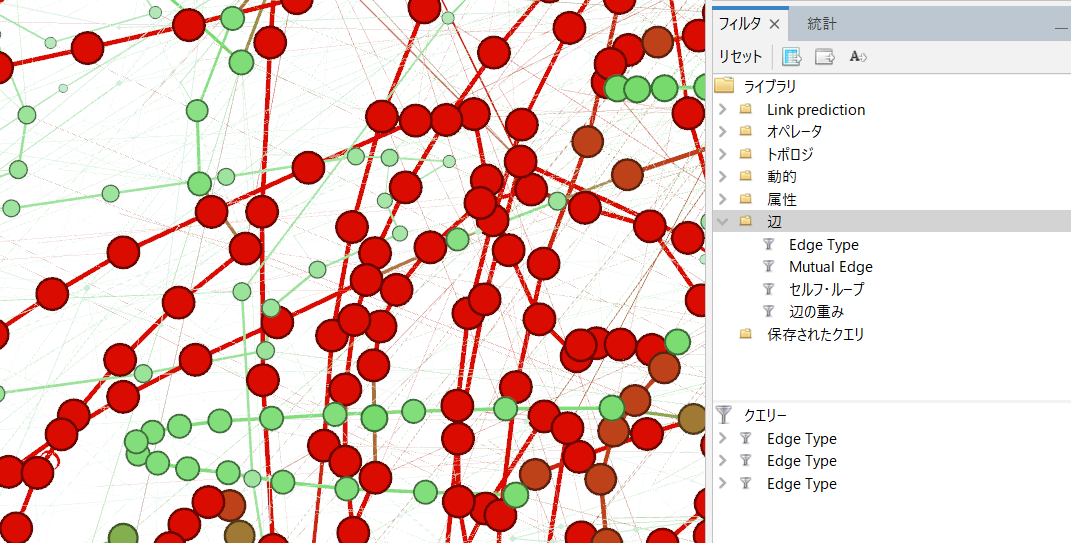
フィルタからは条件を指定してフィルタリングが行える。
統計タブではいくつかの統計テストを実行できる(link)。
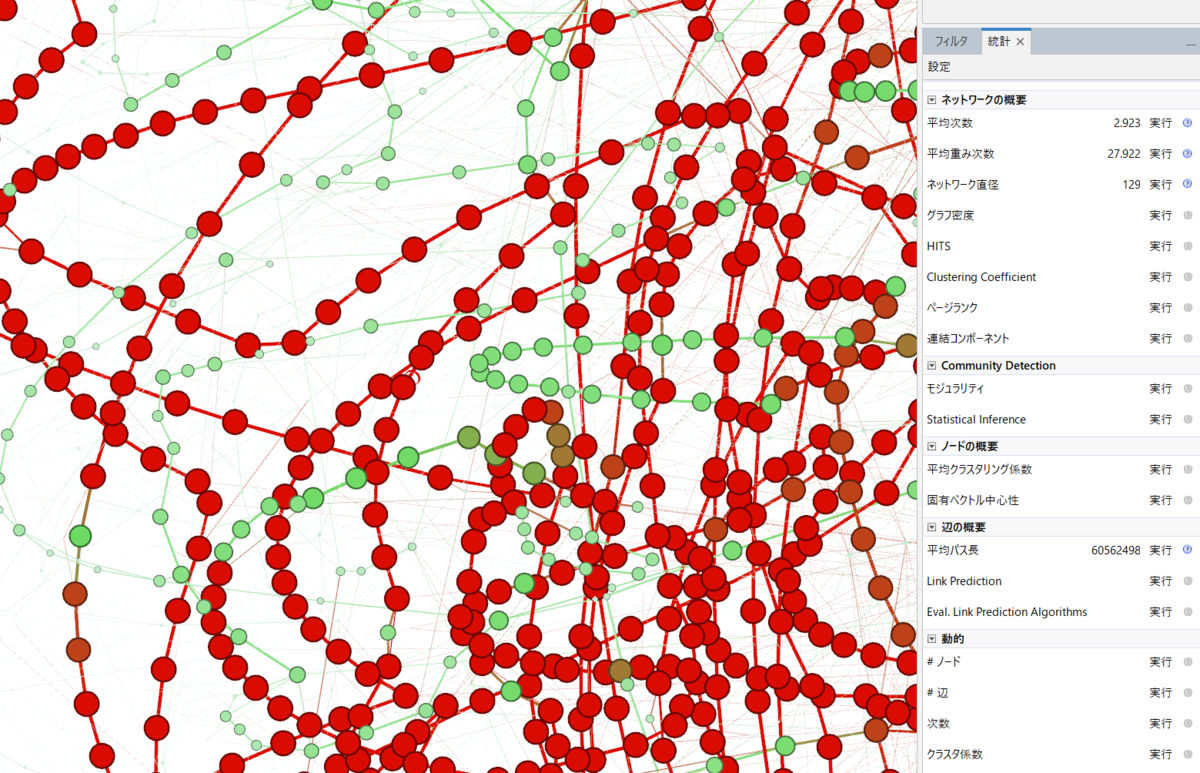

一部の統計テストやクラスタ係数の実行後は、その結果で色付けできる。
計算したクラスタ係数でカラーリング(ここでは疎なノードを強調)
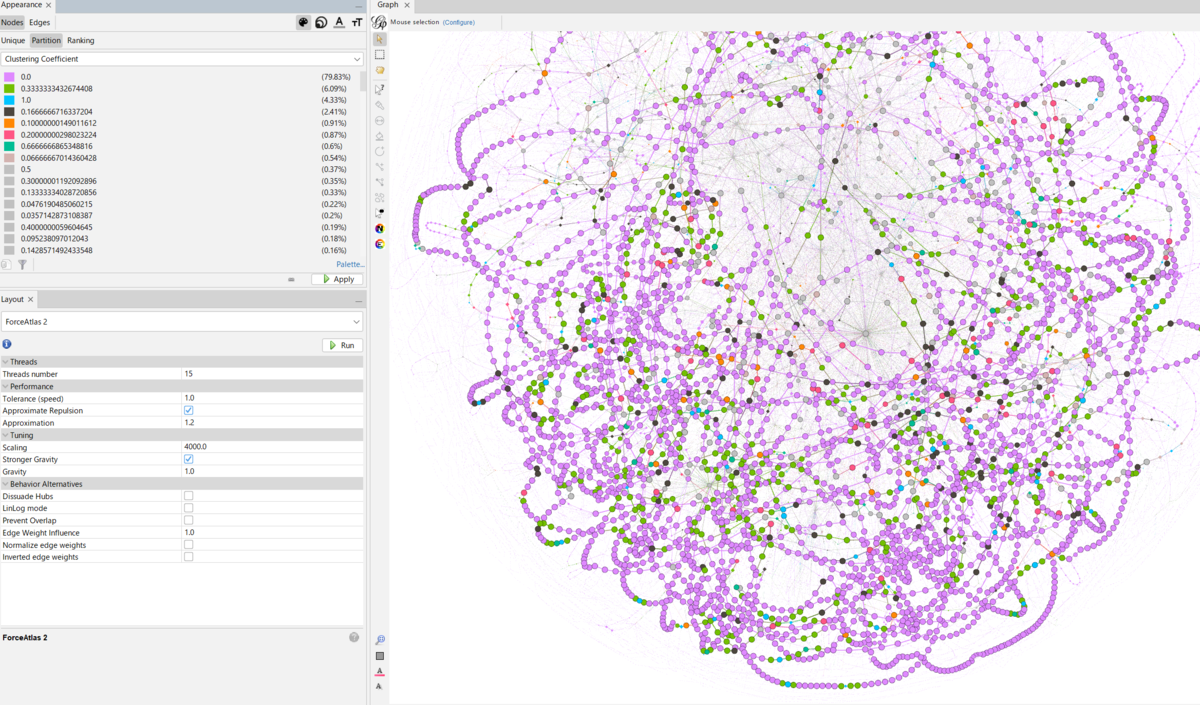
重み付き次数でカラーリング

パネル左のInspectorツールを選択後、ノード上にホバーすると情報を得られる。
コメント
自分がこのソフトウェアを知ったのは10年くらい前の学会のポスターです。中国の方がこのソフトは大きなグラフを扱えることを教えてくれてよく印象に残っています。
引用
Bastian M., Heymann S., Jacomy M. (2009). Gephi: an open source software for exploring and manipulating networks. International AAAI Conference on Weblogs and Social Media.
*1 light.gexfファイルにはノードとしての遺伝子ファミリーとその関係を表す遺伝子ファミリー間のエッジが含まれる。.gexfファイルも同じものが含まれるが、各遺伝子の詳細と遺伝子ファミリー間の関係も含まれる。通常の.gexfファイルは操作が非常に重くなるため、軽いlight.gexfファイルが用意されている(light.gexfはパンゲノムのグラフを操作するために使う)。
参考
関連