進化研究においては、生命のあらゆる領域にわたるゲノム情報の広範な調査が必要である。GenBankを通じて多数のゲノムが利用可能であるにもかかわらず、ゲノム情報の効果的な可視化や比較は、そのサイズなど多くの理由から困難である。ゲノムファイルを解析するための包括的なソフトウェアツールであるゲノムベースの検索・解析パーサーと、National Center for Biotechnology InformationのRefSeqデータベースで利用可能な全ての生物について、注意深くキュレートされた高品質のゲノム統計情報の広範なコレクションを収容するオンラインデータベースを紹介する。ユーザーは、直接検索するか、あらかじめ分類されたグループから好みの生物を選択してデータを取得することができ、出力は、異なるゲノムエレメント(例えば、コード配列、イントロン、tRNA、rRNA、ncRNAなど)について個別に計算された200以上の列の有用なゲノム情報(塩基数、GC含量、シャノンエントロピー、コドン使用率など)を含む表として生成される。データは染色体、ミトコンドリア、プラスティド、プラスミド配列ごとに独立して表示される(該当する場合)。すべてのデータはデータベース上で可視化でき、カンマ区切りの値またはExcelファイルとしてダウンロードできる。ゲノムベースの検索・解析パーサデータベースは登録不要で無料でアクセスでき、http://tacclab.org/gbrap/ で公開されている。
INFO
https://tacclab.org/gbrap/info.html
https://tacclab.org/gbrap/にアクセスする。
種名やRefseq IDで検索できる。
注;データベースの効率を上げるため、細菌名はドロップダウンリストに含まれていない。細菌データにアクセスするには、細菌タブにアクセスする。
出力例
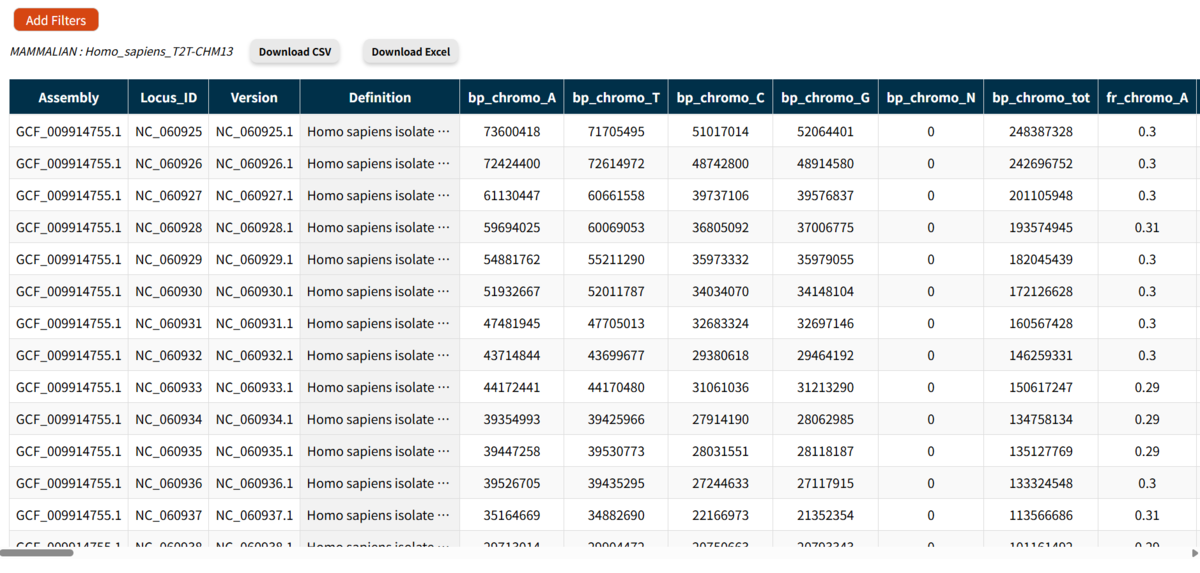
excel形式でダウンロードして開いた。
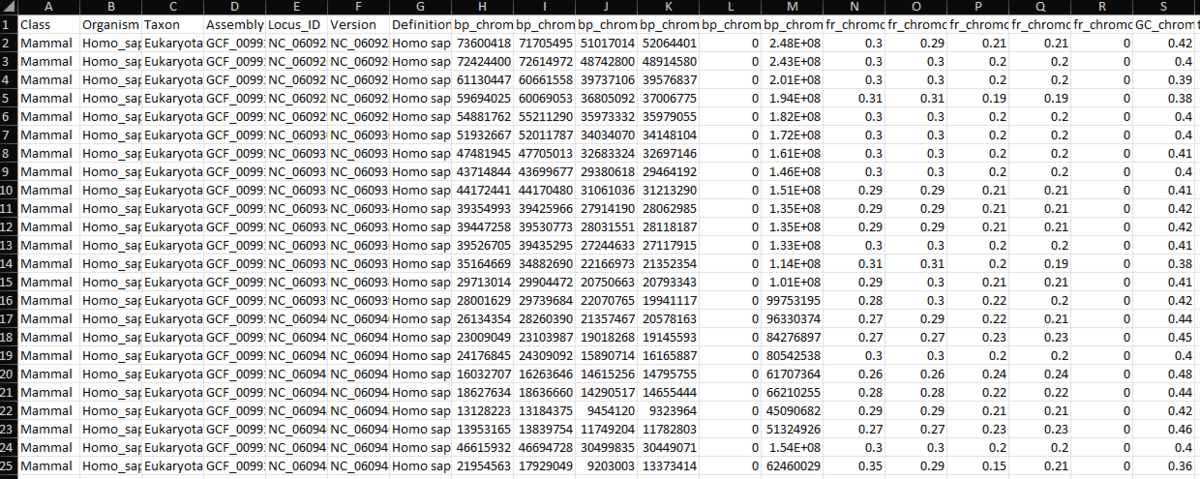
以下の列がある。
Class Organism Taxon Assembly Locus_ID Version Definition bp_chromo_A bp_chromo_T bp_chromo_C bp_chromo_G bp_chromo_N bp_chromo_tot fr_chromo_A fr_chromo_T fr_chromo_C fr_chromo_G fr_chromo_N GC_chromo topo_entropy_chromo chargaff_pf_chromo chargaff_ct_chromo shannon_chromo n_gene_pos n_gene_neg n_gene_tot bp_gene_A bp_gene_T bp_gene_C bp_gene_G bp_gene_N bp_gene_tot fr_gene_A fr_gene_T fr_gene_C fr_gene_G fr_gene_N GC_gene topo_entropy_gene chargaff_pf_gene chargaff_ct_gene shannon_gene bp_gene_overlap_tot n_cds_pos n_cds_neg n_cds_tot bp_cds_A bp_cds_T bp_cds_C bp_cds_G bp_cds_N bp_cds_tot fr_cds_A fr_cds_T fr_cds_C fr_cds_G fr_cds_N GC_cds topo_entropy_cds chargaff_pf_cds chargaff_ct_cds shannon_cds bp_cds_overlap_tot bp_cds_intron_A bp_cds_intron_T bp_cds_intron_C bp_cds_intron_G bp_cds_intron_N bp_cds_intron_tot fr_cds_intron_A fr_cds_intron_T fr_cds_intron_C fr_cds_intron_G fr_cds_intron_N GC_cds_intron topo_entropy_cds_intron chargaff_pf_cds_intron chargaff_ct_cds_intron shannon_cds_intron bp_cds_intron_overlap_tot n_ncRNA_pos n_ncRNA_neg n_ncRNA_tot bp_ncRNA_A bp_ncRNA_T bp_ncRNA_C bp_ncRNA_G bp_ncRNA_N bp_ncRNA_tot fr_ncRNA_A fr_ncRNA_T fr_ncRNA_C fr_ncRNA_G fr_ncRNA_N GC_ncRNA topo_entropy_ncRNA chargaff_pf_ncRNA chargaff_ct_ncRNA shannon_ncRNA bp_ncRNA_overlap_tot bp_nc_intron_A bp_nc_intron_T bp_nc_intron_C bp_nc_intron_G bp_nc_intron_N bp_nc_intron_tot fr_nc_intron_A fr_nc_intron_T fr_nc_intron_C fr_nc_intron_G fr_nc_intron_N GC_nc_intron topo_entropy_nc_intron chargaff_pf_nc_intron chargaff_ct_nc_intron shannon_nc_intron bp_nc_intron_overlap_tot n_tRNA_pos n_tRNA_neg n_tRNA_tot bp_tRNA_A bp_tRNA_T bp_tRNA_C bp_tRNA_G bp_tRNA_N bp_tRNA_tot fr_tRNA_A fr_tRNA_T fr_tRNA_C fr_tRNA_G fr_tRNA_N GC_tRNA topo_entropy_tRNA chargaff_pf_tRNA chargaff_ct_tRNA shannon_tRNA bp_tRNA_overlap_tot n_rRNA_pos n_rRNA_neg n_rRNA_tot bp_rRNA_A bp_rRNA_T bp_rRNA_C bp_rRNA_G bp_rRNA_N bp_rRNA_tot fr_rRNA_A fr_rRNA_T fr_rRNA_C fr_rRNA_G fr_rRNA_N GC_rRNA topo_entropy_rRNA chargaff_pf_rRNA chargaff_ct_rRNA shannon_rRNA bp_rRNA_overlap_tot ATG AAG GTA ACT GCA GAG GCT ATT TCC TGG AAT GAA TCA ACG AGT AAC TCT GTG TTC TTT CTG GGT CTC GAT CAG ACC CTA TTG TAT GGA ATC CTT GTC ATA ACA GAC CAC CCC TAC GCC AGC CGC AAA GGC TGC GTT GGG AGA TGT CAT TCG GCG TTA CCT AGG CCA TAG CGG CCG CGT CAA TAA TGA CGA
塩基頻度、ゲノムサイズ、またはGC含量、コドン使用率、Shannon entropy score、Chargaffの第2パリティルール(PR2)(二本鎖DNAの各一本鎖において、アデニン(A)の割合はチミン(T)とほぼ等しく、シトシン(C)の割合はグアニン(G)とほぼ等しいというもの(Rudner et al.1968))、ncRNA、tRNA、rRNA、最後にはtriplets(ATGやGTAなど)の出現頻度の列も含まている。GBRAPで説明されている。
上のメニューからは各大分類にアクセスできる。
Birds
その他
- scaffold'、'unlocalized'、'contig'、'unplaced'、'patch'、'unknown'のゲノムは、解析中に完全に削除されていて利用できない。このため、生物によってはミトコンドリアのデータしかない場合がある。
引用
GBRAP: A Comprehensive Database and Tool for Exploring Genomic Diversity Across All Domains of Life Open Access
Sachithra Kalhari Yaddehige , Chiara Vischioni , Michele Berselli , Leonardo Alberghini , Massimo Mezzavilla , Tania Bobbo , Cristian Taccioli
Molecular Biology and Evolution, Volume 42, Issue 6, June 2025