エンリッチメント解析は、ゲノムワイド実験で得られた遺伝子セットを解析するための一般的な手法である。ここでは、Enrichrと呼ばれるこの分野のツールの1つを大幅に更新した。Enrichrには、現在、解析やダウンロードが可能な多様な遺伝子セットライブラリの大規模なコレクションが含まれている。Enrichrは現在、102の遺伝子セットライブラリから180 184のアノテーションされた遺伝子セットを含んでいる。Enrichrには、ファジーセットの送信機能、BEDファイルのアップロード、アプリケーション・プログラミング・インターフェースの改善、クラスターグラムとしての結果の視覚化などの新機能が追加されている。Enrichrは、キュレーションされた遺伝子セットの包括的なリソースであり、さらなる生物学的発見のために生物学的知識を蓄積する検索エンジンでもある。Enrichrは次のサイトで自由に利用できる: http://amp.pharm.mssm.edu/Enrichr.
help
https://maayanlab.cloud/Enrichr/help#basics
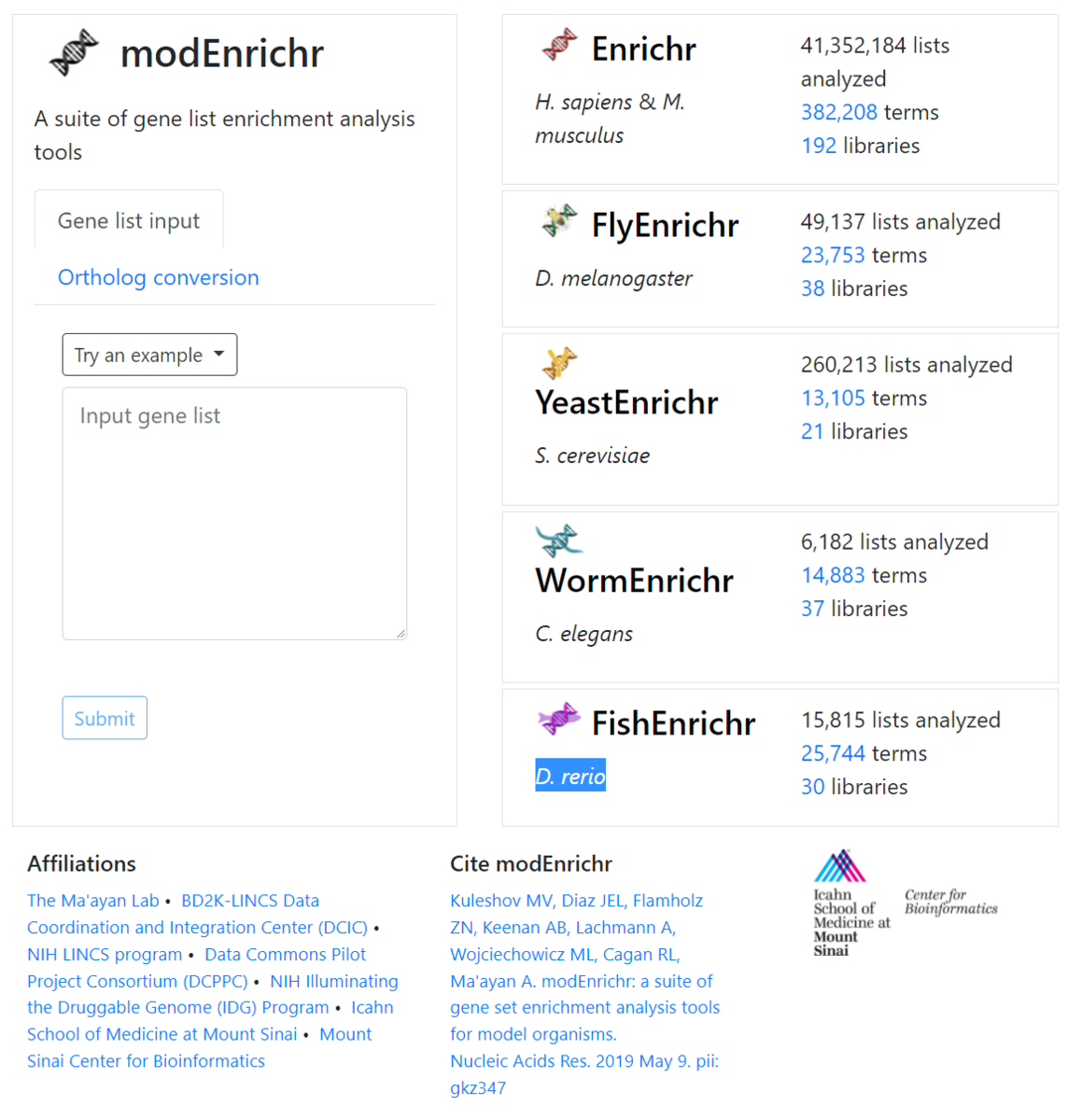
https://maayanlab.cloud/Enrichr/にアクセスする。ヒトとマウス(M. musculus)に対応している。
D. melanogaster、C. elegans、S. cerevisiae、D. rerioにも対応している。
https://maayanlab.cloud/modEnrichr/
Entrez遺伝子シンボルのセットをテキストボックスにペーストする。各遺伝子シンボルは1行ずつ入力する。ここではExampleのSTAT3データを指定した(変動遺伝子TOP100 )。
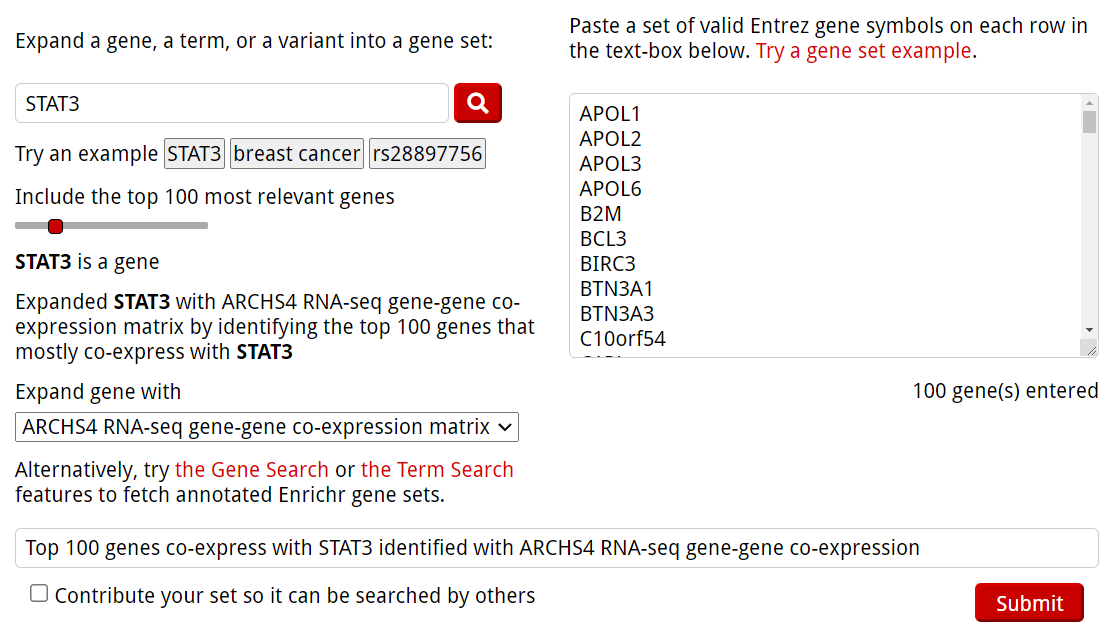
大規模なデータセットの発現行列と比較して共発現している遺伝子のエンリッチメント解析を行う。ここではARCHS4(ヒトとマウスの公開RNA-seqデータの大部分を均一な条件で解析して公開している)を指定。

submitをクリック。
出力例
結果ページでは、分析結果が充実したカテゴリーに分けて表示される。左端はTranscriptionのタブになっている。

結果ページにある各エンリッチメント・タームは、その遺伝子セット・ライブラリによって構成されている。これらのライブラリは、発表された研究や主要な生物学的および生物医学的オンラインデータベースなどの多くの情報源から構築されている。Enrichrのために作成され、Enrichrでしか利用できないものもある。
Transcriptionのタブでは、転写の調節に関わる遺伝子セットライブラリのエンリッチメント解析がリストアップされている。

クリックすると展開される。ちなみに、モバイル端末では左右のスワイプで表示内容を切り替えることができる。
Bar Graph

棒グラフの長さは、その特定の遺伝子セットや用語の重要性を表している。色の明るさも同じで、色が明るいほどその用語の重要性が高いことを示している。初期設定では、すべてのフィギュアの配色が赤になっているが、歯車アイコンをクリックするとカラーピッカーが表示され、配色を変更することができる(棒グラフ、Clustergramのグリッド、ネットワークに対応)。
Table

Enrichrでは、エンリッチメント結果を報告するために4つのスコア:p-value、q-value、rank (Z-score)、およびcombined scoreを実装しているが(詳細はbackgroundについて)、データテーブルではこれらのデータの一部を見ることができる。特定の用語を検索して結果をフィルタリングすることもできる。
Clustergram
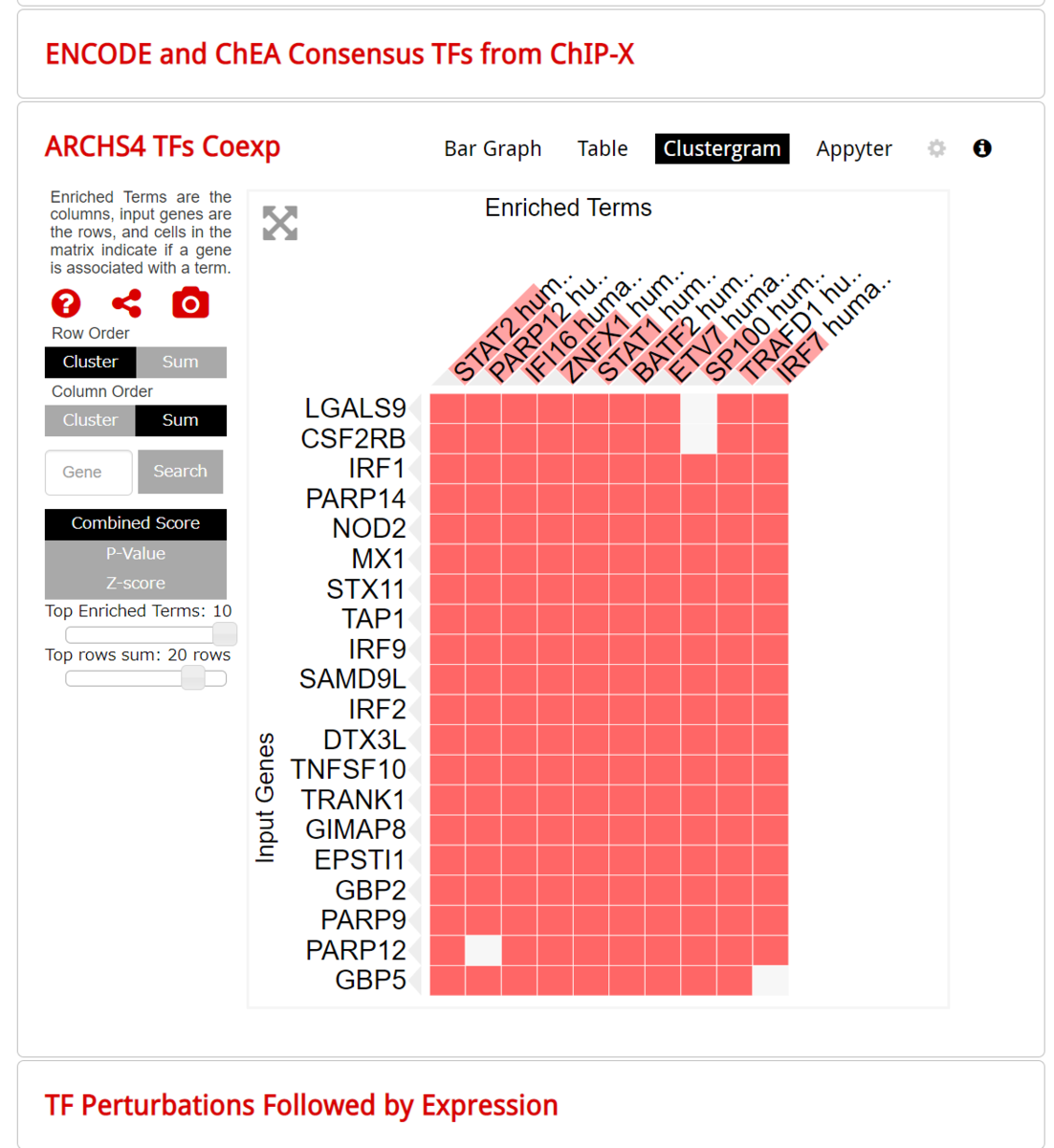
グリッドの各マスは用語を表し、他の用語との遺伝子セットの類似性に基づいて配置されている。マス目が明るいほどその用語の重要度が高いことを意味している。。グリッドをクリックすると、隣接する用語との相関スコアに基づいてグリッドを着色し、有意な用語を白いドットで表す別の表示に切り替えることができる。
Enrichrでは、結果を他の人と簡単に共有することができるようになっている。それには、左のメニューボックスの共有アイコンをクリックする。これにより、分析結果への一時的なダイレクトリンクが提供され、他の人と共有することができる。
Appyter

Enrichr Appyterを使用すると、エンリッチメント解析の結果をすぐに公開できるようなビジュアライゼーションを作成できる。
Appyter使用例


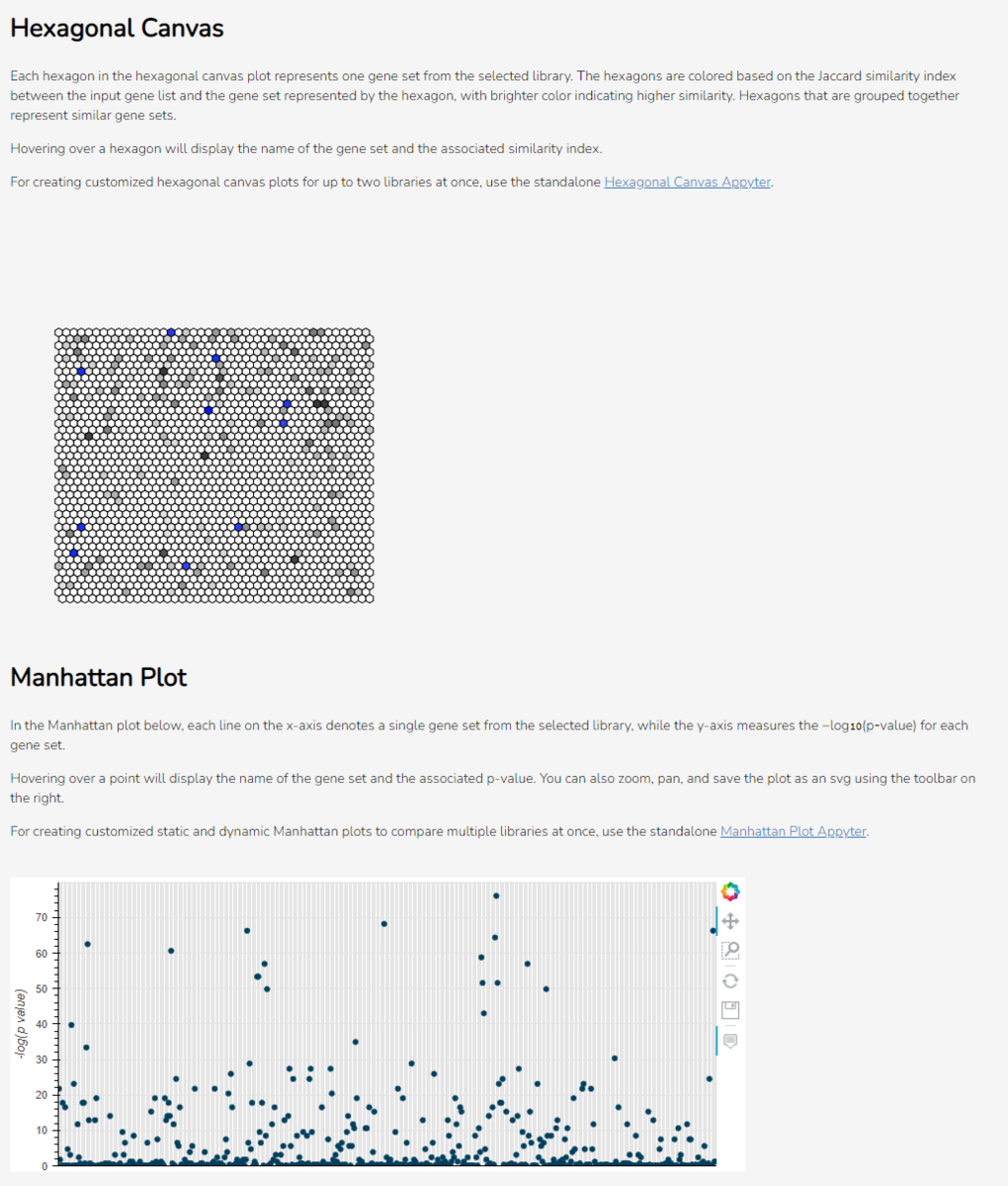

(Appytersカタログは、生物医学研究者がデータを様々な方法で分析し、視覚化することを可能にするソフトウェアプラットフォーム)
左から2番目のタブはPathwayとなっている。KEGGやPanther、Wikipathway、Reactomeなどの結果が表示される。
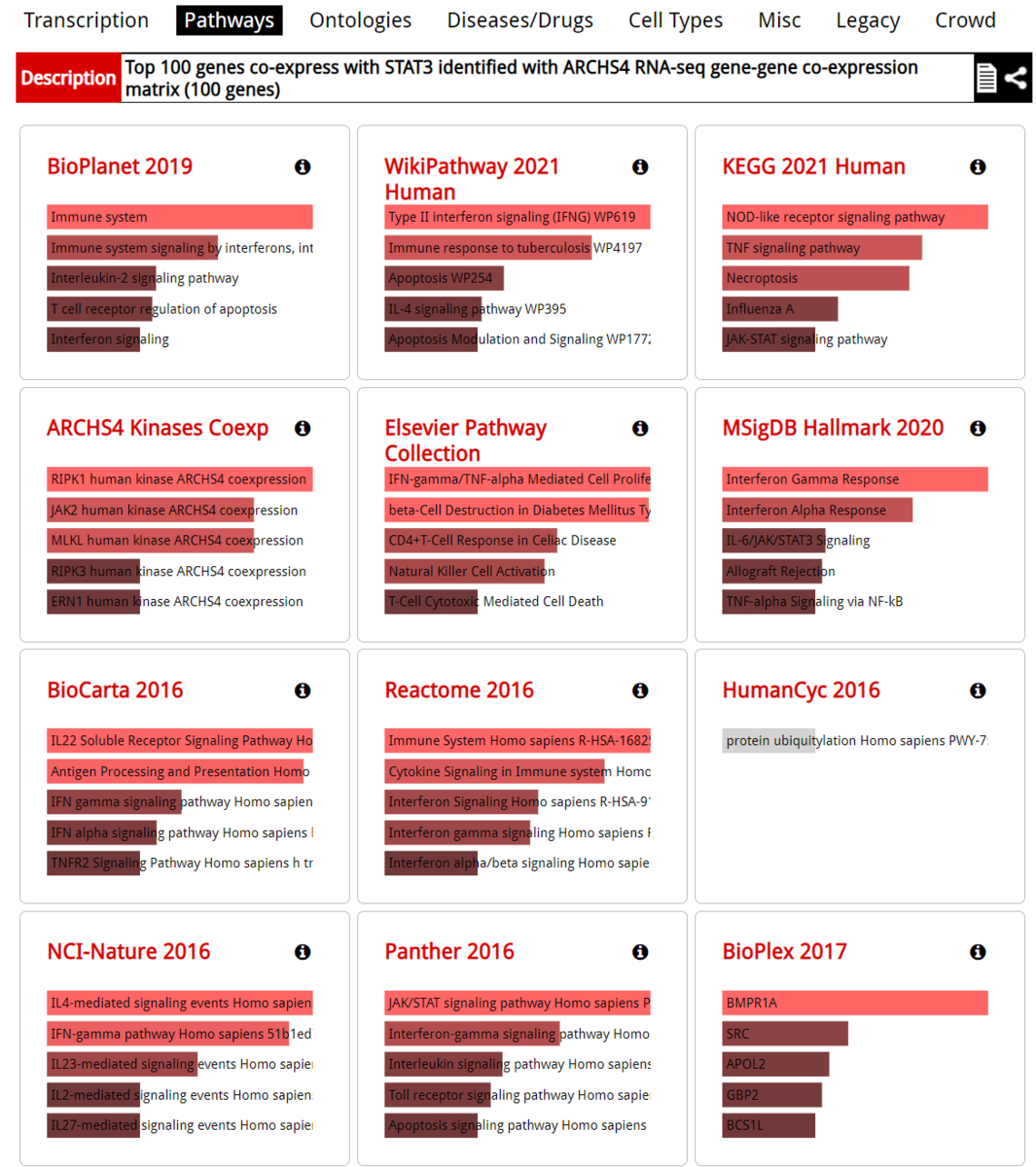
KEGG Human

左から3番目のタブはOntologies。GOなどがある。

4番目のタブはDiseases。ClinVarやCOVID-19などがある。
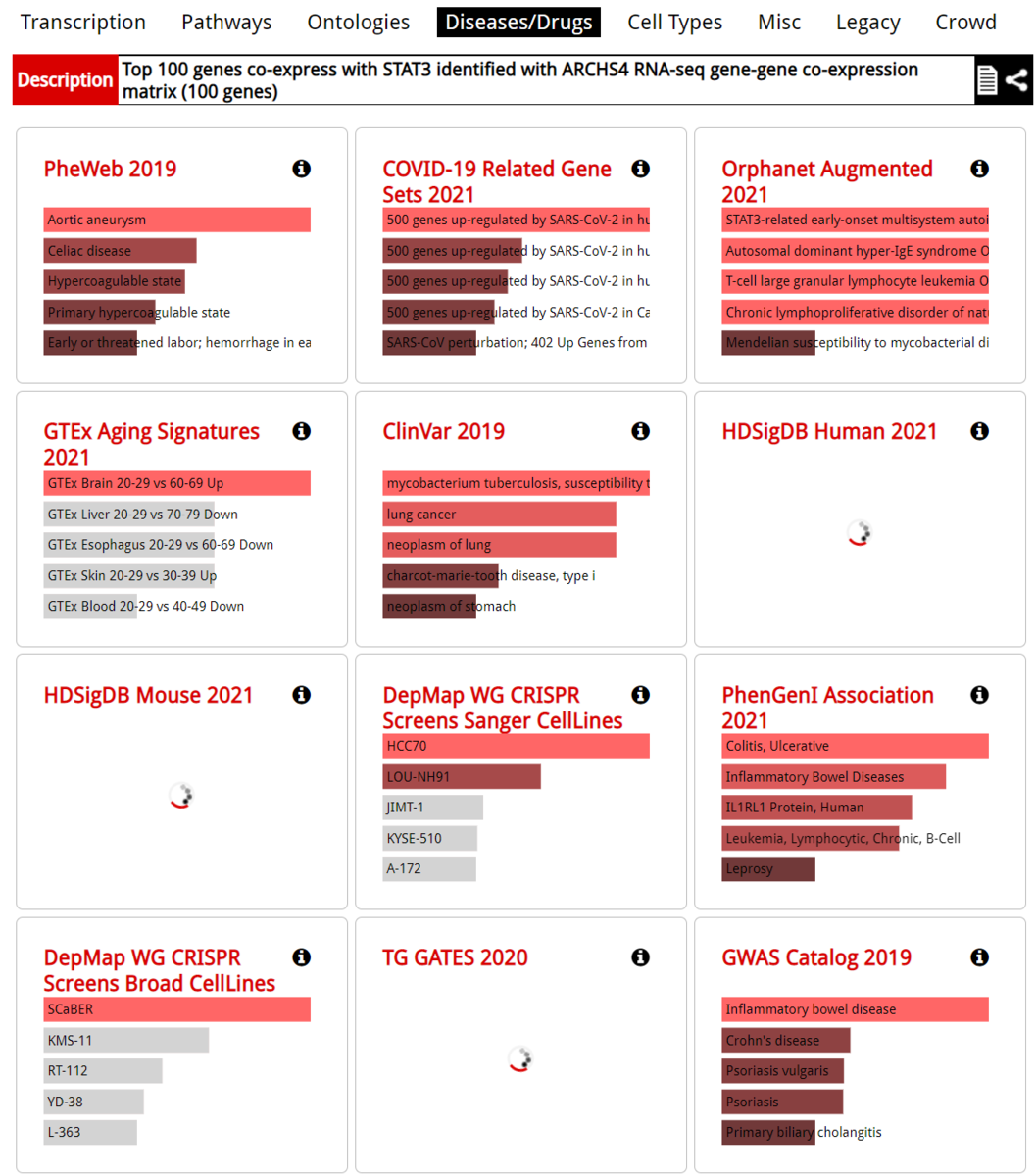
5番目のタブはcell type。

6番目のタブはmisc (多種類)。

7番目のタブはlegacy。KEGG2016などがある。

右端のタブはCrowd(雑多、混ぜ物)。

Enrichrでは、ユーザーアカウントを登録してログインすることもできる。アカウントを持つことで、結果をEnrichrサーバーに永久保存することができ、再アップロードの必要なくセットに戻ることができる。使用するには、ログイン後、セットを保存または共有ボタンをクリックする。これにより、自動的にアカウントと関連付けられる(helpより)。
引用
Enrichr: a comprehensive gene set enrichment analysis web server 2016 update
Maxim V. Kuleshov, Matthew R. Jones, Andrew D. Rouillard, Nicolas F. Fernandez, Qiaonan Duan, Zichen Wang, Simon Koplev, Sherry L. Jenkins, Kathleen M. Jagodnik, Alexander Lachmann, Michael G. McDermott, Caroline D. Monteiro, Gregory W. Gundersen, Avi Ma'ayan
Nucleic Acids Res. 2016 Jul 8; 44(Web Server issue): W90–W97
Enrichr: interactive and collaborative HTML5 gene list enrichment analysis tool
Edward Y Chen, Christopher M Tan, Yan Kou, Qiaonan Duan, Zichen Wang, Gabriela Vaz Meirelles, Neil R Clark, Avi Ma'ayan
BMC Bioinformatics. 2013 Apr 15;14:128
追記
modEnrichr: a suite of gene set enrichment analysis tools for model organisms Open Access
Maxim V Kuleshov, Jennifer E L Diaz, Zachary N Flamholz, Alexandra B Keenan, Alexander Lachmann, Megan L Wojciechowicz, Ross L Cagan, Avi Ma’ayan
Nucleic Acids Research, Volume 47, Issue W1, 02 July 2019, Pages W183–W190
関連