微生物は世界で最も豊富な生物と言われている。原核生物細胞は地球上に約4〜6×10^30個存在し、350〜550×10^15gの炭素を含むバイオマスを構成していると推定されている(1)。原核生物の総数は10^9種にも及ぶ(2)。1980年には、約1800種の細菌・古細菌の種名が承認された細菌名リストに掲載された(3)。その後、原著論文やInternational Journal of Systematic and Evolutionary Microbiology (IJSEM)の「Validation List」に掲載された名前が検証されてきた。2020年9月現在、この数は16,763件に増加している。
原核生物種の記述では、そのタイプストレインを指定する必要があり、その表現型や遺伝子型はよく特徴づけられて記述されていることが多い。タイプストレインから得られた16S rRNA遺伝子および全ゲノム配列は、表現型および化学的分類学的特徴とともに分類学的同定に利用される。このように、タイプストレインは、種の特徴付けや、分類学的な目的のための分離株や株の同定のための重要な参考文献となっている(4)。現在、16,763種のリストアップされた種からのタイプストレインは、130以上の培養コレクションから67,331のカタログ番号として入手可能である。
数十年前から、種の完全なデオキシリボ核酸(DNA)配列は、その系統を決定するための標準的な基準となることが認識されており、それによって分類学的分類が決定されている(5)。ゲノム配列の利用可能性が高まる中、原核生物分類学の重要な測定値として、平均アミノ酸同一性、平均ヌクレオチド同一性(ANI)、デジタルDNA-DNAハイブリダイゼーション(dDDH)などのゲノムベースの方法が開発されてきた(6-8)。2018年1月より、IJSEMでは、新規分類群の著者には、論文掲載の資格を得るために、新規分類群の記述付きゲノム配列を原稿に提供することを要求している。16S rRNAの類似度、ゲノム全体の類似度または距離、表現型や生理学的情報などのデータは、新種を同定するために組み合わせて使用される(9)。
微生物分類学のもう一つの重要な要素は、系統樹の再構築による系統関係の正しい評価である。表現型、化学的分類、遺伝子型の情報は微生物の同定には有用であるが、正確な系統樹を再構築するにはそのような情報では不十分であることが多い。微生物のゲノム配列の利用可能性が高まっているため、原核生物の起源と進化を研究するための系統関係をより包括的かつ正確に描写することが可能になっている。
そのゲノム、したがって代謝および機能的多様性のため、微生物はバイオテクノロジー研究のための理想的なモデルとして機能する。表現型や生理学的な情報を網羅的に収集した上で、タイプストレインのゲノム配列を解析することで、遺伝子と機能を結びつけることが可能となり、微生物の代謝や機能の可能性についての洞察を得ることができる。したがって、微生物のゲノムデータが蓄積されることは、バイオテクノロジー研究を大きく前進させることになる。
微生物学者の多大な努力と、The Genomic Encyclopedia of Bacteria and Archaea (GEBA) (10) のようなコミュニティシーケンシングプロジェクトのおかげで、公開されている型株のゲノム配列の数は急速に増加し続けている。現在、International Nucleotide Sequence Database Collaboration (INSDC)には12,000以上のゲノム配列が登録されている。しかし、多くの微生物のタイプストレインは、まだ配列決定されていない。そこで、世界微生物データセンター(WDCM)GCM 10Kプロジェクト(11)では、世界中の培養コレクションと協力して、有効に公開されている種の全ゲノムデータベースの現在のギャップを埋めるとともに、IJSEMと協力して、新たに記載された種に対して無料の配列決定とゲノムアノテーションサービスを提供している(12)。
微生物ゲノム情報の増加に伴い、これらのデータをホストし、解析するデータベースやサーバーは拡大を続けている。ゲノムオンラインデータベース(GOLD)(13)は、統合微生物ゲノム(IMG)(14)と連携して、微生物ゲノムの包括的なカタログを提供し、微生物ゲノムと微生物ゲノムの解析のためのプラットフォームを提供している。Type (Strain) Genome Server (15)は、包括的な原核生物メタデータリソースBacDive (16)とLPSN (17)に接続されており、ゲノムベースの原核生物分類学のためのハイスループットなウェブサーバーと考えられている。しかし、最新のタイプストレインとそれに関連する包括的なゲノム情報だけでなく、ユーザーフレンドリーな検索可能で比較可能な機能を提供するデータベースがまだ必要とされている。
タイプストレインのゲノム配列へのアクセスを容易にし、その価値を最大化するために、 GCM 10K type strain sequencing projectでは、gcTypeプラットフォームを開発した。このプラットフォームは、他のデータベースから公開されている情報とGCM 10Kプロジェクトのシークエンシングの取り組みを厳格な品質管理基準に従って統合し、強力な標準データ処理パイプラインにより、ゲノム解析やゲノムベースの種同定のためのウェブベースのデータ解析パイプラインを提供する高品質のリファレンスデータベースを生成する。さらに、分類学的、表現型、生理学的情報を型株と関連付けることで、ユーザーが包括的なゲノム解析や機能解析を行うことができる。全体として、gcTypeは、微生物分類学者や微生物学者が微生物のタイプストレインの配列に関する最新情報を収集することを可能にするユニークで有用なリソースである。
gcType Helpより
ゲノム情報は、微生物の特徴を総合的に解読し、メタゲノムを用いて微生物を探索し、自然や人間の生活の基本的な疑問に答えるための分類学的、系統学的、進化学的、機能学的研究を支えるために必要不可欠な情報です。微生物の分類学、進化、機能に関する研究は、今やゲノム時代に突入しています。しかし、国際原核生物体系学委員会(ICSP)で有効な名称が公表されている細菌種や古細菌種のゲノム配列情報にはまだ大きなギャップがあります。真菌類の場合は、さらに大きなギャップが残っています。Global Catalogue of Microorganisms (GCM)の主導による型株配列決定プロジェクトは、微生物のゲノムマップにおける現在のギャップを埋め、ゲノムデータのディープマイニングを通じた研究を促進するための国際的な協調的な取り組みです。本プロジェクトには、2つの中核的なサブプロジェクトが含まれており、1万個の細菌・古細菌タイプ株の配列決定、真菌タイプ株の配列決定、そして特定の科学的目標に基づいたいくつかのサテライトプロジェクトがあります。現在、14の国と地域から20以上の培養コレクションがこのプロジェクトに参加していることが確認されています。
また、関連分野の微生物学者や研究機関からのゲノムデータに関連した研究課題の提出も歓迎しています。詳細については、Dr. Linhuan Wu(wulh@im.ac.cn)までお問い合わせください。
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkaa957/5943199
Species with vauldly published names
Number of type strain genomes(2020年12月現在12,913)
1つクリックしてみる。
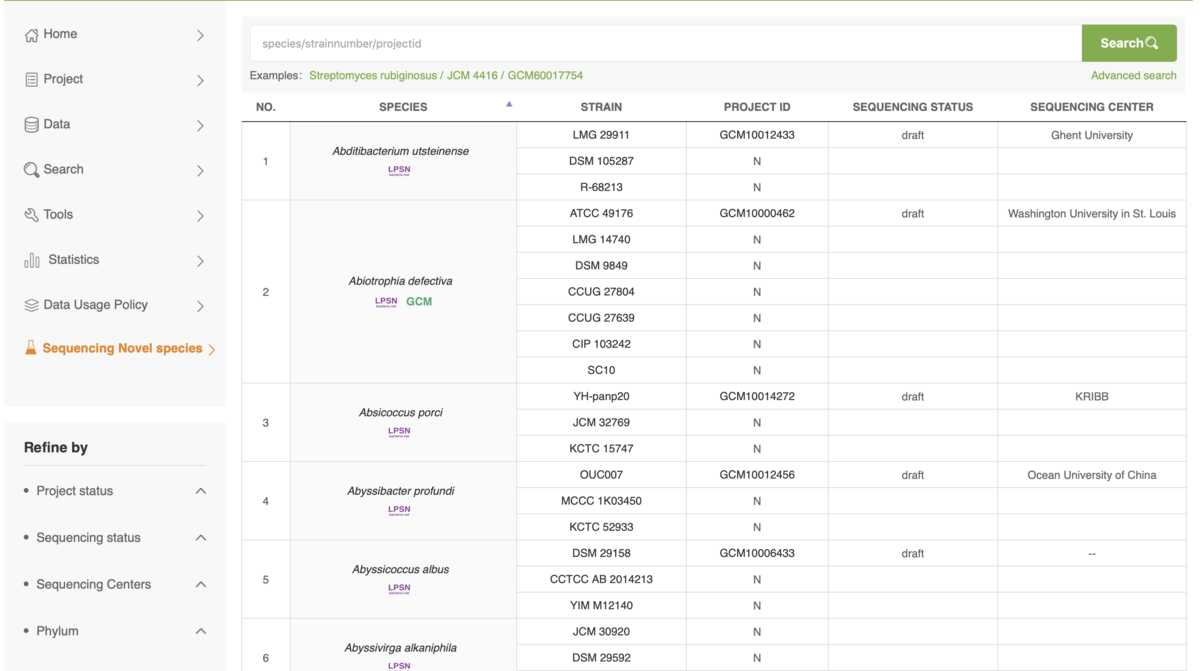
Abditibacterium utsteinense / LMG 29911
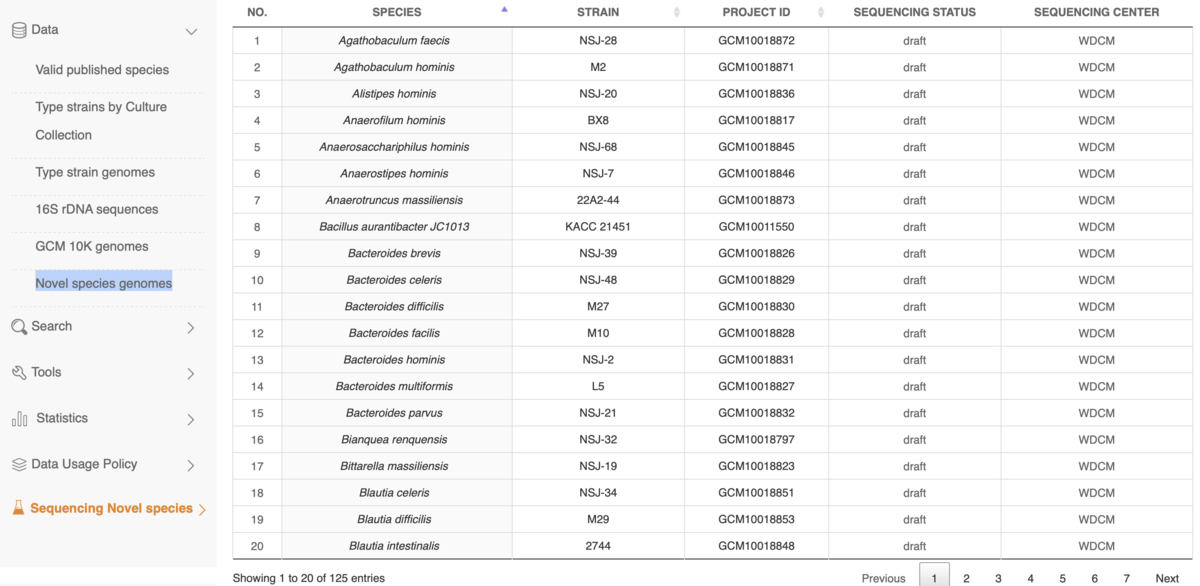
種名と存在するタイプ株が表示されている。GCMのprojectが存在する場合はproject IDとシークエンシングステータス(draft or complete)が示される。種名下の赤字はLPSNへの該当する種へのリンクで、緑の文字はGCNへのリンクになる。

左のメニューからは16S rRNA配列だけの検索もできる。

GCM 10K genomes
Novel species genomes
Searchsからは16S rRNAのBLAST検索もできる。

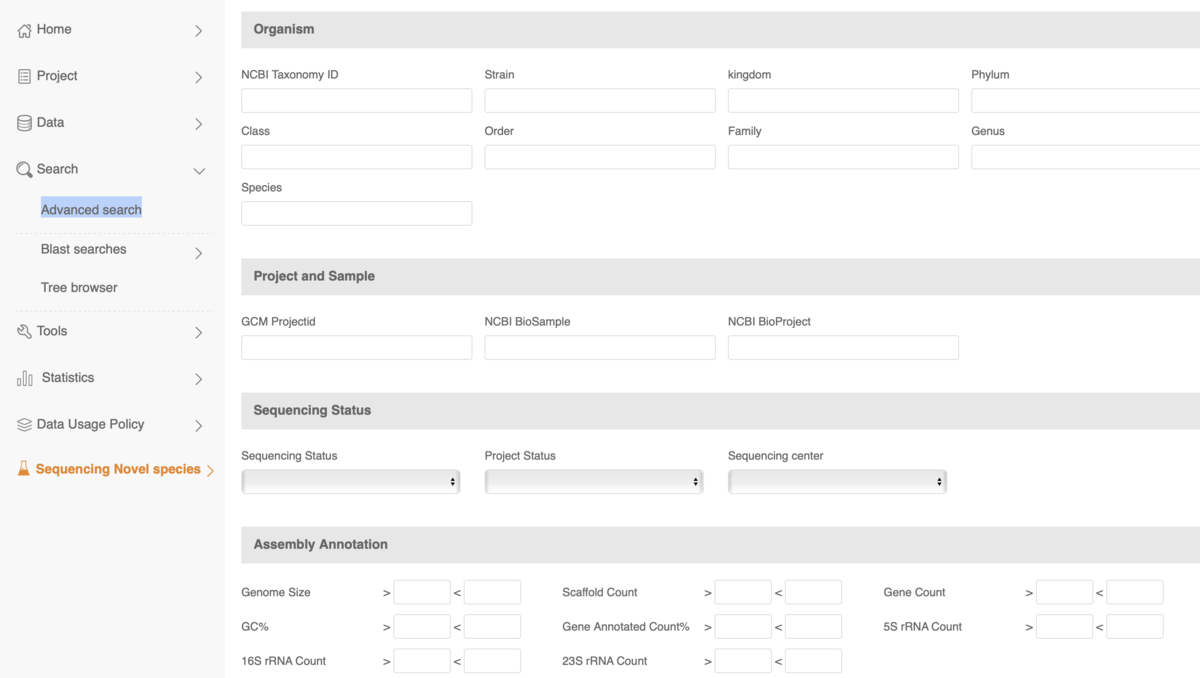
Advanced search
SOPs (Sample Submission for GCM Projects)
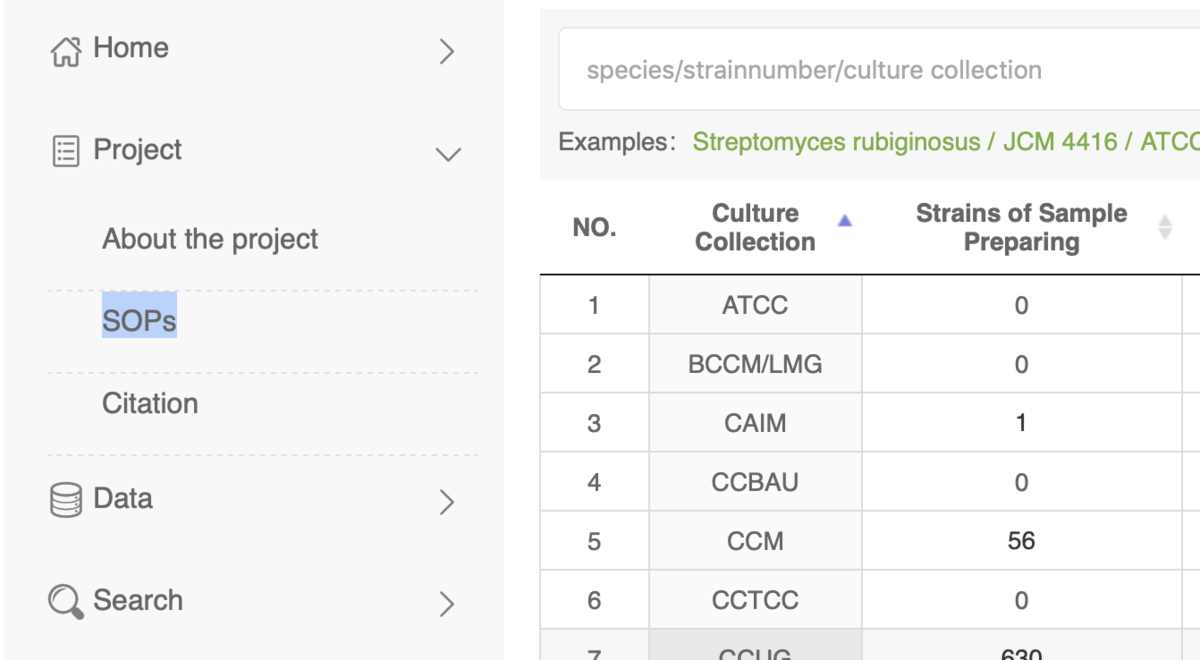
SOPsをクリックするとサンプル提出の流れをまとめたPDFマニュアルが表示される。
他にもいくつかの機能があります。アクセスしてみて下さい。
引用
gcType: a high-quality type strain genome database for microbial phylogenetic and functional research
Wenyu Shi, Qinglan Sun, Guomei Fan, Sugawara Hideaki, Ohkuma Moriya, Takashi Itoh, Yuguang Zhou, Man Cai, Song-Gun Kim, Jung-Sook Lee, Ivo Sedlacek, David R Arahal, Teresa Lucena, Hiroko Kawasaki, Lyudmila Evtushenko, Bevan S Weir, Sarah Alexander, Dlauchy Dénes, Somboon Tanasupawat, Lily Eurwilaichitr, Supawadee Ingsriswang, Bruno Gomez-Gil, Manzour H Hazbón, Marco A Riojas, Chatrudee Suwannachart, Su Yao, Peter Vandamme, Fang Peng, Zenghui Chen, Dongmei Liu, Xiuqiang Sun, Xinjiao Zhang, Yuanchun Zhou, Zhen Meng, Linhuan Wu, Juncai Ma
Nucleic Acids Research, Published: 29 October 2020
関連
