1随時更新
・2024/05/24 古いSNS情報を整理
MinION(ミナイオン)でシーケンスする流れを説明する。

ナノポアに関しては模索中の段階です。書いていることが必ずしも正しいとは限らないことに注意してください。
MinION (Oxford Nanopore) - wiki
公式ツイッター
LONDON CALLING 2022
TOPICやPRESENTATION TYPEでスピーカーの絞り込みが可能
追記
動画マニュアルリンク削除 & HTMLマニュアルについて追記
公式のリンクをいくつか貼っておく。
White papers
https://nanoporetech.com/resource-centre/white-papers
Videos
https://nanoporetech.com/resource-centre/videos
Posters
https://nanoporetech.com/resource-centre/posters
London Calling 2017
https://nanoporetech.com/events/ncm17/watch
London calling 2018 (2018 5/24-5/25)(動画のほか、ダウンロード可能なposterもアップされています)
https://londoncallingconf.co.uk/lc18
Rapid Lambda Control Experiment (SQK-RAD004) protocol PDF
https://store.nanoporetech.com/media/wysiwyg/Rapid_Lambda_Control_Experiment_SQK-RAD004_.pdf
DNA extraction protocol
https://community.nanoporetech.com/extraction_methods
2018 3/27 追記 3/27に登録されたPreprintです。樹木(Eucalyptus pauciflora)を材料にしています。DNA抽出とライブラリ調整前に読むべき内容です。
A comprehensive toolkit to enable MinION sequencing in any laboratory
https://www.biorxiv.org/content/biorxiv/early/2018/03/27/289579.full.pdf
2017年10月現在、フローセル2個とrapid sequencing用の試薬が全て入ったお得なStarter kitを購入することができる。Rapid sequencing kitはLigation seqeucing kitよりスループットは劣るが(Ligation seqeucing kitの70-80%くらい出る)、トランスポザーゼでDNAを処理してアダプターをつけることで、ライブラリ調整を10分程度で行うことができる(PCR必要なし)。starter kitを購入すれば、追加投資がなしにシーケンスを行うことができる(後で記載したUSB3.0 typeCのケーブルは別途必要かも )。
https://store.nanoporetech.com/rapid-sequencing-kit.html
Rpaid sequencingには、DNAを500~1000ngくらい抽出しておく必要がある。DNAがあまり取れない生物では、これはデメリットになる。もう1つ注意点として、starter kitについてくるフローセルはR9.4の1D専用フローセルで変更不可のようである(未確認)。よりハイクオリティな2D(1Dx2)シーケンスを行うには、R9.5のフローセル(リンク)を購入する必要がある。また、オックスフォードナノポア社では、Minion以外のPromethIONやGridIONなどのシーケンサーも選択できる。ゲノムサイズが大きければこれらを選ぶのも手であるし、フローセルをまとめ買いすると少し安くなる様なので、Minionで十分量読むまでひたすらシーケンスする選択もあるかもしれない。
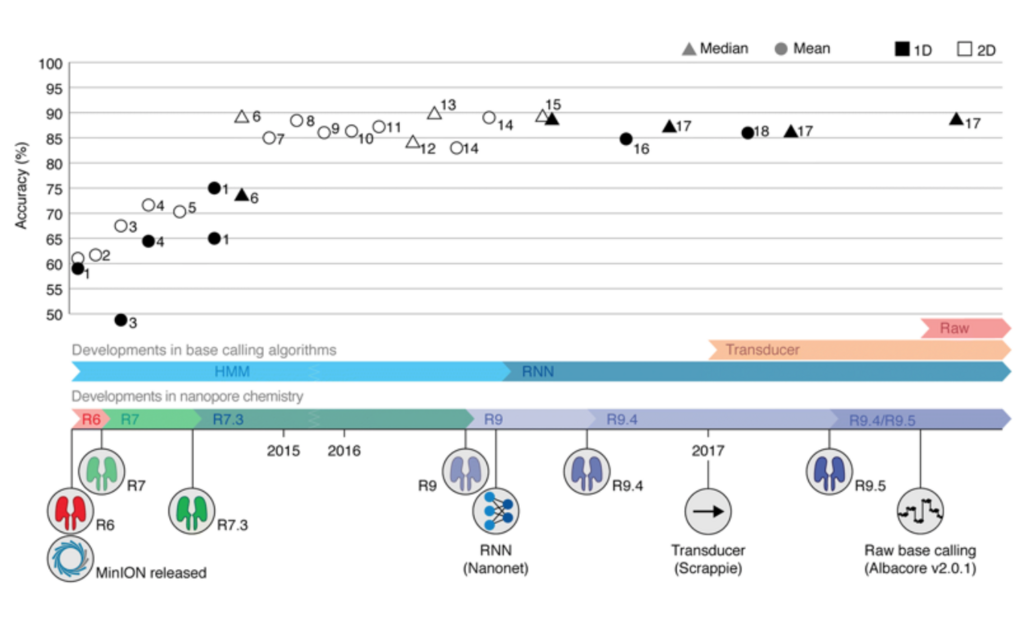
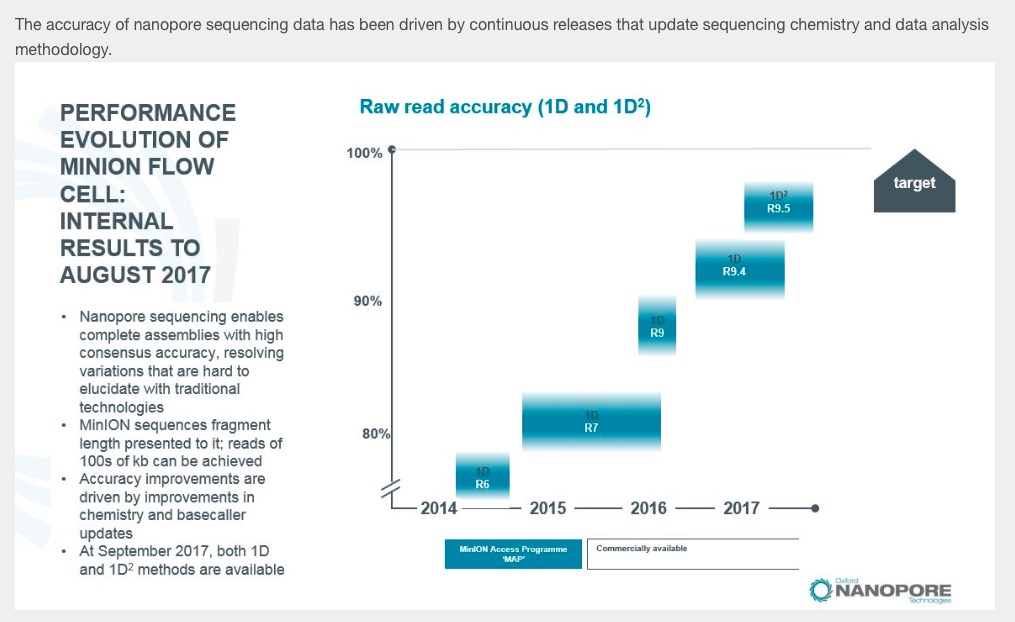
シーケンス精度の変化(平均と中央値)。
From squiggle to basepair: computational approaches for improving nanopore sequencing read accuracy (Franka J. Rang et al, 2018)より。
MinionをつなぐMACやWindowsのハードウエアチェック
https://nanoporetech.com/community/lab-it-requirements
必要な実験器具(外でやるなら工夫する必要がある)
https://nanoporetech.com/sites/default/files/s3/rapid-sequencing-requirements.pdf
MACやPCの設定
https://nanoporetech.com/sites/default/files/s3/MinION-Computer-Requirements-March-17_Final.pdf
DNA抽出
DNAの抽出は、コストを気にしないならカラムを使った方法が良いと思われる。例えば、Pacbioでは
- Qiagen MagAttract HMW kit (100-200 Kb)
- QIAGEN Genomic-tip 500/G (50-100 Kb)
- Qiagen Gentra Puregene kit (100-200 Kb)
などを推奨しているが、nanoporeでもこれらのキットが使える(*1)。ただし動物、植物、昆虫、真菌、バクテリア、ウィルスなど生物により夾雑物の種類は変わるので、一概には言えない。初めてナノポアを使うなら、論文をいくつか読んで、パフォーマンスが良さそうな方法を調べておいた方が良い。DNAが汚いと、全然読めない可能性もある(本当は失敗した方法が知りたいが、そういったデータは論文にならない)。
追記 上にもリンクを載せたが、ヒト、ブタ、ウサギの血清、Spinach、yeast、E.coliなどの DNA Extraction methodsのPDFがダウンロードできる。
https://community.nanoporetech.com/extraction_methods
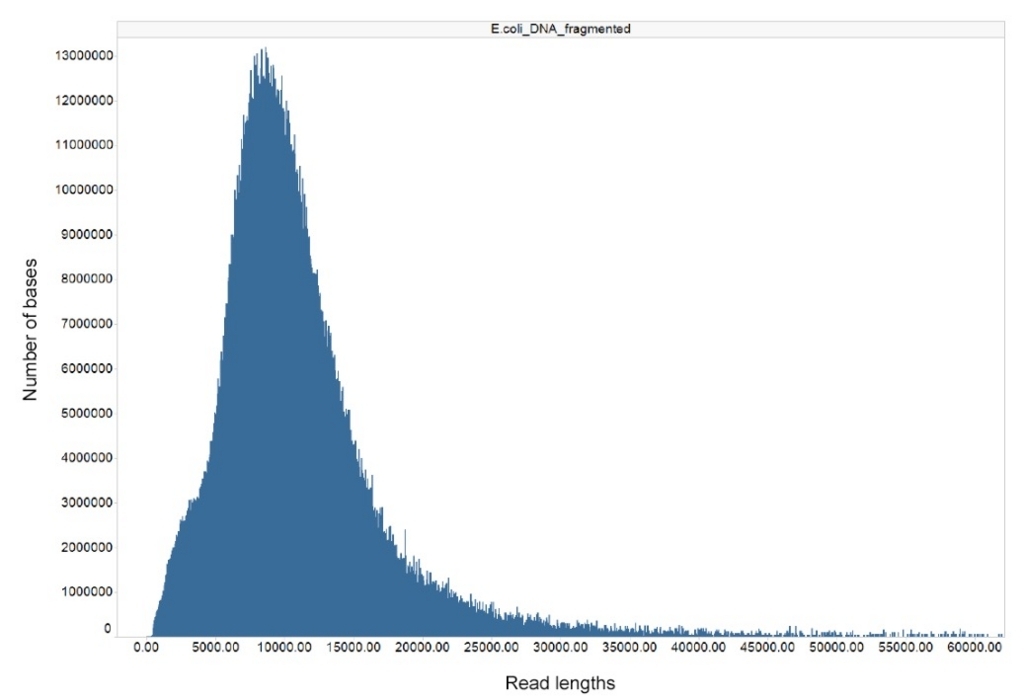
E.coliなら、 O.D0.5でクロラムフェニコール入れて1時間培養し、QIAGEN Genomic-tip 500/G でDNA抽出。Minionの出力は8Gb/48h。リード長は下(公式より転載)。
コストを気にするなら、磁気ビーズを使った高分子量gDNA抽出プロトコルも報告され始めている。
追記
まずこれを読む。大変勉強になる。
http://lab.loman.net/2018/05/25/dna-extraction-book-chapter/#__RefHeading___Toc505877554
追記 Protocols.ioにDNA抽出プロトコルが上がっています。
1、
This protocol gave a read N50 of 25kb on the MinION with decent yields (>5Gb with only 1000 pores) using spider legs @nanopore. No kits needed, pretty cheap. https://t.co/CtwjsowOrP
— BenjaminSchwessinger (@schwessinger) June 5, 2018
2、
3、
https://www.protocols.io/view/high-quality-dna-from-fungi-for-long-read-sequenci-k6qczdw
4、
5、ONT HPのExtraction methods(log inしておく必要あり。プロトコルのPDFをダウンロードできる)
https://community.nanoporetech.com/extraction_methods
6、High Molecular Weight DNA Extraction from Recalcitrant Plant Species for Third Generation Sequencing
7、Ultra-long Read Sequencing for Whole Genomic DNA Analysis
High molecular weight DNA isolation method from diverse plant species for use with Oxford Nanopore sequencing
2020 2/28 ビデオジャーナルの論文のリンクをいくつか張っておきます。
DNAの純度
Minionのシーケンスにはハイクオリティなlong DNAが必要である。
Oxford Nanopore社のHPに記載されているCriteriaを載せておく。
- Purity as measured using Nanodrop - OD 260/280 of 1.8 and OD 260/230 of 2.0-2.2
- Average fragment size, as measured by pulse-field, or low percentage agarose gel analysis >30 kb
- Input mass, as measured by Qubit - 10 pg
- No detergents or surfactants in the buffer
DNAの定量は、low bind tubeに溶かし、Qubitなどの蛍光法により行う。Nanodropなどを使った260nmの測定はRNA、NTP、ごく微量のフェノールなど様々な夾雑物に影響を受けるため、はっきり言って全くあてにならない。夾雑物が疑われるなどの理由でDNA抽出後に追加精製を行うなら、Pacbioのガイドで勧められているフナコシのPowerClean DNA Clean-Up Kitなどが良いかもしれない。土壌サンプル向けのカラムキットで多糖類や腐食酸も除けるようである。
追記 上にリンクを貼ったprepirntでは、260/230が"1程度"の低いサンプルは、シーケンススループットが1桁くらい落ちている(Preprint 表1)。
2019 6/19 追加
London Calling 2019: Stella Loke
Optimising Plant DNA extraction for nanopoe sequencing
DNAの長さ
長い切れていないDNAを抽出できれば、それだけ長いDNAを読む率が長くなる。カビのゲノム解析のペーパー(リンク)では、ゲノム抽出後にBlue Pippinでサイズセレクションすることで、平均サイズが大きく向上したと報告されている。また、このサイズセレクションを行うタイミングは、ゲノムをとった後でなくライブラリ調整後がベストらしい(Omics! Omics!: Nanopore Workshop Notes)。Blue Pippinは国内では日本ジェネティクス社から買えるようである(リンク)。大崎さんのBlue Pippin解説(リンク)。
高分子のDNAを回収するには、DNAの調整時の取り扱いも細心の注意を払う必要がある。ナノポアHPには以下のように記載されている。
- Using wide-bore pipette tips to handle the gDNA
- Mixing gently but thoroughly by inversion, as opposed to vortexing or pipetting
- Avoiding unnecessary freeze-thaw cycles
- Avoiding pH <6 and >9
- Avoiding high temperatures, which can lead to degradation
特に抽出後の確認の電気泳動でゲノムがスメアになっているようなサンプルは使うべきではない。
追記
カラムを使わず低コストに高分子量genomic DNAを抽出する方法
Extraction of high-molecular-weight genomic DNA for long-read sequencing of single molecules
https://www.future-science.com/doi/full/10.2144/000114460#
キットのプロトコル
https://community.nanoporetech.com/protocols
2019 1/11 追記
追加精製
どのような技術でDNA抽出したとしても、ラン前にSPRI技術で追加精製して2kb以上のDNA断片をエンリッチすることで、より効率的なシーケンシングが可能になります。詳細は、上にリンクを載せたExtraction methodsの、SPRI size selection protocol for >1.5-2 kb DNA fragmentsを確認して下さい。以下のHPも参考になります。
https://albertsenlab.org/all-i-want-for-christmas-is-a-terabase-of-nanopore-data/
2019 9/13
publishから時間が経ってますが、以下の論文にはシーケンシングを成功させるノウハウが詰まっています。植物を使っている人以外も読んだ方がいいと思います。
https://onlinelibrary.wiley.com/doi/full/10.1111/1755-0998.12938
ラン
簡単に流れを載せておきますが、Oxfordナノポアのシーケンス技術はコミュニティベースで進化し続けています。ラン前にプロトコルがバージョンアップしていないか、MinKNOWのアップデートはないか確認しておいて下さい。
https://store.nanoporetech.com/media/wysiwyg/Rapid_Lambda_Control_Experiment_SQK-RAD004_.pdf
0、ソフトウエアのインストール
log inしてから右側のsoftware Downloadsを選択。
端末で動くGuppyやAlbacoreもダウンロードできる。
1、ライブラリ調整
1DのR9.4フローセルのRAD-003のプロトコルに従う。~400 ng DNAを7.5µlのボリュームで用意する必要がある。
追記: input DNA量についても、上のPrepirntが詳しく触れられている。DNA repair and end-prepは、0.2pmolのDNAに対して最適化されていると書かれており、これは平均8kbの2本鎖DNAだと1ugになる。平均24kbだと3ugになる(Sequencing library preparationtyより)。
2、フローセルの準備。
フローセルを冷蔵庫から取り出し、ダミーセルと交換する。
3、ソフトの立ち上げ
MinionをつなぐPCから専用ソフトのMinKNOWを立ち上げる。MinKNOWは購入者のログインページから検索すれば見つかるので、前日までにインストールしておく。
MinKNOW立ち上げ時、ホストマシンはネットに接続して、さらにUSB3.0を介してMinionをつないでおく必要がある。
我々だけかもしれないが、MinionのUSB3.0 CタイプポートとPCをつなぐUSB3.0のケーブルが同封されていなかった(間違いでしたらすみません)。
修正 ↓入ってます。

初めにQCを行う。
3、サンプルの投入。
ランの手順はナノポア公式ページに動画で説明されている(購入者アカウントでログインして、以前は右上の検索ウィンドウから"Priming the SpotON Flow Cell Print step"で検索)説明動画(写真のキャプチャ)にリンクしていたが、2018年3月に確認したところ、HTMLマニュアルに切り替わっていた。
Minionのページからマニュアルのリンクに飛ぶことができる(Log inしている必要あり)。そこではランニング手順が記載されている。
3-1~

5-1



 この後もHTML manualは続いている。公式ではlambdaを使っているが、サンプルも同様の手順でランできる。
この後もHTML manualは続いている。公式ではlambdaを使っているが、サンプルも同様の手順でランできる。
You tubeに動画があったのでそれを載せておく。
ポートは弁の役割も果たしているので、動画の順番通りに進める必要がある。我々はををランを一回止めてサンプルを途中で追加投入する際に、priming portを開けてなかったためサンプルがうまくロードできなかった(吸い戻してなんとかリカバーした)。
Live base callを選択すると、リアルタイムで徐々にfast5からfastqに変換してくれる。ただしこれには時間がかかる様である。
ラン前。
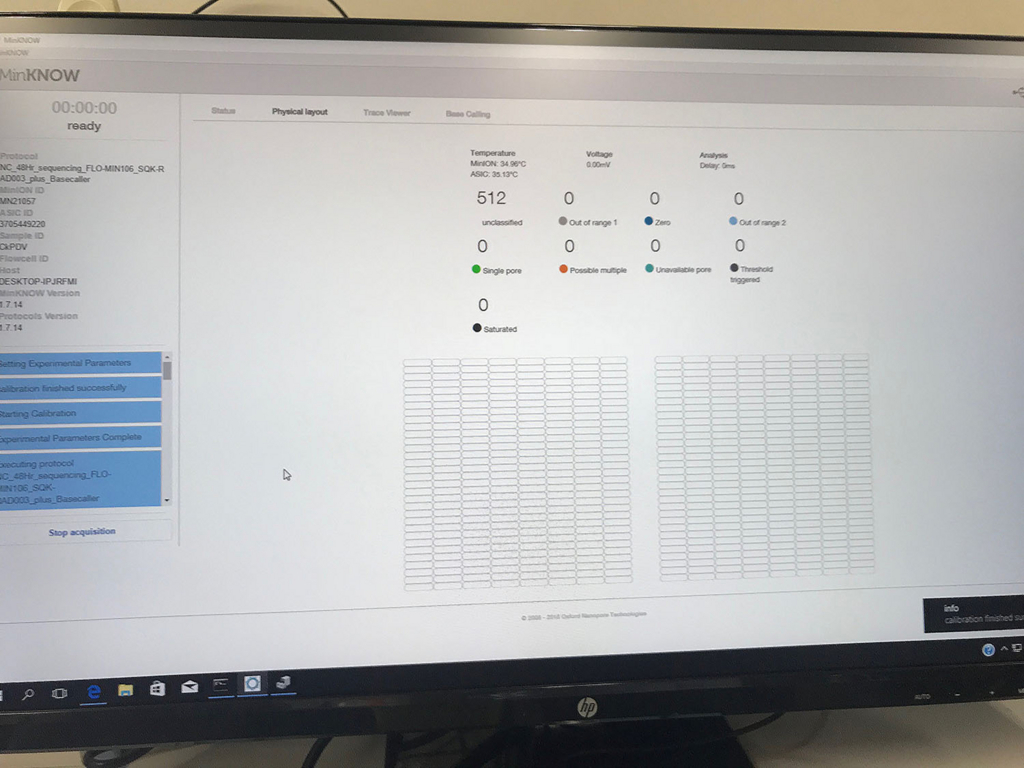
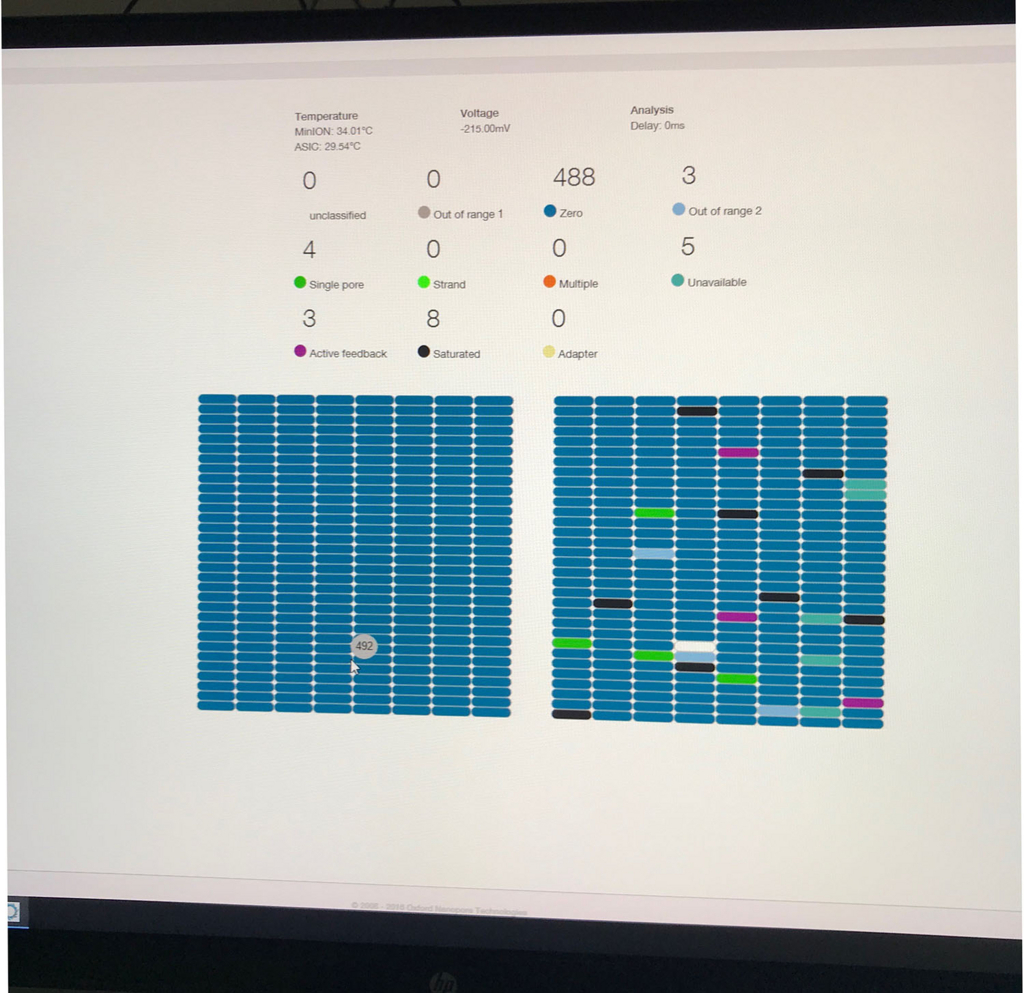
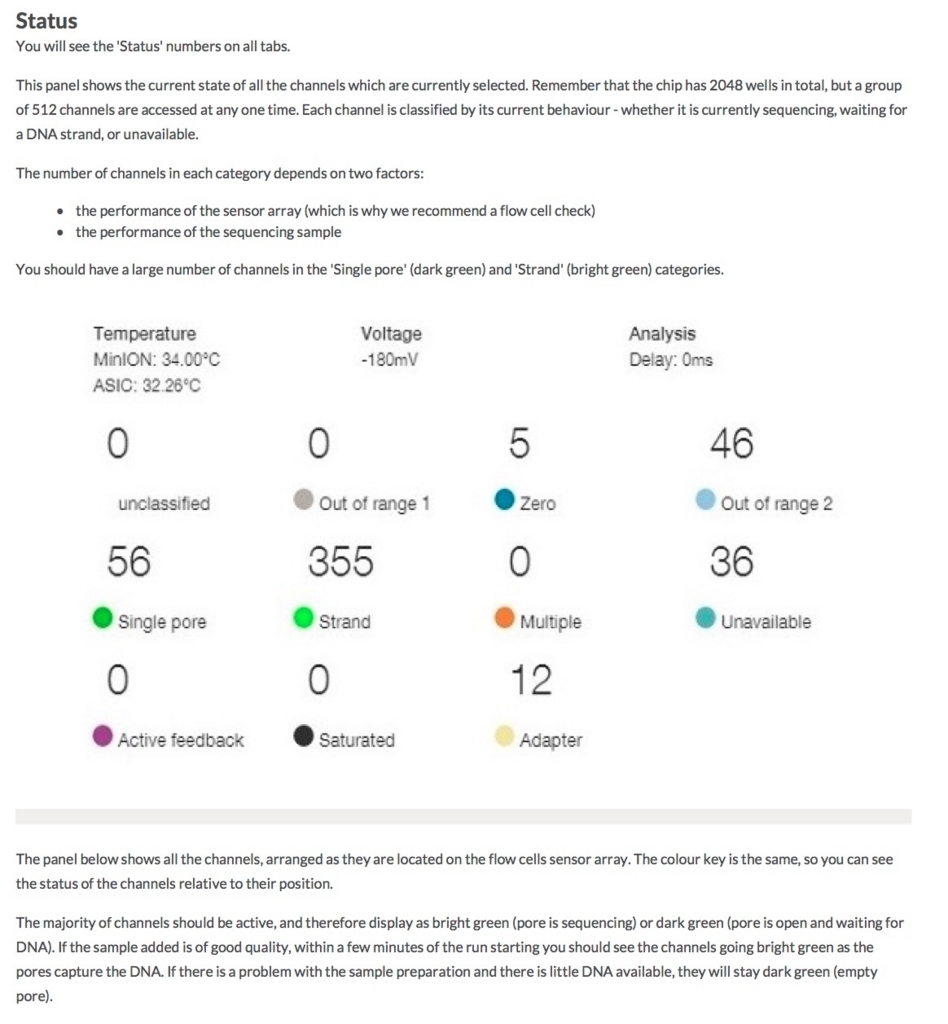
poreの活性を表示する画面(数時間後)。
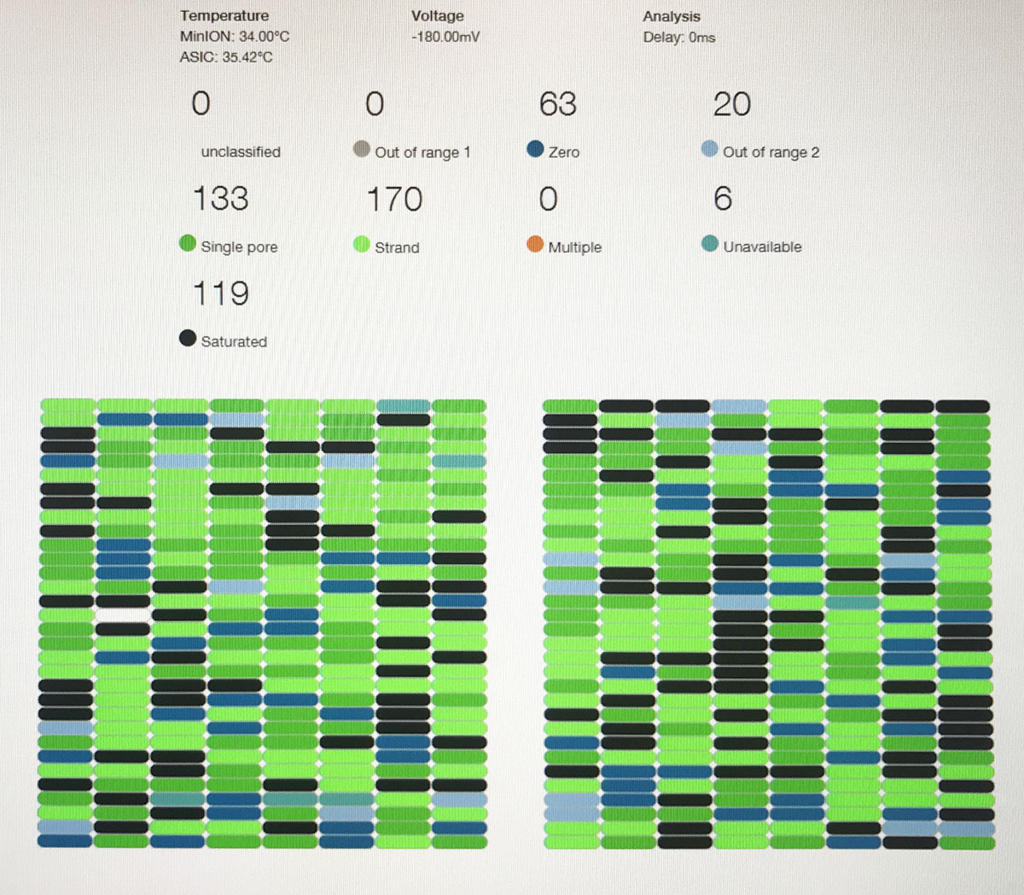
こちらはラン10分後のリード長分布。
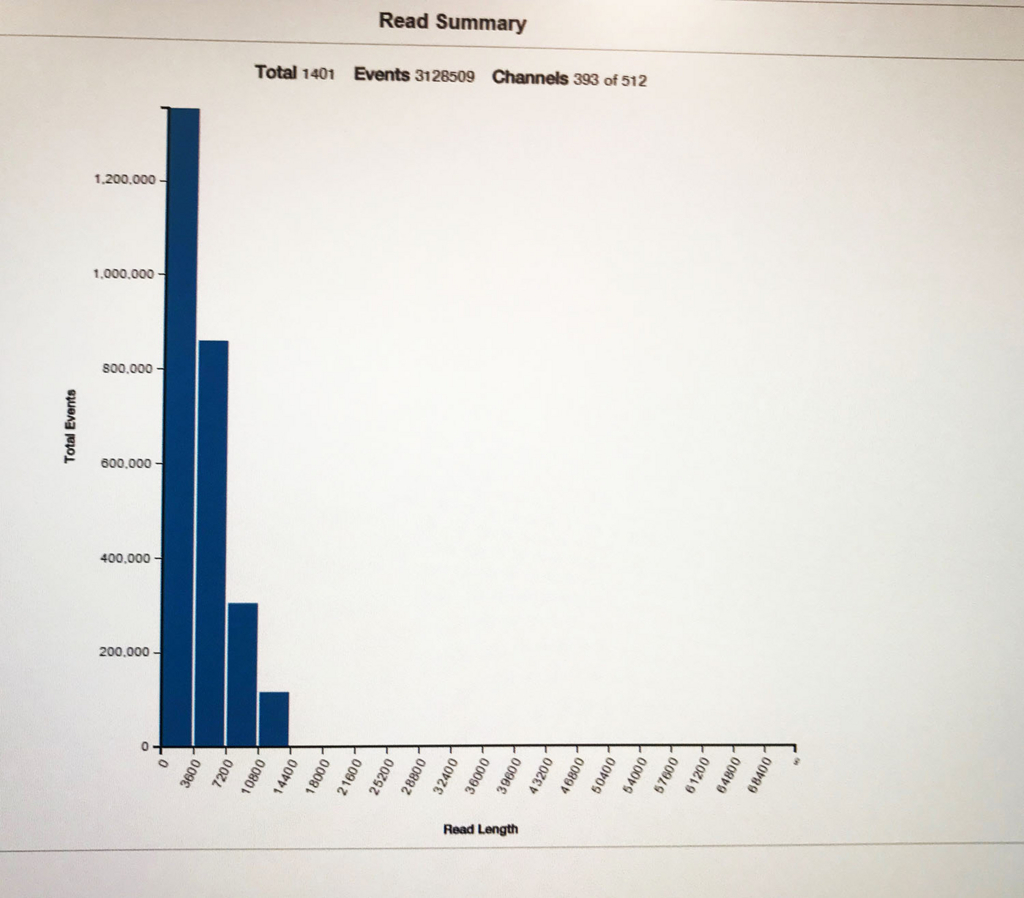
時間とともに、長いリードも増えてくる。
36時間ラン後。まだいくつか緑のセルがある。
48hランすると数万リード得られる。ただしサイズ分布によってリード数は当然変わってくるので、ライブラリによってリード数は大きく変わってくる。(ロングを1-2万読んだデータと、100bp以下のジャンク(10-20%エラーを含むショートリード)をたくさん含む10万リード(5倍)、どっちがアセンブルに有利だろうか)。
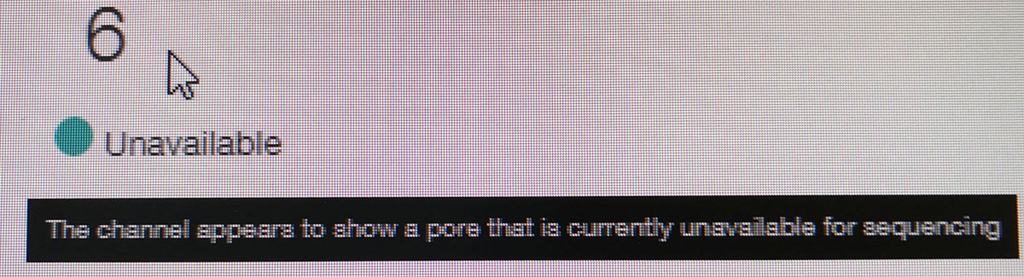
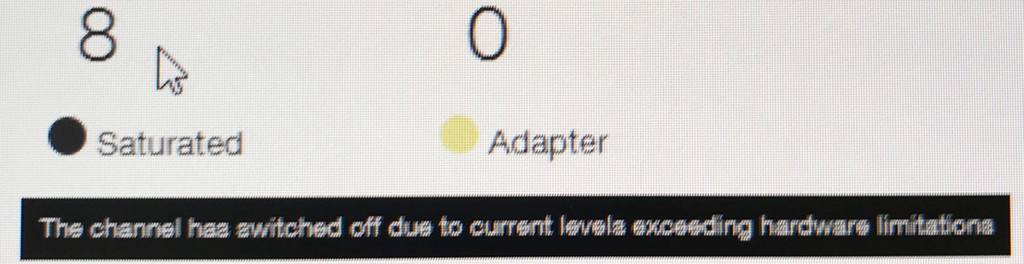
ポアの色は以下のように説明されている。
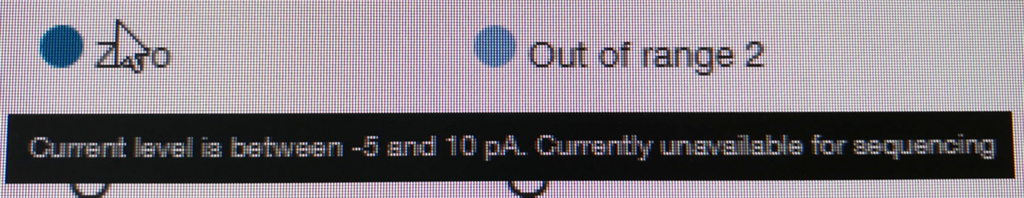
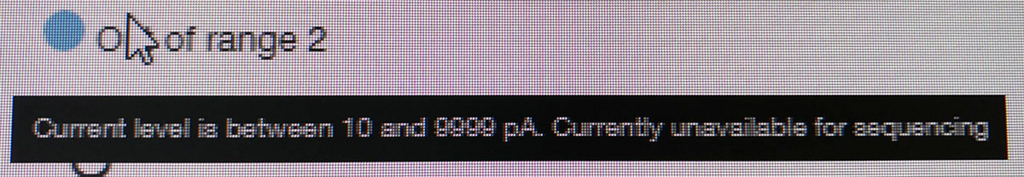
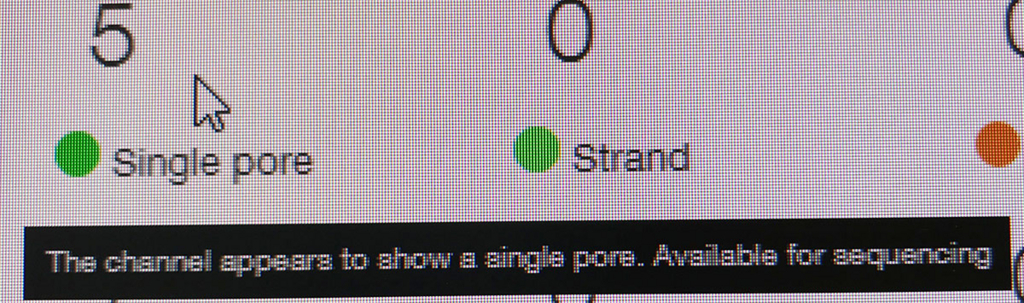
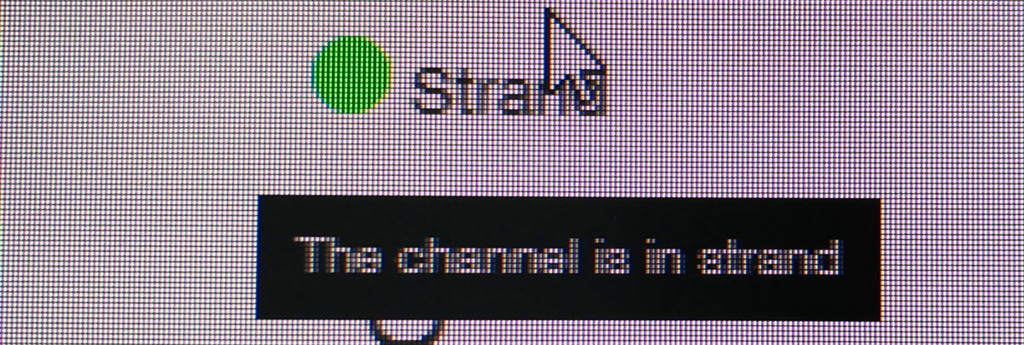
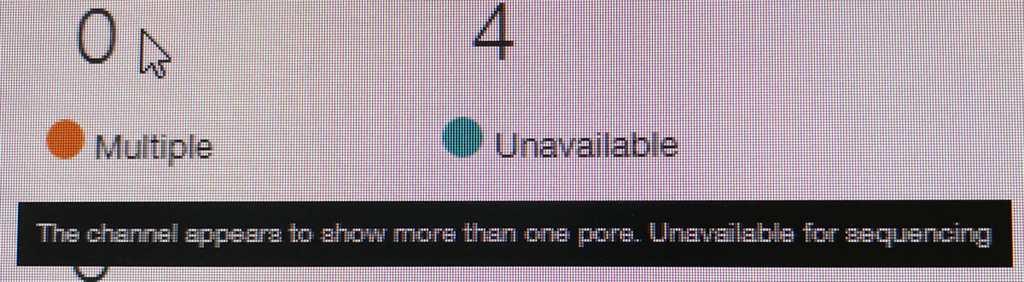
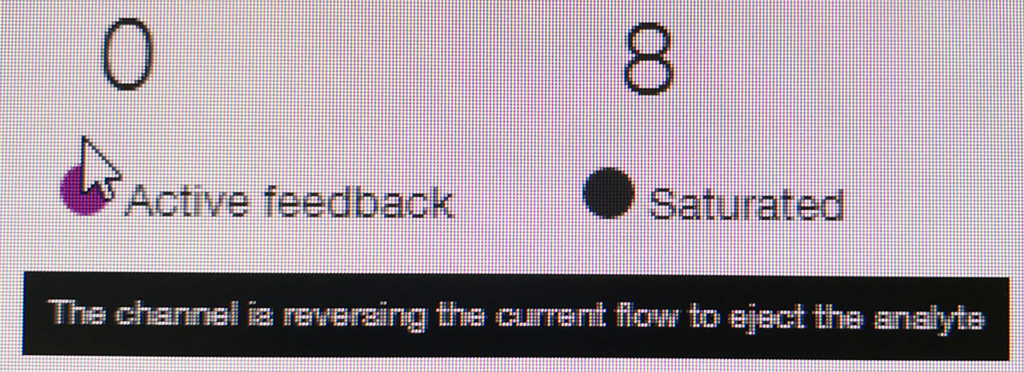
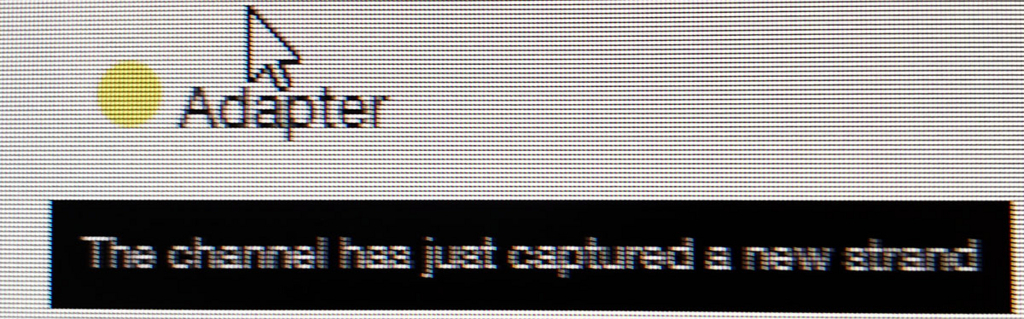
- ラン中のポアは、まずAdapterの黄色になり、それからしばらくすると緑のポアに変わっていく(時折だが一瞬色が変わることもある)。リードが取り込まれてアダプターから徐々にDNAが読まれているからと考えられる。
- 黒色は完全にダメになったポアである。また、オレンジも1つの区画に複数ポアがあるもので、信号が混在してしまい利用できないセルとなる。
- 青いセルも出てないセルである。ただし、途中でサンプルを追加投入すると青の区画も緑に戻るものがいくつかあった。完全にダメなセルではないらしい(未確認)。
- データは指定したディレクトリにどんどん追加されていく。他の方の情報によると、4000リードくらい読むと次のフォルダに切り替わるらしい。48hランすると、いくつかのフォルダができることになる。
公式HPの説明を貼っておく。
追記
Miriam Schalamunさん達のPreprinrtで、"ratio of ‘in strand’ (light green) to the sum of ‘in strand’ plus ‘single pores"を最初の数時間評価し、占有率が悪い時はwashすることを述べている(図8より)。
コマンドラインで動く basecallerもいくつか発表されています。albacoreとguppyはoxford nanoporeのオフィシャルbasecallerです。
ポアの活性やサンプル品質に大きく左右されるので一概には言えないが、読み取り速度は450bpsに調整されているらしい。ここでは2G出力されたと報告されている。また、ヒトゲノムをアセンブルしたという報告では39台のMinionを使い、トータル14,183,584 リード得て、トータル長は91,240,120,433 baseだったと述べられている(30x)。ということは、1台あたり平均363681リードで2.3Gb (23億)読んだことになる。データの大半はLigation kitで構築されたライブラリ由来らしい。本当なら素晴らしいスループットである。また、Nanopore R9 rapid run data release · Loman Labsでも1Dで2G、2Dで0.6G読めたと報告している。
ただし、http://seqanswers.com/forums/showthread.php?t=71774では0.5Gに達しなかったという話もある。SRAに登録されているデータを見ると、さらにばらつきが多い。この登録データでは7台のMinionを使っているようだが、スループットに大きな開きがある(バーコードシーケンスと書かれていないが、もしかしてバーコードシーケンス?)。
また日本でシーケンンスする場合、イギリスで作られて発送されるため、shippingで2週間くらいはどうしてもかかってしまう。その影響はどれくらいあるのだろうか?(早く届くこともあります。前回頼んだ時は1/24に注文して1/28にshipping、30に届きました)。
上記のリンクのLoman labでは、E.coliを1Dと2Dで読んだデータを公開してくれています。興味がある人はアクセスしてみてください。中盤のリンクから1Dと2Dのfast5データと、変換済みfastaがダウンロードできます (fast5は200~300GBくらいあります)。
Nanopore R9 rapid run data release · Loman Labs
追記
1、2018年3月に知り合いが行ったシーケンスでは、3.2Gbと6.1Gb読めていました(raw fastq)。
2、https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5803254/
この論文ではONT Ligation Sequencing Kitで調整したシロイヌナズナゲノムをMinionでシーケンスして3.4Gbの base callを得ています。200kb以上のリードも4本得られたと書かれています。miniasmなどでアセンブルするのが構造変化を検出するためにシンプルで最短の方法だと結論付けています。
3、上にもリンクを貼ったPreprint(リンク)では 、R9.5で安定して6-8Gb読めています。
4、複数のbacteria菌株のシーケンスとアセンブリパフォーマンス比較。MiseqとMinionのハイブリッドアセンブリを行なっている。bacteriaのロングリードシーケンシングでどのくらい読めばいいか参考になる。
https://www.biorxiv.org/content/biorxiv/early/2018/07/05/362673.full.pdf
まとめ
ナノポアのシーケンスに求められるのは"長さ"である。リード数が少なめでも、リピートをカバーできるだけのロングリードが読めていれば、その分だけアセンブルにとって有利になるし、ライブラリ中に1000bp以下の分子のコピー数が多く残っていれば、どれだけリードがたくさん読めていても、アセンブルのcontiguityの改善には限界がある。いかに切れていない綺麗なDNAを抽出してランできるかが結果を左右する。
参考資料
Sample preparation and DNA extraction in the field for nanopore sequencing · Loman Labs
http://seqanswers.com/forums/showthread.php?t=21280
http://seqanswers.com/forums/showthread.php?t=76021
2016年nanopore ワークショップに参加した研究者の方の記事
Omics! Omics!: Nanopore Workshop Notes
SSDの容量について(ログインが必要)
https://community.nanoporetech.com/posts/is-260gb-of-disk-space-eno
R9スループット
http://lab.loman.net/2016/07/30/nanopore-r9-data-release/
追記 2018 09/25
Oxford Nanopore Technologiesの宮本さんが、ブログにアセンブリとSV検出の流れをアップされています。
ナノポアシーケンサーMinIONインプレッション
ナノポアシーケンサーMinIONインプレッション | 酵母とシステムバイオロジー
追記 2018 10/06
MinIONとGridIONのフローセルが新しくなったようです。読み込み速度が強化され、耐久性も72hに伸び、アナウンスによると30Gbのyieldが期待できるようです。
Oxford Nanopore releases ‘Rev D’ flow cells, enabling increase in data yields
追記 2018 10/23
Nanoporeには様々なbasecallerがありますが、Ryan Wickさんがbasecallerを変えた時の違いを調べてまとめられています。
base call時点で大きな違いが出ていますが、nanopolishでmethylation-aware オプション付きでpolishingする限り、polish後の配列のエラー率に差はほぼなくなるようです。
*1
Nanopore公式ページで、枯草菌から複数のキットでDNA抽出してシーケンシングした時のフラグメントサイズ結果が掲載されている。使っているのは、QIAGEN Genomic-tip 500/G、QIAGEN MagAttract HMW DNA Kit、Circulomics Nanobind DNA Kit、QIAGEN Puregene Yeast/Bacteria Kit bになる。シンプルなワークフローで高分子DNAが十分に回収できるMagAttractか、とにかくより高分子DNAが取れるPuregene Yeast/Bacteria Kit(ただし収量が少ない)の使用が推奨されている。また、いずれの方法でも、シーケンシング前にSPRI技術を使ったサイズセレクトを行うことが推奨されている(参考)。(生物によっては、この方法だけで高分子gDNAが取れるとは限らないことに注意してください。細胞壁の破砕、エンドヌクレアーゼの不活性化、不純物によるカラム目詰まり、など想定しておくことはたくさんあり得ます)。
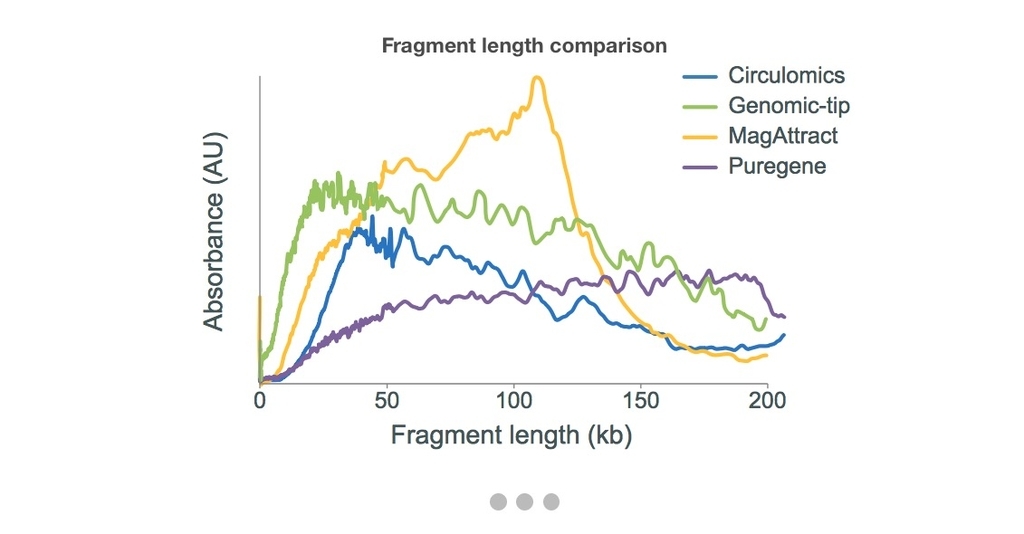
Nanopore公式ページより転載。やりすぎると非常に短いプラスミドも消える可能性があります。実験の目的に合わせて考えて下さい。
ロングリード関連のツール
2019 3/2追記
面白いものが出てますね。原理は不明ですが低分子DNAを除去できるようです。
https://community.nanoporetech.com/posts/circulomics-short-read-eli
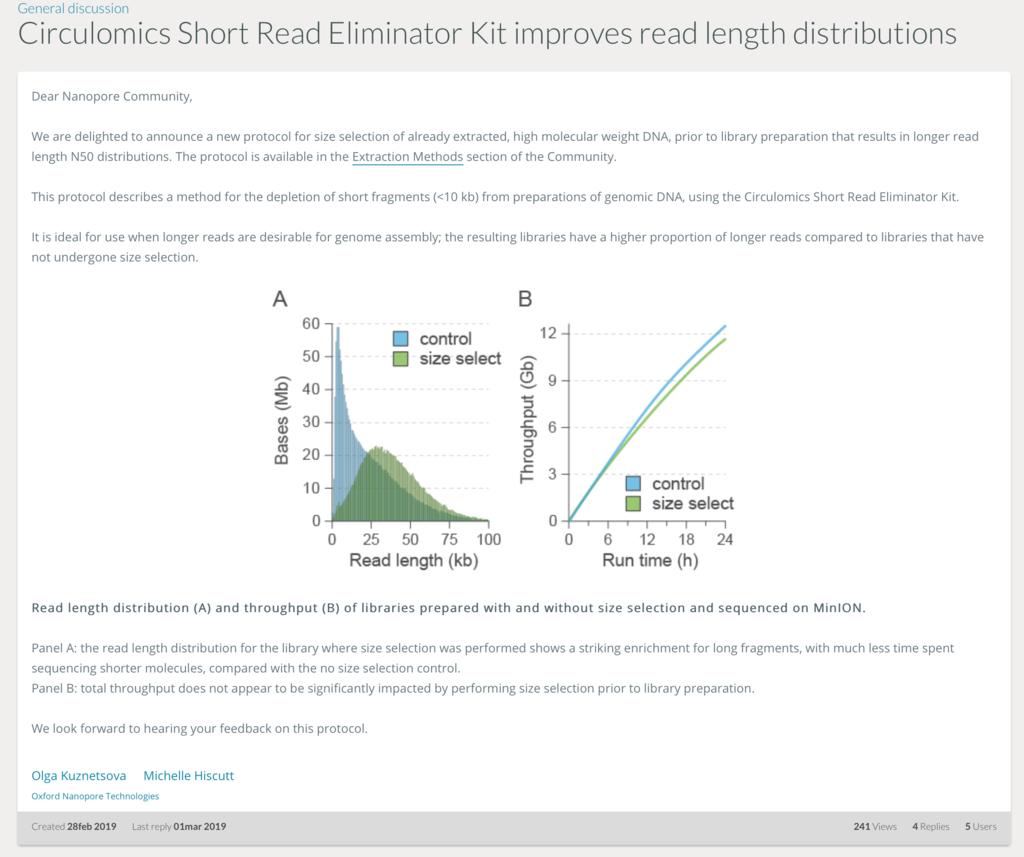
追記
この試薬に限らず当たり前の話ですが、処理することでかなりモル数は減ります。処理後ナノポアでランできるできるだけのDNAを想定して使わないと量が減って失敗します。ご注意ください。
2020 1/26 (植物)うまく使えば、かなりの高分子DNAを精製できるようです。
@boas_pucker talking about @nanopore sequencing:
— Franziska Reiher (@FranziskaReiher) 2020年1月26日
N50 >45kb (R9 flow cell)
protocol summary:
1) CTAB-based plant DNA extraction (+ß-ME)
2) @circulomics SRE purification
3) sequencing started 1h after loading the flow cell#Genomics #LongReadSequencing #Bioinformatics pic.twitter.com/yv6sjwPSbl
2019 3/10追記
日本ゲノム微生物学会 ニュースレターMinION Q&A(16ページ)
https://www.sgmj.org/pdf/newsletter/sgmj_no17.pdf
Stack Exchange --Bioinformatics--
Questions tagged [nanopore]
https://bioinformatics.stackexchange.com/questions/tagged/nanopore
2019 3/15追記
ついに出てきましたね。
Today the R10 nanopore has been released into an early access programme. Internal results show Q50 on a small genome, read more here: https://t.co/k8kdgcPACS pic.twitter.com/m6z4q263u2
— Oxford Nanopore (@nanopore) March 13, 2019
2019 3/18
Flongleが買えるようになりましたね、MinIONかGridIONのデバイスに取り付けて、MinIONより小スケール(126チャネルでMinION の512ポア相当)でランできます。値段は日本円でおよそ60万くらいで、48フローセル付いていて、1年間で小分け出荷できるようです(要確認)。1本あたり110米ドルくらいの計算で、16S/18Sアンプリコンやスモルーゲノムシーケンシングに適してそうですね。そのうちイールドの報告も出て来ると思いますのでtwitterチェックしておきましょう。
2019 3/22
Gordon Sanghera @gsanghea just opened #nanoporeconf reminding us why we are all here together. Please follow our tweets over the next few days from @nanoporeconf! pic.twitter.com/UGKDDUI7Nn
— Oxford Nanopore (@nanopore) May 22, 2019
2019 6/6
マーケティング上の理由なのかイールドが足りないのが理由はよくわかりませんが、Flongleが12パックでも購入できるようになってますね。
その後、1−2個から購入可能になっています。
知人がフロングルについて問い合わせた時の回答
Flongleのフローセルと、MinIONで使用するフローセルとの違い
フロングルは
・126チャネル(MinIONは512)
・フロングルランタイム:約16-24時間
・フロングルフローセルの前にCTCとアダプターが送付される
・フロングルフローセルは一回使用のみで返却の必要なし
・MinION/GridIONのみに使用可能
・9.4.1ポア(1D2キットとは使用不可)
・使用期限:4週間
・保存温度:通常のフローセルと同様
・推奨温度と使用期限を守り、使用前のQCポア数60以下だった場合交換対応
・ Flow cellで1.8Gb記録(22時間ラン)
・アダプターはのちにストアにて販売予定だが、現在必要な場合はフロングルのスターターパックを再度購入する。
7/5 Protocol.ioにmicroalga向けのDNA抽出プロトコルが上がってます。
2019 8/21
Nanopore Day, Tokyo に関するツイート
His some suggestions.. write a date to the box when you receive. Good point.. no date information our package. #nanoporeday pic.twitter.com/pKbMLVuQYu
— Mari Miyamoto (@marimiya_tky) August 21, 2019
MinKNOWトラブルシューティングです。 #nanoporeday pic.twitter.com/zrxn6UzUU8
— :marimiya (@marimiya_clc) August 21, 2019
Nanopore Day, Tokyoに行った人から聞いた話を2点追記。話半分で聞いてください。
- DNAが減ってきてDNAがない状態で電荷がかかり続けると、ポアが早く傷んでしまう。よって減ってきたら素早くウオッシュし、DNAを追加する。これを繰り返すことでシーケンスイールドを上げることができる(=48h放置は良くない)。
- 良くなっているが、R10は結構厳しい。dual channel?で読んでいるが、まだ発展途上。(当たり前だが)TGSの問題であるホモポリマーシーケンスエラーは解決していない。
#GenomeScience2019 - Twitter Search
Go sign up to our YouTube channel and hit the bell to subscribe: https://t.co/HnkozDJF49 - videos featuring the long read community with live QAs pic.twitter.com/cWmgDsDvMr
— Nick Loman (@pathogenomenick) September 3, 2019
Working on protocols to get longer reads using ligation prep: see @DrT1973’s bead free ligation protocol posted on https://t.co/5lWjp4enoN - aiming to improve long read yields
— Nick Loman (@pathogenomenick) September 3, 2019
Rocky Mountain adventures in Genomic DNA sample preparation, ligation protocol optimisation / simplification and Ultra long read generation
Rocky Mountain adventures in Genomic DNA | Long read club
A new pore coming as well... #genomescience2019 pic.twitter.com/Q5PCIEaSLA
— Matt Loose (@mattloose) September 3, 2019
Clive talking about 2D coming back.... @nanopore at genome science. pic.twitter.com/t9tYPQNniu
— Matt Loose (@mattloose) September 3, 2019
Fire Monkey HMW DNA extraction
15mins of Fire Flower treatment on a very challenging DNA input sample more than doubles average and N50. Long read numbers remain the same. 48hrs runs on LSK109/MinION @nanopore pic.twitter.com/b96L2jZu9M
— Georgios Patsos (@GeorgiosPatsos) September 9, 2019
Improved Long Shot (LSv3/LSK109) protocol on Fire Monkey extract is generating a long tail on MIN106D @nanopore. MinKNOW estimates top read of 575kb after 1hr of sequencing! pic.twitter.com/mjPBQcNAYt
— Georgios Patsos (@GeorgiosPatsos) July 15, 2019
Bacteria to DNA in less than 1 hr! No post-extraction size selection required due to optimal molecular ratio of long to short DNA fragments. At Q11 N50~50Kb, Yield 5+Gb, and 2,000+ reads above 100Kb using a MIN106D flow cell LSK109 @nanopore . (Predicted coverage at Q7 >3700x) pic.twitter.com/osctzsHEub
— Georgios Patsos (@GeorgiosPatsos) June 13, 2019
Thank you! We are currently covering mammalian cells (see previous tweet) and bacteria. Also we offer Fire Flower which is a spin-column size selection method for already extracted DNA, for more details: https://t.co/aZps9rhuaM and georgios@revolugen.co.uk
— Georgios Patsos (@GeorgiosPatsos) June 14, 2019
https://revolugen.co.uk/revolugens-dna-extraction-technologies/
良さそうですね。
有償のワークショップもあります。
2019 12/6追記
CB: We are now introducing R10.3 - our full release version. It optimises single molecule accuracy as well as uplift in consensus accuracy improvements - done through better signal:noise. This is the 'fixed up' R10, starting in a good place and wil be optimised #nanoporeconf pic.twitter.com/Od48sUAsqn
— Oxford Nanopore Events (@NanoporeConf) 2019年12月5日
In development R11 @nanopore Twin pair with R10. Complementary 2 pore system #nanoporeconf pic.twitter.com/7HizYcGD0d
— Alexander Wittenberg (@AW_NGS) 2019年12月5日
2020 1/14
R10.3
Today we’ve introduced R10.3 flow cells to the store. Building on R10 — which was recently shown to produce 99.995% single molecule consensus accuracy — R10.3 provides increased throughput and better raw read accuracy. Read more: https://t.co/HCGcPptPjB pic.twitter.com/8KlCp65ClO
— Oxford Nanopore (@nanopore) 2020年1月13日
2/22
Officially releasing my protocol to generate ultra-long ONT reads with an N50 >150 kb! The protocol was used to produce the reads shown below (2.12 Gb of data with an N50 ~193 kb) and can be accessed here: https://t.co/5I4GERa1Xw @longreadclub pic.twitter.com/Z03YKeUpOp
— Glennis Logsdon (@glennis_logsdon) 2020年2月21日
3/15
"We provide a high-resolution map of SARS-CoV-2 transcriptome and 22 epitranscriptome using nanopore direct RNA sequencing and DNA 23 nanoball sequencing."
The architecture of SARS-CoV-2 transcriptome https://t.co/8NMqvIRdht #bioRxiv
— bioRxiv (@biorxivpreprint) 2020年3月14日
4/5
Here is the in progress github with how to make custom primers work with RAMPART and artic network protocols. (both nanopolish and medaka). I'll write up some of this in more detail soon.#coronavirus@NetworkArtic@nanoporehttps://t.co/iClnRxJNqS
— James Ferguson (@Psy_Fer_) 2020年4月3日
5/9
6/19
Accuracy of @nanopore sequencing is complex – are you interested in raw, consensus, variant calling, methylation or test accuracy? We’ve summarised the latest performance and the tools needed to achieve this at this page https://t.co/7oCW2pzQak #nanoporeconf
— Oxford Nanopore (@nanopore) 2020年6月18日
7/26
Brassica napusゲノムアセンブルの査読前論文。"6X with reads longer than 100Kb"などのウルトラロングシークエンシングをしている。テクニック全般において非常に勉強になる。
https://www.biorxiv.org/content/10.1101/2020.07.22.215749v1
(2020年12月にpublish) https://academic.oup.com/gigascience/article/9/12/giaa137/6034787)
10/21
精度がほぼQ20に達する。
Big accuracy update coming in the next version of Bonito 🚀 v0.3.0 combines everything we have learned with structured and unstructured approaches - @dcpage3, Tim and myself are working hard on the finished touches this week - watch this space 👀 https://t.co/QulhwCuPB9
— Chris Seymour (@iiSeymour) 2020年10月20日
12/7
SR: This week Oxford Nanopore has generated modal *raw-read accuracy* of 99.1% (99%=Q20) using a new chemistry with Bonito, delivered on internal validation sets with a substantial fraction of these raw reads above Q20 #nanoporeconf pic.twitter.com/bH3JjdVZxP
— Oxford Nanopore (@nanopore) 2020年12月2日
12/26
ロングリードシークエンサーのためのゲノムDNAの調製法を公開しました。
— Y. Hirose (@YuuHirose1983) 2020年12月25日
微生物ならなんでもいける…はず!!https://t.co/itWbV5Rp0G
2021
1/21
Ultra long, high throughput, and high accuracy? #nanobind ultra long working beautifully on R10.3 on @nanopore PromethION. 90 Gb at 83 kb N50. Great way to start the year off! @kotturi pic.twitter.com/I6L58KRFuL
— Circulomics (@circulomics) 2021年1月19日
2/2
精度がQ20を超える
Bonito v0.3.5 live -
— Chris Seymour (@iiSeymour) 2021年2月1日
- new dna_r10.3_q20ea model
- performance improvements (upto 35%) from @qsirelkhatim @EpiSlim
- interupting basecalling with ctrl+c will now exit gracefully
- ability to finetune a pretrained model on new data
https://t.co/DYdir9VLni pic.twitter.com/R8vgaiJ2EG
植物の組織サンプルから開始し、7日以内に高品質のドラフトゲノムを作成するための完全なデノボ植物ゲノム作成ワークフローの設計 (ONT & Pacbio)。
LeafGo: Leaf to Genome, a quick workflow to produce high-quality De novo genomes with Third Generation Sequencing technology https://t.co/2kbmLrSLW0 #biorxiv_genomic
— bioRxiv Genomics (@biorxiv_genomic) 2021年1月26日
ロングリードライブラリ調製に使用される試薬のゆっくりとしたピペッティングを自動化する3Dプリント可能な装置。手動のスローピペッティングに比べてライブラリーの平均リード長を増加させた。
Consistent ultra-long DNA sequencing with automated slow pipetting https://t.co/nihqfZ0411 #biorxiv_genomic
— bioRxiv Genomics (@biorxiv_genomic) 2020年9月20日
MinIONを使用したSARS-CoV-2ゲノムのRNA抽出からハイスループットシークエンシング、及びオンライン可視化ツールを使用した解析までの包括的な作業プロトコル
High Throughput Nanopore Sequencing of SARS-CoV-2 Viral Genomes from Patient Samples https://t.co/17dKZ25WGM #bioRxiv
— bioRxiv (@biorxivpreprint) February 10, 2021
2/23
小さなプラスミド配列を回収する能力を評価する目的で、ライゲーションライブラリとラピッドライブラリのアプローチを比較している。
Figure 1 gives a simplified illustration of why we think this is the case: due to their size, small circular plasmids can avoid fragmentation during DNA extraction, leaving no ends for adapter ligation. Rapid preps, in contrast, don't depend on DNA ends.
— Ryan Wick (@rrwick) 2021年2月22日
(3/11) pic.twitter.com/Kkp8qPlLmQ
Recovery of small plasmid sequences via Oxford Nanopore sequencing | bioRxiv
3/1
A new "Q20+" @nanopore sequencing kit is now being tested by a number of developers in the Community. This has produced raw single-molecule, single-pass sequencing reads above a modal 99% accuracy, or Q20+ internally and externally. Read more: https://t.co/hd5FSTNEg9 [1/2] pic.twitter.com/zWQC7Hmub2
— Oxford Nanopore (@nanopore) 2021年3月1日
3/4
ロングリードメタゲノミクスのための高分子量DNA抽出法を比較した。メタゲノミクスの観点からは、非常に長いシーケンスよりも中程度の長さのシーケンス(10〜50kbp)がたくさんある方が興味深い。それはアセンブリメトリクスを改善し、より多くのシングルコピーコア遺伝子とrRNA遺伝子で分類学的プロファイルにも影響を与える。
My first pre-print as a postdoc in the @Meren lab!
— Florian Trigodet (@FlorianTrigodet) 2021年3月4日
We compared high molecular weight DNA extraction methods for long-read metagenomics using @nanopore and found out that the best extraction method is not the one yielding the longest DNA fragmentshttps://t.co/eh9azbWgwc
2022 1/25 Mol Ecol Resour. 2022 Jan 23. doi: 10.1111/1755-0998.13588 (pubmed)
3/6
This @GoogleHealth @ucscgenomics work led by @kishwarshafin @benedictpaten describes methods to call variants in @nanopore data, matching Illumina SNP accuracy, phase @nanopore @pacbio data, and polish ONT assemblies. Kishwar described the work at: https://t.co/VMs2pZI4Ow 1/n https://t.co/dbefQZgHFR
— Andrew Carroll (@acarroll_ATG) 2021年3月5日
3/25
Ultra-long @nanopore sequencing kit just released in store. Looking forward to combine this with @KeyGeneInfo ultra HMW DNA isolation for crops to obtain loooooong reads. https://t.co/KIQ5TMKlCU pic.twitter.com/jovqCg2uZx
— Alexander Wittenberg (@AW_NGS) 2021年3月24日
4/23
The preprint from the Singapore Nanopore Expression Project (SG-NEx) is online! The SG-NEx data is a systematic resource for @nanopore long read RNA-Sequencing, including direct RNA, cDNA, and PCR cDNA with matched short read data and spike-in RNAs. 1/n https://t.co/HlY6CB50NY
— Jonathan Göke (@JonathanGoeke) 2021年4月23日
5/8
DNA smoothly loaded on the flow cell 😎. @nanopore pic.twitter.com/RjSHmTQE3M
— Tiphaine Le Roy (@Titicaccae) 2021年5月7日
5/17
Why does this #nanobind ultra long @nanopore read length distribution look so jagged? It's a 12-plex ultra long bacteria run with full length plasmid reads! Imagine 50X full length coverage of 200 kb plasmids. Learn more @NanoporeConf this week! pic.twitter.com/ZeSm7fDLhw
— Circulomics (@circulomics) 2021年5月17日
5/20
Oxford Nanopore Eventsいくつかツイートを貼っておきます。
Guppy 5 is now available with Bonito CRF style models #nanoporeconf
— Chris Seymour (@iiSeymour) 2021年5月20日
- Fast 🚀
- Accurate 📈
- Calibrated Qscores ⁉️https://t.co/zT5nsj4P6g
SR: In summary on accuracy #nanoporeconf pic.twitter.com/JMVS3uRPy0
— Oxford Nanopore Events (@NanoporeConf) 2021年5月20日
RSD: Q20+ early access coming soon, please register your interest here: https://t.co/XMcei5a7D6 #nanoporeconf
— Oxford Nanopore Events (@NanoporeConf) 2021年5月20日
Q30 accuracy using Duplex @nanopore reads. Just two copies! 🦨 Great work! #nanoporeconf pic.twitter.com/5RhLmD7u8G
— Alexander Wittenberg (@AW_NGS) 2021年5月20日
Now @RosemaryDokos on the details of the products. Transparent Pricing at the different flowcell levels #NanoporeConf pic.twitter.com/WeP5sIA3g4
— Albert Vilella (@AlbertVilella) 2021年5月20日
JC: Over the last two years we’ve driven improvements to deliver a real jump in raw read accuracy — from around 92 to >98%. These improvements have come from algorithm and chemistry improvements #nanoporeconf pic.twitter.com/cWu9UThDFW
— Oxford Nanopore Events (@NanoporeConf) 2021年5月20日
JC: Theoretical output from a MinION-sized piece of silicon would be 3.9Tb per day from a single flow cell. More realistically 60% pores, 60% utilisation would still be 58Gb per hour, or a human genome in under an hour #nanoporeconf
— Oxford Nanopore Events (@NanoporeConf) 2021年5月20日
6/2
ヒトGRCh38やシロイヌナズナTAIR10参照ゲノムのように、最も精査されたゲノムでさえ、生物学的に重要なセントロメアやその他の大きな繰り返し領域を表現できていない。このプレプリントでは、ウルトラロングリードシーケンシングを用いて、5つのセントロメアをすべて解明したCol-CENシロイヌナズナゲノムアセンブリを構築した。
Pleased to announce our new preprint! We used @nanopore to assemble the complete Arabidopsis Col-0 centromeres. Then we analyzed data from mutant lines to learn about methylation/DSB patterns. A fun and interesting collaboration with @hendersi. https://t.co/KZqwGhULnU
— Michael Alonge (@malonge11) 2021年6月1日
6/15
Rapid Sequencing Kit で普通にやるとリード長N50 15 kb くらいにしかならんけど、最初のフラグメンテーションの後にShort Read Eliminator を掛ければ解決するんちゃうかとずっと思っていたのをついに試してやった。 pic.twitter.com/1dmkAt4t78
— 内藤健 (@drk0311) 2021年6月15日
7/1
Want to see more videos on how to turn stringy UHMW DNA into ultra long @nanopore reads? Register for tomorrow's webinar https://t.co/Hf0obDonB8 @mitenjain @rgrubinstein pic.twitter.com/Vu5yLlecSF
— Circulomics (@circulomics) 2021年6月30日
7/15
GBE latest: Ultralong Oxford Nanopore reads enable the development of a reference-grade perennial ryegrass genome assembly https://t.co/uFTrxaPGHw
— OfficialSMBE (@OfficialSMBE) 2021年7月11日
7/17
純粋な高分子量DNAの抽出には限界があり、植物や菌類では特に困難である。この問題を解決するために、本研究では、ロングリードシーケンシングのための高分子量DNAの抽出、クリーンアップ、サイズ選択のプロトコルを紹介する。紹介するプロトコルを用いて、オックスフォード・ナノポア社のMinIONでシーケンスを行うと、リード長N50値が30-50kb、リード長が200kbを超え、出力が15-30Gbpになる。これは、様々な植物、真菌、動物、バクテリアのサンプルでルーティンに達成された。
The 2nd Lab Protocol paper was published yesterday! "High-molecular weight DNA extraction, clean-up and size selection for long-read sequencing" from @dnawhisperer & colleagues https://t.co/PBYwJVno4l.
— Lenny Teytelman (@lteytelman) 2021年7月16日
(Part of the @protocolsIO-@PLOSONE partnership) 1/3
Benchmarking ultra-high molecular weight DNA preservation methods for long-read and long-range sequencing https://t.co/v1mBZR7SXG #bioRxiv
— bioRxiv (@biorxivpreprint) 2021年7月14日
ONTによるゲノム配列の決定とアセンブルには、ロバストな実験デザインが必要だが、真核生物を対象とした研究はほとんどない。いくつかのモデル生物のシーケンスおよびアセンブリのベストプラクティスを特定するために、シミュレーションおよび経験的なONTとDNAライブラリを使用した新しい結果を紹介する。
9/8
バナナゲノムのT2Tアセンブリ(バージョン4アセンブリ)の報告。ONTのリードも使用されていて、いくつかのアセンブラを検討した結果、NECATが一次アセンブリに使用されています(Supplementary Table.8に比較結果)。結果を見ると、リピートはかなり存在していますが、DH Pahangというホモ接合2倍体が使用されていて、これがアセンブリが奏功しているポイントの1つになっているのでしょうか。
9/21
「実験医学別冊 最強のステップUPシリーズ ロングリードWET&DRY解析ガイド シークエンスをもっと自由に!」の書籍届いた!手に取ってみれるとやはり嬉しいですね。あらためて執筆陣のみなさま、羊土社のみなさまに感謝!https://t.co/JsMJPh7704 pic.twitter.com/nEkI0jze78
— Kazuharu Arakawa (@gaou_ak) 2021年9月21日
10/19
低入力量での抽出およびライブラリ調製アプローチを評価する実験を考案した。従来のスピンカラムを用いたDNA抽出ではなく、磁気ビーズを用いた最適なビーズビート法を用いることで、分子の長さ、インテグリティスコア、DNA収量のいずれも向上することが分かった。考案した迅速な抽出プロトコルと低入力量のライブラリ構築により、単一の昆虫(Drosophila melanogaster)から、125Mbp / 参照ゲノムの85 %、BUSCO遺伝子の96.9%以上を網羅し、コンティグN50が1.2Mbp以上で、染色体アームサイズのコンティグを含むデノボアセンブリを、600GBP以下の安価な消耗品コストで生成することができた。
How low can you go? Driving down the DNA input requirements for nanopore sequencing https://t.co/PAjPYMabml #bioRxiv
— bioRxiv (@biorxivpreprint) October 18, 2021
Adaptive sampling
As DNA passes through a nanopore, its sequence is decoded in real time. Using adaptive sampling, it's possible to reject sequencing reads that are not of interest — leaving the nanopore available for your targeted region of interest: https://t.co/CPH1YYxukk #TechTuesday pic.twitter.com/vD1NqYu022
— Oxford Nanopore (@nanopore) October 19, 2021
11/11
We are there now @kirk3gaard! Check out our updated pre-print @nanopore R10.4 long-read data enables near-perfect bacterial genomes from pure cultures and metagenomes without short-read or reference polishing 1/12 https://t.co/OUxFSd8sbk
— Mads Albertsen (@MadsAlbertsen85) 2021年11月11日
Some key take-aways. The modal raw read accuracy for @nanopore R10.4 is approx., 99% 2/12 pic.twitter.com/2jujMeGU99
— Mads Albertsen (@MadsAlbertsen85) 2021年11月11日
R10.4 is still early days, and will improve further in the future. We encourage everyone to download the raw data from ENA and give it a spin! We are blown away that it now seems possible to obtain near-perfect bacterial genomes using @nanopore alone! 12/12
— Mads Albertsen (@MadsAlbertsen85) 2021年11月11日
12/4
The rapid @nanopore improvements means that I had to update my slides from last years. R10.3 was roughly were R9.4.1 is today and well R10.4 simplex is now ~99% and for duplex things start to get really crazy. A few more iterations and people will stop talking about accuracy? pic.twitter.com/ewr9HdM1Gl
— Rasmus Kirkegaard (@kirk3gaard) December 2, 2021
Just running some more 🌽 duplex cells. Below some hot of the press (not whole dataset) results of R10.4/LSK112. 99.9% median accuracy. Not only @PacBio can generate HiFi reads. #nanoporeconf #maize #accuracy pic.twitter.com/Eb4sfG5RQF
— Alexander Wittenberg (@AW_NGS) December 2, 2021
All our raw data is available online on @nanopore R10.4 (zymo + complex metagenome). Also includes Illumina, R9.4.1, R10.3 and PacBio CCS - see links in here: https://t.co/OUxFSd8sbk
— Mads Albertsen (@MadsAlbertsen85) December 3, 2021
注;duplexリードは、は1分子DNAのtemplate鎖とcomplement鎖を連続してシークエンシングして得られる新方式のリード。上のツイートの通り、条件が揃えば非常に高い精度の配列決定結果を得られるらしい。2021年5月の発表)
12/31
ナノポアセンサーアレイ技術(Oxford Nanopore Technologies社のMinIONデバイス)を用いて、バーコード付きの出力鎖を高並列に直接検出することにより、DSD回路の活性をリアルタイムで動態測定できる多重化シーケンスフリー読み出し方法を提案する。ディープラーニングを用いて、生のナノポア信号から直接、人工レポータープローブを1分子レベルで高精度に検出・分類できることを示す。次に、臨床的に関連するマイクロRNA配列の多重検出において、この方法の有用性を実証する。これらの結果は、DSDの出力帯域幅を蛍光分光法よりも1桁増加させ、DNA回路の読み出しや携帯型ナノポア装置を用いたプログラム可能な多重分子診断における新しいパラダイムの基礎を築くものである。
Year end buzzer-beater preprint! We present a new readout method for DNA strand displacement reactions using @nanopore arrays: A nanopore interface for high bandwidth DNA computing https://t.co/b6gRslEMCJ
— Jeff Nivala (@jeffnivala) 2021年12月31日
2022
1/3
Kit 12 & R10.4 flow cells are now available in Store, enabling >Q20 raw read accuracy or around Q30 Duplex accuracy, for high-accuracy consensus sequencing & variant calling. Find out more: https://t.co/AemezQNyMO pic.twitter.com/lDOmTCxQSf
— Oxford Nanopore (@nanopore) 2022年1月3日
1/4
SLOW5 is now published!
— Hasindu Gamaarachchi (@Hasindu2008) January 4, 2022
~performance
~space savings
~accessibilty
Thank you all for testing and feedback.
Fun exercise: Anyone with a set of FAST5 with relatively short reads (e.g., SARS-CoV-2 amplicons, CDNA), convert to SLOW5 using slow5tools and observe the file size! https://t.co/jb9tcF27FE
1/12
Just one @nanopore R10.4 MinION cell of A. thaliana Col-0 generated 14Gb of data and an assembly more contiguous and accurate compared to public @tair_news v10.1 release. Blog post: https://t.co/CHLncE6keg #PAGXXIX #NGS #accuracy pic.twitter.com/5AgpYnaebW
— Alexander Wittenberg (@AW_NGS) January 12, 2022
Excited to share some new work from us today on ultra-rapid genome sequencing for the diagnosis of critically ill children and adults.
— euan ashley (@euanashley) January 12, 2022
In critical care settings, genetic diagnosis can shorten stays, direct precision therapy, save money, and help with family planning. 1/ pic.twitter.com/Wq0tGRd5m0
1/13
Happy to share our work on repeat-calling errors in nanopore sequencing of telomeres with @lh3lh3 and #matthewmeyersonhttps://t.co/DHbKy8DsZj
— KT8 (@kartong88) January 13, 2022
1/20
一緒にTBAを主催している久野先生の論文がPLOS Biologyから出版
— Haruka Ozaki (尾崎遼) (@yuifu) 2022年1月20日
ゲノム編集の奏功を、ナノポアによるターゲットシーケンシングと深層学習ソフトウェア(DAJIN)で識別・分類する手法🧬https://t.co/55u1XaROSOhttps://t.co/46uJHjqoNR
当研究室大学院生の鈴木さんもお手伝いしました
1/22
Nanopore sequencing has improved dramatically.
— Daniel Giguere 🧬 (@DanielJGiguere) January 20, 2022
Here are 3 takeaways from the newest developments 👇🧵
1/25
”存在量はべき乗則に従うことが多く[7]、このことは、メタゲノムサンプルの配列決定によって、ある種の生物種を深くカバーし、他の種のカバー率が低いか部分的になるデータが得られることを意味することがある。希少な種のシークエンスの出力を最大化する効果的なエンリッチメント戦略は、この弱点と生物多様性の盲点に対処するものである。オックスフォード・ナノポア・テクノロジーズ(ONT)のAdaptive Sampling Concept(選択的シーケンスと呼ばれることもある)は、ソフトウェア制御によるエンリッチメントの一形態と言える。分子の最初の数百塩基を調べ、その分子が「ターゲットの分子」であるかどうかを判断し、ターゲットから外れた分子は、孔に流れる電流を逆流させて排出し、孔を解放して新しい分子を捕らえる。
我々は、DNA分子の長さがAdaptive Samplingの効率と効果に及ぼす影響を調査し、MAGと診断の両方のアプリケーションにおける有用性を判断したいと考えた。ここでは、既知の相対的存在量とリード長分布が与えられたメタゲノム・コミュニティで可能なエンリッチメントレベルを予測する数学的モデルを提示する。”
[Self promotion warning] Very happy to see our @nanopore adaptive sampling metagenomics paper come out today in @GenomeBiology - particular credit to Sam Martin and Darren Heavens, who somehow get through life without Twitter. @NanoporePapers https://t.co/xNFkyswkRs
— Richard Leggett (@richardmleggett) January 24, 2022
1/28
"当研究室では、ゲノム配列決定パイプラインの自動化を図るため、ロボット生物学研究所の両腕型人間型ロボット「Maholo LabDroid」に、細胞溶解液からの有機溶媒によるゲノムDNA抽出を行うようプログラミングを行った。フェノール・クロロホルム抽出の自動化については、我々の知る限り、今回が初めての報告である。"
Automated phenol-chloroform extraction of high molecular weight genomic DNA for use in long-read single-molecule sequencing. https://t.co/g7L88eIjgJ #bioRxiv
— bioRxiv (@biorxivpreprint) 2022年1月28日
(DOI: doi: https://doi.org/10.1101/2022.01.26.477939のフル動画リンク)
"In order to automate the genome sequencing pipeline in our laboratory, we programmed a dual arm anthropomorphic robot.." https://t.co/Yy10Qu6XwL pic.twitter.com/IN8K08Fcsx
— Theo Sanderson (@theosanderson) 2022年1月28日
(* リンクされているプレプリントの著者ではない)
A friendly reminder on what measuring DNA by NanoDrop (a.k.a. random number generator) can actually tell you👇
— Daniel Giguere 🧬 (@DanielJGiguere) February 15, 2022
3/4
”これは、基本的にベースコールモデル(fast, hac, sup)と chunks_per_runner の値のリストを取り、それらを繰り返し実行する非常に初期のドラフトコードです。Guppy実行の詳細(ベースコール率、サンプル/秒を含む)を記録し、またGPU使用統計も記録してます。時間が許す限り、より多くの機能を追加したいのですが、これは現在の形で一部の人に役立つかもしれないと思いました。”
It's been awhile since I put some documentation together, so I decide to spend some of my time being sick channelling something useful.
— Miles Benton (@miles_benton) February 11, 2022
So here is a rough and ready script that aims to help quickly optimise @nanopore Guppy basecalling on GPUs: https://t.co/A4HSms5vRg
1/n
"第3世代のロングリードシーケンスが植物ゲノム科学に変革をもたらす。Oxford Nanopore TechnologiesとPacific Biosciencesは競合するロングリードシーケンス技術を提供し、植物科学者が大規模で複雑な植物ゲノムでさえ調査できるようにする。シーケンシングプロジェクトは1つの研究グループで実施することができ、小規模な植物ゲノムのシーケンシングは数日で完了できる。また、陸上植物の起源と進化に関連する基本的な疑問を解決するために、複数の種のゲノムを大規模に調査することが多くなった。配列決定装置や使いやすいソフトウェアが入手しやすくなったことで、より多くの研究者がゲノム解析に携わることができるようになった。現在の課題は、2倍体や多倍体のゲノム配列を正確に解析することと、単一の参照ゲノム配列からパンゲノムグラフに切り替えることで種内多様性をよりよく反映させることである。”
Excited to share our review article about long read sequencing & plant genomics @qpb_journal:https://t.co/S2inSjLX4e
— Boas Pucker (@boas_pucker) March 11, 2022
Fruitful collaboration with @IkerIrisarri, @Jan_de_Vries & Bo Xu. Thank you!#Genomics #Bioinformatics
論文中では植物の高分子DNA抽出方法についても引用して議論しています。
4/26
"Long-read nanopore sequencing provides significant clinical utility when assessing the parental origin of de novo variants."
Another example of the clinical utility of long-read @nanopore sequencing in the #NHS. This time to determine the parent-of-origin of a de novo variant in UBE3A. 🧬
— Christopher Watson (@ChrisM_Watson) 2022年4月26日
Written with @MedeshEamonn and colleagues as part of @NEYGenomics.
Available here: https://t.co/HHzbfyW0ou. .
4/30
Comparison of R9.4.1/Kit10 and R10/Kit12 Oxford Nanopore flowcells and chemistries in bacterial genome reconstruction https://t.co/F0Cj5fYM00 #bioRxiv
— bioRxiv (@biorxivpreprint) 2022年4月30日
5/11
Finally @nanopore Q20+ chemistry has arrived into our lab. I'm super excited to test it! pic.twitter.com/WAawXYigVR
— Jan Gawor (@gaworj) May 11, 2022
5/14
"75種の多様なイネ属を選択し(Supplemental Table S1)、Oxford Nanopore Technologiesロングリード(平均デプス68.71×)(Supplemental Table S2)およびIlluminaショートリード(平均デプス69.04×)(Supplemental Table S3)プラットフォームを用いてシークエンシングし、de novoアセンブルした。このコンティグを修正して染色体レベルのスキャフォールドにアセンブルしたところ、平均N50は33.08Mb、平均BUSCO(Benchmarking Universal Single-Copy Orthologs)スコアは98.25%に達した。また、他のバッチから得られた13ゲノム(Supplemental Table S4)、および公開データベースから得られた25ゲノム(Supplemental Table S5)も本研究に含まれる”
Long-read sequencing of 111 rice genomes reveals significantly larger pan-genomes https://t.co/I3NlMPRSqs pic.twitter.com/LAwlO6f5gd
— pangenome_papers (@pangenomepapers) 2022年4月9日
5/21
CB: we want to start with a complex mix of proteins - a complex sample like blood, yeast, bacteria, and run on the same hardware that we use for DNA. So anyone could do complex proteomics on a MinION, GridION, PromethION. This is hard, very difficult #nanoporeconf pic.twitter.com/F3eWzUydWV
— Oxford Nanopore (@nanopore) 2022年5月19日
CB: as you can see here we are starting to generate profiles for more than one amino acid. They are very reproducible. #nanoporeconf pic.twitter.com/Yxq70QW6Oz
— Oxford Nanopore (@nanopore) 2022年5月19日
SR: Raw read ‘simplex’ accuracy with Kit 14 and R10.4.1 nanopore modal accuracy 99.2 % (Q21) at 400 basepairs/sec. #nanoporeconf pic.twitter.com/RwykfXfY8m
— Oxford Nanopore (@nanopore) 2022年5月19日
SR: results from ultra-long duplex reads and HG002 dataset with 260 bps condition. Modal accuracy ~Q30. Longest, Q30 duplex read: 260 kbases. Longest Q40 duplex read: 144 kbases. Longest read that perfectly aligns to a human reference: 72 kbases #NanoporeConf pic.twitter.com/N6sDX9wTiA
— Oxford Nanopore (@nanopore) 2022年5月19日
LJ We have developed more than 8500 pores. There are new longer pores in the @nanopore R&D for people where the current homopolymers are not covered yet. How long do people really need? @NanoporeConf pic.twitter.com/63UxGqVriE
— Rasmus Kirkegaard (@kirk3gaard) 2022年5月19日
LJ: Kit 14 will allow users to tune their accuracy & output needs by modifying run conditions — programmable in the software. Early work has shown 99.6% raw read accuracy can be achieved when run at 28 degrees. At 34 degrees 99.2% is possible whilst generating a much higher yield pic.twitter.com/LRMDVxpIgn
— Oxford Nanopore (@nanopore) 2022年5月19日
LJ: A quick update on modal single-molecule accuracy. Older baselines (Kit 9/10) have lower accuracies but fast motor (420 bases/sec). The Kit 12 has Q20+ accuracies but slow motor (250 bases/sec) – there was a need to break the output/accuracy trade-off. #nanoporeconf pic.twitter.com/PHLcEuJuP5
— Oxford Nanopore (@nanopore) 2022年5月19日
JC: new ASICs are being assessed in-house. The next generation of ASIC is designed for low noise & low power to enable the development of new product formats. #nanoporeconf pic.twitter.com/dBJqaj8lHT
— Oxford Nanopore (@nanopore) 2022年5月19日
Nanopore-related things to keep an eye on: .pod5 [pore open data] (new file format), Dorado (new production basecaller), P2 (small PromethION), Mk1D (iPad pro + MinION combined[!]).
— Christopher Watson (@ChrisM_Watson) 2022年5月20日
6/24
”市販のプラスミドシーケンスオプションの代替として、オックスフォード・ナノポア・テクノロジーズのMinIONデバイスを使用した費用対効果の高い正確なプラスミドシーケンスおよびコンセンサス生成手順について説明する。"
7/6 (2021年のプレプリントの査読された論文)
”Oxford Nanopore R10.4を使用することで、ショートリードやリファレンスベースの研磨を行わずに、単離株やメタゲノムからほぼ完成された微生物ゲノムを生成できることを示す。(中略)7種の細菌と1種の真菌からなるZymo mock のシーケンスを通して、Oxford Nanopore R9.4.1 および R10.4 データからほぼ完成した微生物ゲノムを得ることができるかどうかを評価した。R9.4.1のデータとは対照的に、R10.4ではIllumina polishingの追加によるアセンブリ品質の大きな向上は見られなかった(図1c、補足図1)(補足;ONTだけで十分に高品質ということ)。R9.4.1からR10.4へのアセンブリ精度の向上は、ホモポリマーのコール能力の向上によるところが大きい(図1b、補足図2、図3)。(中略)R10.4データでは長さ<11 bpまでのホモポリマーの大部分が正しく分解されていた(補足図4)。”
珪藻Phaeodactylum tricornutumのT2Tアセンブリ(GCAリンク)。Oxford Nanopore MinIONフローセルR9.4.1とイルミナが使用されている。テロメア配列を組み立てるための戦略などとても勉強になります。
Our telomere-to-telomere genome assembly for my favourite diatom, P. tricornutum, is now published! https://t.co/7y0E84steb pic.twitter.com/sMEJ8u0jtI
— Daniel Giguere 🧬 (@DanielJGiguere) 2022年7月5日
ENAにFAST5のtar.gz (およそ102GB)も登録されていますね(リンク)。
7/13
Our new Kit 14 chemistry, for Q20+ raw read accuracy, is now available to purchase from the store as part of an open early access program. Kit 14 allows users to tune experiments for high-accuracy, high-yield #nanopore sequencing as required. Learn more: https://t.co/9RWnF47oqD
— Oxford Nanopore (@nanopore) July 12, 2022
7/18
"ナノポアリードは、ショートリードアセンブラによってどの程度アセンブルできるのでしょうか? 1/8"
I recently wondered: how well would @nanopore reads assemble in a short-read assembler? I.e. can ONT reads pass as Illumina reads in a de Bruijn graph assembly algorithm? Here are the results!
— Ryan Wick (@rrwick) July 18, 2022
1/8 pic.twitter.com/WZiGpKVjWc
8/22
If you missed my poster #isme18. Main messages: Long @nanopore reads enable recovery of HQ MAGs from soil!!! However, to cover the majority of the microbial community you need to sequence VERY deep due to the complexity of the environment. pic.twitter.com/FOaKxPTaiB
— Rasmus Kirkegaard (@kirk3gaard) August 18, 2022
しかし植物のベースコールもかなり改善したなー。Simplex call でも20あたりにピークが来てるのは嬉しい。 pic.twitter.com/OfTwVNvO3u
— 内藤健 (@drk0311) August 22, 2022
9/5
Researchers from the @DanaFarber have developed a strategy to basecall telomeric reads using a tuned nanopore basecaller, resulting in improved recovery and analysis of telomeric regions with minimal negative impact on other genomic regions. Learn more: https://t.co/xnspD1k8Vv pic.twitter.com/fPEOM3K1lB
— Oxford Nanopore (@nanopore) September 5, 2022
9/6
ナノポア最新ゲノム解析キットのKit14を用いて、ヒトHG002サンプルのテストランを行いました。従来キットに比べて、400bpsモード、260bpsモードのいずれもデータ量はそれほど低下することなく、顕著な精度改善がみられることが確認できました。#nanopore pic.twitter.com/VWP0M6ChwV
— 株式会社ジーンベイ (@GeneBayInc) September 6, 2022
9/10
R10.4
— Kosuke Kataoka (@kataksk) September 10, 2022
同じfast5をHacとSupでベースコールした結果
Qualityのピークは<20→<25
短いリードは削られてる感じ pic.twitter.com/HCU4mNfqK7
10/19
From what I can tell, the first freshwater angelfish genome was just published by a high schooler using a MinION to sequence the DNA of his dead pet: https://t.co/PVTPL1fcSb
— Rich Abdill (@richabdill) October 18, 2022
10/20
Multiple tweets discussed systematic errors at CC[A/T]GG sites in whole plasmid @nanopore sequencing service provided by @plasmidsaurus and @PrimordiumLabs. Here are my two cents on (#1) what causes the errors & (#2) what (not) to do to get reliable sequence of your plasmids. 1/n
— Gang Fang (@iamfanggang) October 19, 2022
11/18
Wonder what this little thing can do. Will we ended up with a P2 tower like for minion or will top-up packs to match the starter pack? What is it and what is it not? pic.twitter.com/rNE5v6DpTy
— Ola Wallerman🇸🇪 (@OWallerman) November 16, 2022
Hifiasm HiFi+UL integration is ready for beta testing. This new mode takes @PacBio HiFi and @nanopore ultra-long reads as input and produces longer phased contigs. It also works with trio or Hi-C for chromosome-long phasing. Add option --ul to provide UL reads. https://t.co/uwOyFBg9GO
— Heng Li (@lh3lh3) November 18, 2022
11/28
Another @nanopore sequencing blinder from @JoStockton1 in @beggs_lab:
— Andrew Beggs (@adbeggs) November 27, 2022
Apparently you can't amplify methylated DNA and maintain the methylation - well you can!
Top sample - conventional WGA
Middle sample - methWGA
Bottom sample - original sample pic.twitter.com/GxuAOnJlH9
12/8
Improvements in @nanopore sequencing reported at @NanoporeConf : A bit more than 50 % of duplex reads on native human DNA. Very few errors on a 5 kb fragment on duplex reads pic.twitter.com/HsaShCwOGJ
— GeT_Genotoul (@GeT_Genotoul) December 6, 2022
Not your standard iPad Pro case. Combining @Apple silicon with @nanopore technology for mobile DNA/RNA sequencing. The future is here 🚀 pic.twitter.com/cuAurvaHei
— Kieran H (@Kieran_Hejmadi) December 7, 2022
12/10
Duplex 率を上げてくれるツール!既存のデータにも使えるようです。是非お試しください!! https://t.co/sb3blr6AIK
— :marimiya (@marimiya_clc) December 9, 2022
12/15
ロングリード用にゲノム抽出しようと核単離を試みたら、うちの子は破砕後に最初のbuffer入れただけでこんなネバネバに。核単離どころではなくなったのだけど、リンク先のwash bufferで2回洗ったら改善しました。無事に高分子DNAの抽出done。https://t.co/HAix43tVSH pic.twitter.com/Fsx3a30Cq0
— こひみず (@shzk79) December 13, 2022
12/20
Excited to finally get R10.4.1+Q20 & looks like @nanopore only assembly is the way to go. Major yield & accuracy boost a bonus to our upcoming effort to sequence all of family Drosophilidae (@PetrovADmitri @danrdanny @danielrmatute @DarrenObbard @_hgellert @mbeisen et al) pic.twitter.com/MO8rhRx9of
— Bernard Kim (@Bernard_Y_Kim) December 21, 2022
ショウジョウバエ科のハエのONT onlyロングリードシークエンシング。一連のツイートで、R9.4.1+IlluminaとR10.4.1+Q20を比較している。シークエンシングデータも公開されている。
12/23
Nanoporeを使って長いリピート配列(20bp x 203, 40bp x 24)を含むミトコンドリアゲノムのを決めた、という原稿を書きました。頻繁に使われるillumina seqとmitozの組み合わせだとコントロール領域がほとんど失われたミスアセンブリになってしまいます。https://t.co/XrHtBkLCP7
— Takeshi Igawa (@islander_34) December 23, 2022
2023
1/12
Unidirectional single-file transport of full-length proteins through a nanopore https://t.co/HEVRHmkJmk pic.twitter.com/Ww6sgWs3Ob
— Nature Biotechnology (@NatureBiotech) January 9, 2023
1/24
The microalga Chlamydomonas reinhardtii can survive without telomerase and even without telomere sequence at some chromosome ends, but at the expense of extensive genome rearrangements. That's what we show in this new preprint:https://t.co/ljcfS1B8H5
— Zhou Xu (@Zhou_Xu_) January 24, 2023
2/11
ロングリードデータからがんの構造異常を検出するソフトウェア、nanomonsvについての新しいバージョンのプリプリントをアップロードしました。このバージョンでは新しいクラスの異常である"Single breakend SV"の検出方法、それによって何がわかるか?を示しております。https://t.co/0xK5QIWvNf
— 白石 友一 (@friend1ws) February 9, 2023
2/20
"ナノポアシーケンスでは、ロングリードによりMAGのアセンブリ品質が大幅に向上するため、微生物のメタゲノム解析に用いられることが多くなっているが、推奨されるDNA量は通常、環境サンプルの回収可能なDNA量よりも多くなっている。ここでは、Nanoporeライブラリー調製のために推奨量より少ないDNA量について、シーケンス品質、群集組成、アセンブリ品質、MAGsの回収率を測定し、評価した。”
MAGs: Dancing the Nanopore limbo - Nanopore metagenomics from small DNA quantities for bacterial genome reconstruction https://t.co/miYHZZbBG4
— Long-read metagenomics-Papers (@LR_metagenomics) February 17, 2023
2/22
Very excited that our nanopore sequencing protocol book of Methods in Molecular Biology series is finally out in print! Huge thanks to the contributing authors and Springer editors! https://t.co/6ePxQhXERv pic.twitter.com/dv0gCRRWsj
— Kazuharu Arakawa (@gaou_ka) February 22, 2023
3/2
Finally figured out how to upload the the R10.4.1 @nanopore fast5 data for the @ZymoResearch HMW mock in the @NCBI SRA that I produced last summer @JMF_Vienna . Enjoy some mock data and a bit of dorado benchmarks here: https://t.co/Fej9kor4vx https://t.co/vFk5nWxgnv
— Rasmus Kirkegaard (@kirk3gaard) March 2, 2023
3/3
ONT MinIONと高い互換性のあるイルミナのショートリードのライブラリーの作成
(Accepted October 21, 2022)
3/21
Automated Telomere-To-Telomere crop genome assembly using only @nanopore sequencing presented at international conferences; https://t.co/sNS1OHURIE #AGBTAg #nanoporecomf #T2T pic.twitter.com/m8xJb4LiDP
— Alexander Wittenberg (@AW_NGS) March 21, 2023
3/22
#nanoporeDayParis : Why limit yourself to short reads #singleCell when you can get isoforms with @nanopore ? Shared by @Genomique_ENS in lightning talks session. Building on a community trend. pic.twitter.com/Sp9eAVnOOz
— Olivier (@olivierlucas29) March 21, 2023
Very excited about this project with Andy Parrish @ExeterGenomes on long-read WGS for rare disease diagnostics! This SIL1 case where LR-WGS helped to resolve a complex duplication is a great example of where @nanopore WGS could add value to R14 service 🙌 @SWGLH @RDExeter @NHSgms pic.twitter.com/XwsbmCOXTa
— Gerald Goh (@gerald_goh) March 21, 2023
3/31
"オックスフォード・ナノポア・テクノロジー社のプラットフォームを用いたダイレクトRNAシーケンス(dRNA-seq)は、エピトランスクリプトミクスの分野を変革する有望な展望を持ち、近年ますます普及してきている。dRNA-seqから得られるリードは、RNAの塩基修飾やポリAテールの長さに関する解読可能な情報を含みながら、遺伝子転写物の全長までカバーする。dRNA-seqの可能性を探る多くの研究が発表されているが、DNAシーケンスと比較して、シーケンスの精度やエラーパターンはまだ十分に研究されておらず、特徴もあまりわかっていない。多様な生物のネイティブRNAサンプルや合成in vitro転写RNAを含む公開データセットについて、dRNA-seqのシーケンス精度を評価し、系統的エラーパターンを特徴付けた。"
Sequencing accuracy and systematic errors in nanopore direct RNA sequencing https://t.co/83SGrQDOpu #biorxiv_bioinfo
— bioRxiv Bioinfo (@biorxiv_bioinfo) March 31, 2023
4/7
We just updated our CARD protocol preprint with the new Kit14 data for cell lines! As expected, the biggest improvement was in indel calling. Outside of homolymers and tandem repeats (~1/3 of indels in GIAB benchmark) F1-score is 0.997. https://t.co/1I0AWFT5eC pic.twitter.com/hePWfdugiO
— Misha Kolmogorov (@MishaKolmogorov) April 6, 2023
https://github.com/nanoporegenomics/card_nanopore_wf
("ONTデータのバリアントコールとde novoアセンブリのためのpieplines。シングルフローセルONTシーケンスプロトコルに最適化されている。NIHのアルツハイマー病と関連認知症センターで、数千のヒト脳ゲノムのシーケンスと特性解析に適用されている。")
"Q:バルクとシングルセルのRNAシーケンスの精度は?A:期待したほど正確ではありません。実際、シングルセルレベルでは、PCRバイアスが大きく影響しています。(中略)。ナノポアキット14のケミストリーは、イルミナやPacBioよりも正確です。ホモトリマー補正を使えば、ほとんどのCMIエラーを補正できます。(RNA-seqの文脈で)なぜONTの方が精度が高いのか、というのが私たちの質問でした。私たちは、これはPCRの増幅エラーが原因ではないか、と考えました。PacBioとIlluminaはシーケンスの一部としてPCRを使用しますが、nanoporeはそうではありません。PCRは確かに影響を及ぼし、私たちが予想した以上の効果がありました。" (ONTの文脈を中心に抜粋)
New preprint from our group. Q: How accurate is bulk and single-cell RNA sequencing? A: Not as accurate as we expected. In fact, at the single-cell level, PCR bias has a significant effect. We quantify this experimentally. A 🧵 https://t.co/xXAMe1ajGB #SingleCell
— Adam Cribbs (@AdamCribbs) April 7, 2023
4/21
SituSeq, our method for offline, remote, in situ 16S rRNA Nanopore sequencing and analysis has now been published! @nanopore @ucalgaryscience https://t.co/rgbCRlatkH
— Jackie Zorz (@jkzorz) April 20, 2023
5/1
What a dream!! We have 26 metagenomes from the #Arctic #Qikiqtarjuaq using @nanopore to explore the microbes from brines,sea ice cores and sea water. Working together with an amazing team @el_pableras @ICMCSIC @PolarCSIC and Marcel Babin and team @LavalUniversity. Stay tuned!❄️ pic.twitter.com/mWME6kKdEb
— AcinasLab (@AcinasL) April 29, 2023
5/5
New blog post:https://t.co/bKb6gDQ1eU
— Ryan Wick (@rrwick) May 5, 2023
How accurate is an ONT-R10.4.1-only bacterial genome assembly? Short answer: ~Q50, but it depends!
(1/5)
LONDON CALLING 2023関連(自分が理解できたものだけ)
RNA seq関係
Oxford Nanopore was the first to enable direct RNA sequencing. The new kit (RNA 004) and flow cell for direct RNA sequencing demonstrates increased accuracy and output, with the potential to unlock a new field of biological analysis. #nanoporeconf https://t.co/mchKktqzAd pic.twitter.com/2VPEsubBlA
— Oxford Nanopore (@nanopore) May 18, 2023
@Libby_Snell_ introducing the new @nanopore direct RNA004 kit. Great increases in yield and accuracy. Looking forward to giving this a whirl ourselves. #NanoporeConf pic.twitter.com/AUuBnWN2wJ
— Mike Clark (@ClarksysCorner) May 18, 2023
@martinalexsmith at @NanoporeConf : “direct RNA is the cream of transcriptomics” 👌👌👌 pic.twitter.com/p9CEzF7GyY
— Roxana TEODOR (@roxana_teodor) May 18, 2023
Hanlee Ji describes how @nanopore RNA sequencing enables reading full length transcripts and monitor which allele within a single cell the transcript came from. One cell, one allele, one molecule, no amplification 🤯#nanoporeconf #LC2023 pic.twitter.com/1Y04gLYyyY
— Rasmus Kirkegaard (@kirk3gaard) May 18, 2023
その他
新しいMinIONいい感じ! https://t.co/b4MF2bWdzM
— :marimiya (@marimiya_clc) May 18, 2023
The small form factor minION evolving into a tricorder like device that can dock into iPad Pro (sequencing as … an Apple accessory!) - due out Q3 this year #nanoporeconf pic.twitter.com/zxRrfBnq5j
— Ewan Birney (@ewanbirney) May 18, 2023
Looking forward to running this on a MacBook Pro soon pic.twitter.com/QZJeDfVHL5
— Matt Loose (@mattloose) May 18, 2023
Benchmarking @nanopore basecallers on @awscloud #nanoporeconfhttps://t.co/t8bMf7YKLk
— Chris Seymour (@iiSeymour) May 18, 2023
🚀🚀🚀 #NanoporeConf https://t.co/1HPMAKPD5e
— Oxford Nanopore (@nanopore) May 18, 2023
On library prep at #Nanoporeconf, a description for PCR-free methods showing the difference between ligation (max output) and rapid mode (10minutes, minimal lab equipment needed). Ultralong reads (ULR) also enabled, all Kit14. pic.twitter.com/7xEUu3apD4
— Albert Vilella (@AlbertVilella) May 17, 2023
Mk1D: the next generation of MinION portable sequencing.
— Oxford Nanopore (@nanopore) May 18, 2023
Designed to be compatible with the iPad Pro which provides 5G connectivity & accelerated M1 processing. This combination delivers cutting edge nanopore tech with a market leading mobile platform #nanoporeconf pic.twitter.com/rkRHkfjrfO
We are making more, simpler, data analysis workflows. EPI2ME’s interface enables users of all expertise to perform end-to-end analysis, anywhere. Preconfigured workflows support broad applications, now including infectious disease, human variation and single cell. #nanoporeconf pic.twitter.com/ruuKrmc67p
— Oxford Nanopore (@nanopore) May 18, 2023
Did you know that @NEBiolabs #Monarch HMW DNA Extraction Kits are recommended by @nanopore for Ultra-Long DNA sequencing?
— New England Biolabs (@NEBiolabs) May 18, 2023
https://t.co/ugLDvufIfh#NanoporeConf
Webb #NanoporeConf structural variant calling benchmarking pic.twitter.com/capIc4oj6X
— Keith Robison (@OmicsOmicsBlog) May 18, 2023
Why methylation detection matters? @nanopore enables rapid access to native DNA information. #nanoporeconf #LC2023 https://t.co/XhW71uOTz8
— Rasmus Kirkegaard (@kirk3gaard) May 19, 2023
Follow this for a comprehensive account of our tech update from @AlbertVilella - and there’s a digest of it here too: https://t.co/qzq4dAeR8m #nanoporeconf https://t.co/KdbpHhPLbw
— Oxford Nanopore (@nanopore) May 20, 2023
5/26
"テロメアは、老化や染色体の完全性において中心的な役割を担っている。我々はONTロングリードシーケンスを用いて、植物に寄生する最も壊滅的な線虫であるMeloidogyne incognita, M. javanica, M. arenariaのゲノムを、他に類を見ないほど連続した状態で構築した。線虫に進化的に保存されているテロメリックリピート(TTAGGC)nは、これらのゲノムには見出されなかった。さらに、テロメラーゼ酵素や線虫のテロメア関連タンパク質のオルソログの証拠も見つからなかった。その代わりに、コンティグの片方の端に存在する種特異的な複合リピートが確認された。"
Combining ONT sequencing , bioinformatics and cytogenetics we unzipped the genomes of the most devastating plant-parasitic nematodes and identified new kinds of species-specific unilateral telomeric repeats, opening many perspectives. https://t.co/Auzf6JcqGc pic.twitter.com/TmJwGxzDpc
— Etienne GJ Danchin (@edanchin) April 20, 2023
6/21
Interested in 16S rRNA amplicon sequencing on @nanopore ? Check out our preprint (first-authored by the amazing @xuan_l1), where we validate high-throughput UMI-based sequencing of 16S rRNA genes using Q20+ chemistry. TLDR: 3 is the magic number!🪄🧬 https://t.co/JngP1y0lza (1/8)
— Ryan Ziels (@RyanZiels) June 20, 2023
Some @nanopore updates. We got 69 Gb on a bird genome on the Promethion. This is a huge improvement over the 40 Gb we were seeing. Final N50 was 16 kb. We used 1 year old spin column DNA. Also we have a marine invertebrate running for 120 plus hours. At 90 Gb of data. Unreal. pic.twitter.com/UMYjguXO0w
— Matthew Miller - 🧬 Biodiversity Genomics (@biodiversityge1) June 20, 2023
7/11
Our paper is out today! Bacterial genomics has exploded in recent years, but, with a few exceptions, researchers have ignored bacterial epigenomes. We analyzed hundreds of Bacteroides (and friends) isolates and found astonishing epigenomic diversity.https://t.co/SUpvmBbcLD
— Mike Tisza (@MikeTisza) 2023年7月10日
7/13
Just polished a @nanopore 10.4.1 assembled genome with illumina data using Polypolish and POLCA. Result: 1nt total changed in a 10.2 Mb genome. Nanopore assembly without Illumina polishing is right now. (ill data QC: 99% mapped, total 188nt/10.2M not covered in high GC organism) pic.twitter.com/FwYpdg7nLp
— Tue Sparholt Jørgensen (@tuesparholt) 2023年7月12日
9/1
What if a high schooler had the potential to sequence a genome at their fingertips? 🤔 CSHL’s @DNALC is collaborating with @nanopore to develop educational infrastructure and resources with the potential to make in-class DNA sequencing routine! 🧬https://t.co/xFwBBgkStw
— Cold Spring Harbor Lab (@CSHL) 2023年8月31日
12/7
Simplex is moving towards Q30.
— Oxford Nanopore (@nanopore) December 6, 2023
A tweaked version of the motor with existing basealling model improves accuracy to Q24. The new motor will seamlessly integrate into existing kits in Q1 2024, increasing robustness to provide consistent Q20+ in current conditions. #nanoporeconf pic.twitter.com/7cz2EsfTnM
With the tweaked motor + next-gen basecaller + Research conditions, the R&D team have achieved Q28 with simplex – with lots of headroom to make further improvements. #nanoporeconf pic.twitter.com/fZ4KCQwFAz
— Oxford Nanopore (@nanopore) December 6, 2023
TurBOT is a benchtop solution designed to offer automated extraction, library preparation, sequencing, basecalling, and data analysis for multiple samples, all within a single device.
— Oxford Nanopore (@nanopore) December 6, 2023
Register your interest: https://t.co/JLKT0aBDKa #nanoporeconf pic.twitter.com/xffkYkAUvl
Mk1C support
Basecalling speed continues to increase with the upcoming Dorado release (V5).
— Oxford Nanopore (@nanopore) December 6, 2023
This improvement is now beyond the capability of Mk1C. Therefore, we are bidding the Mk1C a fond farewell.
A discontinuation plan is in place and will be communicated next year. #nanoporeconf pic.twitter.com/EeAohg2ELb
"過去20年間で、標本の同定と種の発見の両方にDNAバーコーディングが有効であることが示されてきた。世代にわたる努力を反映し、DNAバーコード参照ライブラリーは現在120万種をカバーしており、その80%は動物界からのものである。~
バーコーディングはDNA抽出、PCR、シーケンシングの順で行われ、2017年までは最後のステップがコストの大半を占めていた。高度にmultiplexingされたサンプルからバーコードをリカバーすることで、Pacific BioSciences社のSequelプラットフォームはコストを90%削減し、検体あたり7.50ドルから0.25ドルに減少した。Sequel IIでは、5倍大きなプールを解析することで、シークエンシングを検体あたり0.05ドルに引き下げた。Sequelプラットフォームは高忠実度のバーコードを安価に生成するが、2つの限界がある。第一に、資本コスト(50万ドル)と年間サービスコスト(5万ドル)が高いため、中核施設への導入が制限される。第二に、同社のフローセルは十分に高価(2Kドル)であり、プロトコル開発の妨げとなっている。オックスフォード・ナノポア・テクノロジーズ社のシークエンサーは、高い資本コストとサービスコストから逃れることができるが、配列忠実度が低いため、これまでは分析コストはSequelを上回っていた。しかし、最新のフローセル(R10.4.1)の性能向上により、この差がなくなるかもしれない。この研究では、通常のMinIONフローセルが10万検体由来のアンプリコンプールを特性解析できるのに対し、Flongleフローセルは数千検体由来のアンプリコンプールを処理できることを示した。検体あたり0.01ドルというDNAシーケンシングは、今やバーコードワークフローの中で最も安価なステップである。DNA抽出のための簡素化されたプロトコルと超低容量PCRを組み合わせることにより、標本からDNAバーコードまで0.10ドルで移行することが可能になる。"
Can you sequence 100,000 DNA barcodes in a single@nanopore run? Find the answer in this new paper on barcoding arthropods using the latest generation of Nanopore flow cells to facilitate cost effective DNA monitoring of species!🧬👏https://t.co/wkZCnxqNzd
— BIOSCANEurope (@BIOSCANEurope) December 4, 2023
12/20
It hasn't been long since I last quantified @nanopore accuracy, but the release of Dorado v0.5.0 demanded another test:https://t.co/27iewu1Vbe
— Ryan Wick (@rrwick) 2023年12月18日
Biggest improvement I've seen in a while! Most of these ONT-only bacterial genomes are now >Q60 😲
My first preprint with the lab is out 🎉!
— Gregor Diensthuber (@GDiensthuber) 2023年11月29日
We benchmarked the ability of our own basecalling models to detect RNA modifications using @nanopore direct RNA sequencing.
Grab a coffee and check out the thread below 🧵!https://t.co/V8PJfHAtEP
1/10
2024/01/21
サイエンスZERO 未来が加速する!DNA解読“ナノポアシークエンサー
初回放送日: 2024年1月28日
just completed @Nanopore R9 vs. R10 for 48 different bacteria... identical workflows, only diff was R10 flowcell v14 chemistry
— Jonathan Jacobs 🧬🦠🕹️🏕️ (@bioinformer) 2024年2月8日
honestly rather underwhelmed
base calling much higher, but...
fewer & shorter reads, lower throughput
no difference in hybrid assembly quality
* R10フローセルに移行する場合のややネガティブな話題(たくさんの菌のゲノムを決定されている)。ベースコーリング精度は高いがリード数が少なく、そして短くなってしまい、かつハイブリッドアセンブリの品質に差はないという話。
"16S rRNA遺伝子と23S rRNA遺伝子を統合するプロセスの結果、曖昧さが減少し、より優れた亜種の分離が可能になり、より優れた種および亜種の相対的な細胞存在量の推定が可能になった。"
Full 16S and 23S rRNA gene-based,strain-level resolution of the microbiota of a mock bacterial microbiome using ONT nanopore sequencing https://t.co/HngRpszl35 🧬🖥️
— Stephen Turner (@strnr) 2024年2月16日
(注;この論文の著者ではない)
Please RT. Our method paper is finally published in Tree Physiology. "Efficient selection of a biallelic and nonchimeric gene-edited tree using Oxford Nanopore Technologies sequencing" https://t.co/e5E7QJlelA
— Naoki TAKATA (高田 直樹) (@naotak96) 2024年2月22日
#treephys #treephysiology #nanopore
2/29
"DNAシーケンシングによるそのプラスミド解析は、サンプル中のプラスミドDNAの存在量の少なさに悩まされている。ナノポアアダプティブサンプリングは、シーケンスプロセス中に興味のないDNA分子を拒絶することで、これらの問題を克服することができる。本研究では、2つの異なるアダプティブサンプリングツールを用いて、既知の細菌分離株における低存在プラスミドの濃縮に対するアダプティブサンプサンプリングの適用を評価した。その結果、有効期限切れのフローセルでも有意な濃縮が達成できることを示した。アダプティブサンプリングを適用することにより、de novoプラスミドアセンブリの質を向上させ、シーケンス時間を短縮することもできた。しかし、本実験では、標的配列と非標的配列が類似領域にまたがる場合のアダプティブサンプリングの問題点も浮き彫りになった。"
Ulrich J, Epping L, Pilz T, Walther B, Stingl K, Semmler T, Renard BY.0.Nanopore adaptive sampling effectively enriches bacterial plasmids. mSystems0:e00945-23.https://doi.org/10.1128/msystems.00945-23
https://doi.org/10.1128/msystems.00945-23
3/7
PromethION 2 Integrated (P2i) is moving to open early access.
— Oxford Nanopore (@nanopore) 2024年3月7日
With its onboard compute and integrated screen, the self-contained device is bringing high-quality, real-time sequencing to a wider range of labs.
Learn more: https://t.co/8rEKIWSuhV #WYMM pic.twitter.com/2rRnrovr2y
4/6
"転写産物は潜在的な治療標的であるが、細菌の転写産物は生物学的に未解明のままである。我々は、新たに作成したONTダイレクトRNAシーケンスデータを用いて、Escherichia coli K12株およびE2348/69株(Bacteria:γ-Proteobacteria)の転写産物を予測するアルゴリズムを開発・適用し、同時にListeria monocytogenes Scott A株およびRO15株(Bacteria: また、Listeria monocytogenesのScott A株とRO15株(細菌:ファーミキューテス)、緑膿菌SG17M株とNN2株(細菌:γ-プロテオバクテリア)、Haloferax volcanii(古細菌:Halobacteria)の転写産物を、公開されているデータを用いて予測した。500万件以上の大腸菌K12 ONTダイレクトRNAシーケンスリードから、2,484のmRNAが予測され、予測された大腸菌タンパク質の半分以上が含まれている。予測された転写産物の数は、予測に使用された配列データの量に基づいて株によって異なるが、調査されたすべての株において、予測されたmRNAの平均サイズは1.6-1.7 kbpであり、予測された細菌の5'-および3'-UTRのサイズの中央値は30-90 bpであった。細菌および古細菌の転写産物のアノテーションが不足していることから、ほとんどの予測は新規転写産物であるが、大腸菌E2348/69 LEE病原性アイランドにおいて転写後に生成された転写産物や病原性に関連する低分子RNAなど、これまでに特徴づけられたmRNAやncRNAも多数予測した。すべての株について、100-200 bpの範囲の低分子転写産物、および10 kbpを超える転写産物を予測した。"
Direct ONT RNA-seq!
— Cameron Thrash (@jcamthrash) 2024年4月4日
Deciphering Bacterial and Archaeal Transcriptional Dark Matter and Its Architectural Complexity https://t.co/a299auWiiQ
(この論文の著者ではない)
4/19
"PECATは、Nanopore R9、PacBio CLR、またはNanopore R10リードのみを使用して、二倍体ゲノムを効率的にアセンブルする。PECATは、他のアセンブラと比較して、より連続したハプロタイプ特異的コンティグを生成する。特に、B. taurus (Bison×Simmental)ではNanopore R9リードを使用してほぼハプロタイプを分離したアセンブルをし、人のHG002とNanopore R10リードの組み合わせでは59.4/58.0 MbのフェーズブロックNG50のハプロタイプを分離したアセンブルを達成している。"
De novo diploid genome assembly using long noisy reads. #GenomeAssembly #LongReads #NoisyReads @NatureComms https://t.co/CXuat4BWYf
— Javier Santoyo (@jsantoyo) 2024年4月18日
(この論文の著者ではない)
4/19
Ready for >Q30 ONT reads? Herro error correction now supports both R9.4.1 and R10.4.1. simplex reads w/ @domstanojevic @DehuiLin (CHM13 -R9.4.1) #nanopore #longreads pic.twitter.com/LgijFkgxsq
— Mile Sikic (@msikic) 2024年4月19日
5/8
Is @nanopore duplex basecalling beneficial for bacterial genome assembly? I did some tests, and it seems like it might be!
— Ryan Wick (@rrwick) 2024年5月8日
New blog post here:https://t.co/4nUVbhgQZy
We have released some raw @nanopore data for benchmarking purposes, along with some basecalled outputs.
— James Ferguson (@Psy_Fer_) 2024年5月1日
We use the subset of 20k or 500k for benchmarking tools and methods a LOT!
We thought they would be helpful to others too.
4/5kHz R10.4.1 & RNA004https://t.co/tY28fvnEle pic.twitter.com/fu165FYpDW
We provide the raw data in SLOW5 format, however it is very easy to convert to pod5 (or fast5 if you really need to) using blue-crab or slow5toolshttps://t.co/ophmDhcuW7https://t.co/JH245NdjYg
— James Ferguson (@Psy_Fer_) 2024年5月1日
If you are only interested in part of the genome or want to create your own subset from those we uploaded without downloading the full file, try slow5curl. A tool to selectively download only the reads you want/need, saving time and moneyhttps://t.co/5AynainDnq
— James Ferguson (@Psy_Fer_) 2024年5月1日
5/9
Copy+paste following commands to get chr22 from a 1TB BLOW5. <1h on a ~50Mbps.
— Hasindu Gamaarachchi (@Hasindu2008) 2024年4月21日
wget https://t.co/Mzpbb4O8yo -O s5curl.tgz
tar xf s5curl.tgz && cd slow5curl-v0.2.1
wget https://t.co/mmZm7i4F61 -O rid.list
./slow5curl get https://t.co/wsAiIEEiDU --list rid.list -o chr22.blow5 https://t.co/PQNHTK7ilb pic.twitter.com/kOFuEy7fbc
(3/29)
ヒトゲノム計画の10倍程度の規模(300億円、15年計画)でRNAを解析するRNome計画の立案についての記事。Nanoporeを使ってやるらしい。Science誌。https://t.co/UtpCMWjyxx
— Jun Yasuda (@jyasuda1) 2024年3月29日
(3/14)
Last year I re-sequenced a genome using R10 @nanopore flowcells. I already had a super good ref for this strain, so I can assess the quality of the reads vs basecaller model by checking the % of identity. The median of Dorado's sup simplex reads is 99.5%!! 🤯1/n #genomics pic.twitter.com/i0DvPjJhHf
— S. Lorena Ament (@LoreAment) 2024年3月14日
5/23
LONDON CALLING 2024関連
Nobel Prize Laureate, Carol Greider, kicked off this year's #nanoporeconf discussing the use of nanopore sequencing to reveal conservation of chromosome end-specific telomere lengths pic.twitter.com/XRi9lY6sg1
— Oxford Nanopore (@nanopore) 2024年5月22日
Groundbreaking work from @ProfDanielKim showcasing the potential of RNA liquid biopsies for early cancer detection #nanoporeconf pic.twitter.com/dk8FDCdggr
— Oxford Nanopore (@nanopore) 2024年5月22日
(* リキッドバイオプシー(液体生検) ;血液や他の体液を用いて患者の負担を少なくして分析する技術。この発表タイトルだと、RNA分子をシークエンシングして解析することで、がんやその他の疾患の早期診断やモニタリングを行うという内容と推測される)
#nanoporecomf Tetraploid coffee genome assembled with just one flow cell! #T2T pic.twitter.com/Ln17SoFoRl
— Alexander Wittenberg (@AW_NGS) 2024年5月21日
@nanopore Congratulations with your 10 years! This is just the beginning. #nanoporeconf pic.twitter.com/Wbx24CNm2n
— Alexander Wittenberg (@AW_NGS) 2024年5月22日
One order of magnitude more output from direct RNA sequencing using the latest chemistry! Inspiring talk by Laura White from University of Colorado at #nanoporeconf pic.twitter.com/5U1FhG0txV
— Cristina Gamba (@gamba_cristina) 2024年5月22日
Through a combination of chemistry and machine learning advances, we have delivered a very large improvement in direct RNA sequencing accuracy. pic.twitter.com/TmkaD5NgGV
— Mike Vella (@vellamike) 2024年5月22日
Introducing the MinION Mk1D. #nanoporeconf pic.twitter.com/5slfdsB3Uf
— Oxford Nanopore (@nanopore) 2024年5月22日
Exciting results showing 5hmC hemi-methylation in whole mouse cerebellum. Uniquely enabled by @nanopore duplex sequencing. #NanoporeConf pic.twitter.com/aPXgu6cO0A
— Marcus Stoiber (@Stoibs11) 2024年5月22日
Q30 simplex achieved internally by ONT. 🤯 @The__Taybor @nanopore #nanoporeconf pic.twitter.com/qMbSUz9Op1
— Reindert Nijland (@ReindertN) 2024年5月22日
Transformer models #nanoporeconf pic.twitter.com/J7vBNDvybj
— Alexander Wittenberg (@AW_NGS) 2024年5月22日
At @nanopore we have successfully sequenced a yeast chromosome, telomere to telomere, in one read. pic.twitter.com/YlKvSy0QBb
— Mike Vella (@vellamike) 2024年5月22日
BAM, FASTQ or POD5?
— Oxford Nanopore (@nanopore) 2024年5月22日
Unsure about different nanopore file types? This question came up during the workshops yesterday and we thought it was a useful discussion for the rest of the Community to hear.
For most applications BAM is the way to go. #nanoporeconf pic.twitter.com/pgCUYXf9ih
Best. Automated. Human. Assembly. Ever. From @Dorylophile #NanoporeConf pic.twitter.com/XeM6FYUxmt
— Marcus Stoiber (@Stoibs11) 2024年5月21日
(Doradoでエラー事前にエラー修正したONTリードとPore-Cを組み合わせると、自動のアンブリだけでhumanの2倍体染色体の30個の染色体(hap1とhap2)のT2Tアセンブリ+5個のscaffoldsレベルT2Tアセンブリが達成できたということらしい。)
Introducing ElysION, previously known as TurBOT.
— Oxford Nanopore (@nanopore) 2024年5月23日
Steph talks us through the fully automated sample-to-answer device. pic.twitter.com/BNFXGWSpmj
In her update @RosemaryDokos, shared some of the incredible breakthroughs over the last 10 years.
— Oxford Nanopore (@nanopore) 2024年5月23日
13,000 publications.
1 Nanopore Community.#nanoporeconf pic.twitter.com/JRTRGYen23
5/27
Last speaker of the RMedI symposium @ircm_mtl is Bastien Paré from @nanopore revealing the latest developments of their sequencing platform for #RNA research. pic.twitter.com/0ShmZdguhu
— Martin Sauvageau (@sauvagem1) 2024年5月24日
6/15
Error corrected ONT data assembled with Hifiasm. Almost as easy to work with as HiFi data https://t.co/hCHofcwzVE
— Willem van Rengs (@RengsWillem) 2024年6月14日
10/26
"オックスフォード・ナノポア・テクノロジー社のダイレクトRNAシーケンスは、全長RNA分子のシーケンスとトランスクリプトームおよびエピトランスクリプトームの解析のための戦略を提供する。現在、tailfindrやnanopolishのような定評のあるツールのほか、最近の2つのディープラーニングモデルを含む、ポリ(A)テール長の推定に利用可能なツールがいくつかある: DoradoとBoostNanoである。しかし、ゴールドスタンダードデータセットに対するこれらのツールの精度のベンチマークは限られている。本論文では、ポリ(A)の尾部長が既知であり、ポリ(A)の尾部長の推定精度を測定するための貴重なアプローチを提供する合成RNA標準(Sequins)を用いて、4つのポリ(A)推定ツールを評価する。4つのツールはすべて、正しい値の12%以内に収まる平均尾長の推定値を生成した。Doradoは、実行時間が比較的速く、変動係数が小さく、ベースコールとの統合が容易であるため、全体的に好ましいアプローチとして推奨される。”
We used synthetic RNA to investigate accuracy of inferring polyA length from native RNA sequencing on @nanopore https://t.co/kEP6exer3e
— Lachlan Coin (@lachlanjmc) October 26, 2024
Nanopore Ultra-long Sequencing and Adaptive Sampling Spur Plant Complete Telomere-to-Telomere Genome Assembly #resource #MolecularPlant https://t.co/2NjdjSFlaI pic.twitter.com/JIL31htocz
— Molecular Plant & Plant Communications (@MPlantPCom) October 25, 2024
2025
03/04
"D. melanogasterの最初のTGSデータセットがリリースされたとき(Kim et al. 同じデータセットが2つの困難なY染色体領域を解決していたことを考えると、これはさらに驚くべきことであった(Carvalho et al. 2015; Krsticevic et al.) Carvalhoら(2016)はこの偏りを、Kimら(2014)の塩化セシウムDNA精製の使用によるもので、これは密度に基づいてDNAを分離する。軽いATrich配列は主DNAバンドから分離したバンド(「サテライトバンド」;Altemose 2022、およびその中の参考文献を参照)を形成し、ショウジョウバエのY結合イントロンの多くがATに富むサテライトDNAを含むことから(Reugelsら 2000)、彼らは欠失エクソンがこれらのサテライトに富む画分とともに誤って廃棄されたと提唱し、報告されたシーケンスの偏りは、サンプル調製における偶然のアーチファクトによるものと思われた。
ここで我々は、以前に観察されたバイアスは塩化セシウム精製によって引き起こされたものではなく、DNA抽出方法にかかわらず、現在のすべてのTGSデータセットに存在する系統的な問題であることを報告する。すなわち、最新のTGSプラットフォーム(Nanopore Q20 200× coverage; Kim et al. 2024; PacBio HiFi 50× coverage; Shukla et al. 2024)を用いたディープシーケンスでは、D. melanogasterのY連鎖遺伝子のいくつかのタンパク質をコードするエクソンを完全にアセンブルできないのに対し、65×カバレッジのイルミナでは、これらの同じエクソンを忠実にアセンブルできる。さらに調べたところ、これらのエクソンは生のリードにはほとんど、あるいは完全に存在せず、一貫して単純なサテライト配列(例えば、(AATATAT) n )の近くに位置していた。我々は、これらのサテライトが複数のステップでTGSシーケンシングを妨害することを発見した: (i)リードの開始が阻害され、サテライト領域内で開始するリードが非常に少なくなる。(ii)リードの伸長が阻害され、サテライトが存在するとリードが短くなる;"
Strong sequencing bias in Nanopore and PacBio prevents assembly of Drosophila melanogaster Y-linked genes https://t.co/Gq63DMIQ5w #biorxiv_genomic
— bioRxiv Genomics (@biorxiv_genomic) March 1, 2025
長い繰り返しのサテライトによって強い再現性でロングリードシークエンシングバイスが発生するという報告です。思ったよりゲノムアセンブリがうまくいかない方はこれが関係しているかもしれません。
4/18
Serious boost from @nvidia 3090 to 4090 and then again to 5090 for @nanopore basecalling 🚀🧬🖥️ https://t.co/xKu0k9tUhX pic.twitter.com/flHFKCpS1k
— Rasmus Kirkegaard (@kirk3gaard) April 18, 2025
5/5
I am really enjoying this. Science for the fun of it. Totally worth my personal investment on all these reagents, plastics and hardware pic.twitter.com/Yk2LiDT2YZ
— Alessio D. Nahmad (@alessionahmad) May 5, 2025
2025 London Calling関係
Simply amazing what you can do with @nanopore 😍 https://t.co/HSz7KfgZB4
— Pedro Vilanova 🧬 (@pfvilanova) May 21, 2025
Tweet 1/2
— Daniela Bezdan (@Daniela_Bezdan) May 21, 2025
ONT enables direct detection of DNA/RNA modifications without consensus calling. Recent accuracy benchmarks show:
• 6mA: 99.65%
• 5mC (CpG): 99.60%
• 5mC/5hmC (CpG): 99.24%
• 4mC/5mC: 97.64%
Progress shown from NCM 2022 to #LC2025.#nanoporeconf @nanopore… pic.twitter.com/EFfEF2fQaI
Oxford Nanopore sequencing will be ubiquitous for multiomics….and the next step is proteomics.
— Oxford Nanopore (@nanopore) May 21, 2025
First panel-based proteomics assays, before driving towards full protein sensing in the future. #nanoporeconf pic.twitter.com/l5Z0RTjaF8
With the increased capabilities of Hopper and Blackwell GPUs for machine learning inference and the optimisations in Dorado v1.0 we are introducing a new class of model “hyper” for Q28+ simplex accuracy available on request #nanoporeconf pic.twitter.com/w3JFrKI3NI
— Chris Seymour (@iiSeymour) May 22, 2025
Marcel Nelen, UMC Utrecht.
— Keith Robison (@OmicsOmicsBlog) May 22, 2025
Must get to 30X on single flowcell without washing & reloading for ISO certification as those introduce risk of sample swap pic.twitter.com/E1O9mNNTYX
Human phased T2T (20 T2T chromosomes + 9 further scaffolds) assembly from standard ligation library + 10X PoreC on a single PromethION flowcell (the unreleased high output chemistry)#Nanoporeconf pic.twitter.com/1SuBYjUMlc
— Keith Robison (@OmicsOmicsBlog) May 22, 2025
I get a bit tired of seeing new iterations on the new @nanopore SmidgION ASIC rather than simply finding one in my mailbox one day 📬📦. We gotta get a solution for the home lab/at home testing and a replacement for the flongles for teaching nucleotide sequencing. pic.twitter.com/7Peo0yeiA9
— Rasmus Kirkegaard (@kirk3gaard) May 22, 2025
最後はナノポアシークエンサーのASICの派生を繰り返し出して、コストが上がり続けることに対して警鐘を鳴らしている。
Doradoのhyper modeのベースコール精度はより向上。しかしさらに多くのベースコール時間がかかる。
Got a chance to take the @nanopore hyp(er accuracy) model for a spin. Pretty decent improvement in read quality 🚀 but expect to spend quite some more GPU hours 😉
— Rasmus Kirkegaard (@kirk3gaard) June 4, 2025
(Reads were mapped with minimap2 and identity scores from nanoplot were used for phred score calculations) pic.twitter.com/NlDkfr7fr5
GPUによるベースコール速度の違い。余裕があればRTX5090が費用効果は良さそう。
Thanks to whoever quickly confirmed that it also goes for 4070 Ti Super getting 25 % faster 🎉 https://t.co/nLwitWchvN pic.twitter.com/dAP4rOuEzO
— Rasmus Kirkegaard (@kirk3gaard) June 4, 2025