Proksee (https://proksee.ca) は、細菌ゲノムのアセンブル、アノテーション、解析、可視化のための、強力で使いやすく、機能豊富なシステムをユーザーに提供する。Prokseeは、イルミナのシーケンスリードを、圧縮されたFASTQファイル、または生、FASTA、GenBank形式のアセンブル済みコンティグとして受け入れる。また、GenBankアクセッションや以前に生成されたProkseeマップをJSON形式で使用する事もできる。Prokseeは、アセンブルを行い(生の配列データの場合)、グラフィカルなマップを生成し、マップのカスタマイズやさらなる解析ジョブの起動のためのインターフェイスを提供する。Prokseeの特筆すべき特徴として、アセンブリのカスタムリファレンスデータベースを介して提供されるユニークで有益なアセンブリメトリクス、解析結果を個々の塩基分解能で表示および比較するために深く統合された高性能ゲノムブラウザ(Proksee専用に開発)、結果をマップにスムーズに追加したり他のフォーマットで検索および探索できる組み込み解析ツールの増え続けるリスト、データ共有や研究の再現性のためのグラフィックマップ、解析結果およびログファイルをエクスポートするオプションがある。これらの機能はすべて、ユーザーの需要に合わせて簡単に拡張でき、ウェブサーバーの堅牢性と応答性を保証する、慎重に設計されたマルチサーバーのクラウドベースシステムで提供されている。
help
Tutorial
https://proksee.ca/tutorials/getting_started
https://proksee.caにアクセスする。
New projectsのページが表示されている。最初に右上のLoginからアカウントを作成しておく。1分くらいで作れる。ログインすると、ページの右上にユーザーメニューが表示される。現在のストレージと(200MBのストレージが付属している)、所有しているプロジェクト/ジョブの数を確認できる。
注;ユーザーアカウントは任意だが、プロジェクトを他のデバイスやブラウザで簡単に利用できるようになるため推奨されている。また、ログインして作ったプロジェクトは削除されることはことないが、ログインなしのプロジェクトは7日後に削除される。
Prokseeの新しいプロジェクトは、ゲノム配列、シーケンスリード、またはCGView.js JSONファイルから開始できる。ゲノム配列を指定する場合、Genbank、EMBL、またはFASTA形式のファイルのゲノム配列を用意する。配列の長さは1,000~1,000万塩基の範囲、コンティグ数は1000以下である必要がある。NCBIのGenBankアクセッションも受け付ける。遺伝子アノテーションも表示されるので、fastaではなくGenbank、EMBL形式のファイルを準備した方が簡単だが、Proksee内でprokkaのアノテーションを行なってそのアノテーション結果を読み込むこともできるため、fastaでも大きな支障はない。

画像の下の方にあるのはexample。スタートを押すと使用できる。
生のシークエンシングリードは、シングルまたはペアエンドの2つのFASTQファイルとして指定する。各ファイルは1GB未満である必要がある。ファイル拡張子は、fastq、gz、zipのいずれかが使用できる。
サーバーの容量を節約するため、アップロードされたリードファイルはアセンブリが完了した後に削除される。また、ログインしていない時の新しいセッションのプロジェクトは、最終保存日から7日後に削除される。また、セッションプロジェクトに関連するジョブは、プロジェクトの最終保存日に関係なく、開始後7日目に削除される。
出力例
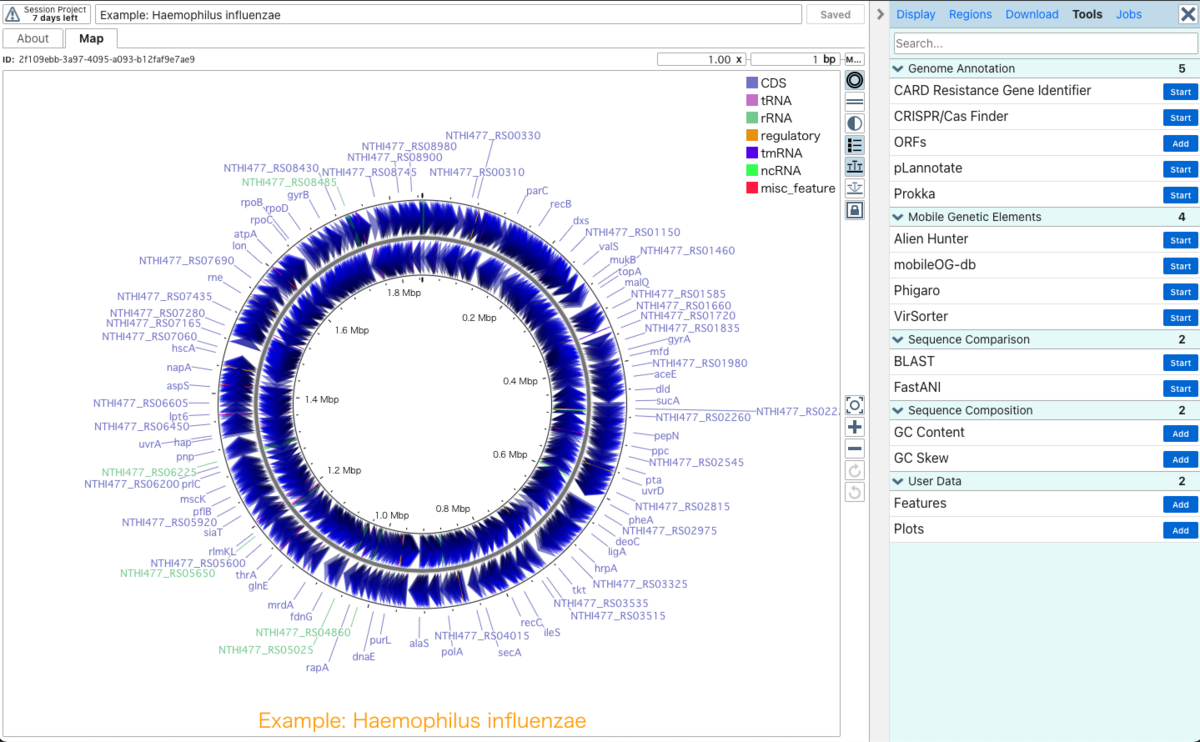
図は自由に拡大縮小、スクロールできる。

右上のボタンから視覚化方法を変更できる。=をクリックすると線状表示に切り替わる。

他のボタンで右上の凡例なし、カラー反転モードに切り替えてみた。
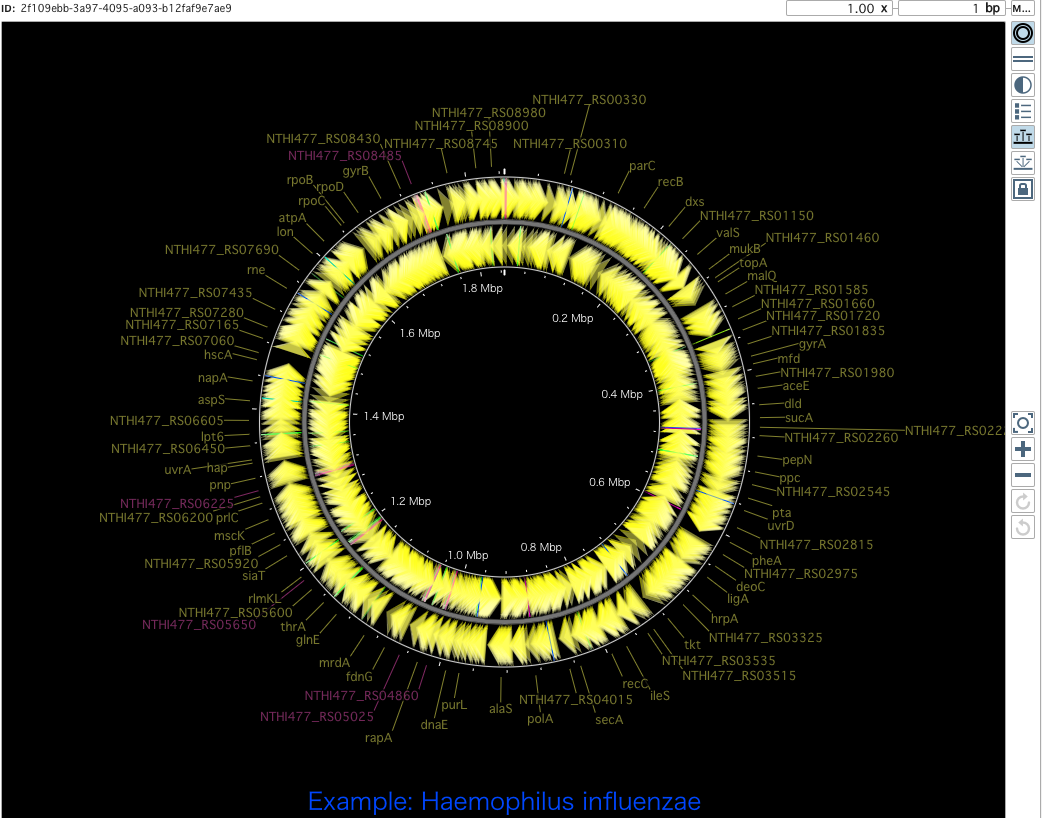
倍率は右上からも細かく変更できる。

右側のメニューから視覚化方法を非常に細かく調整できる。
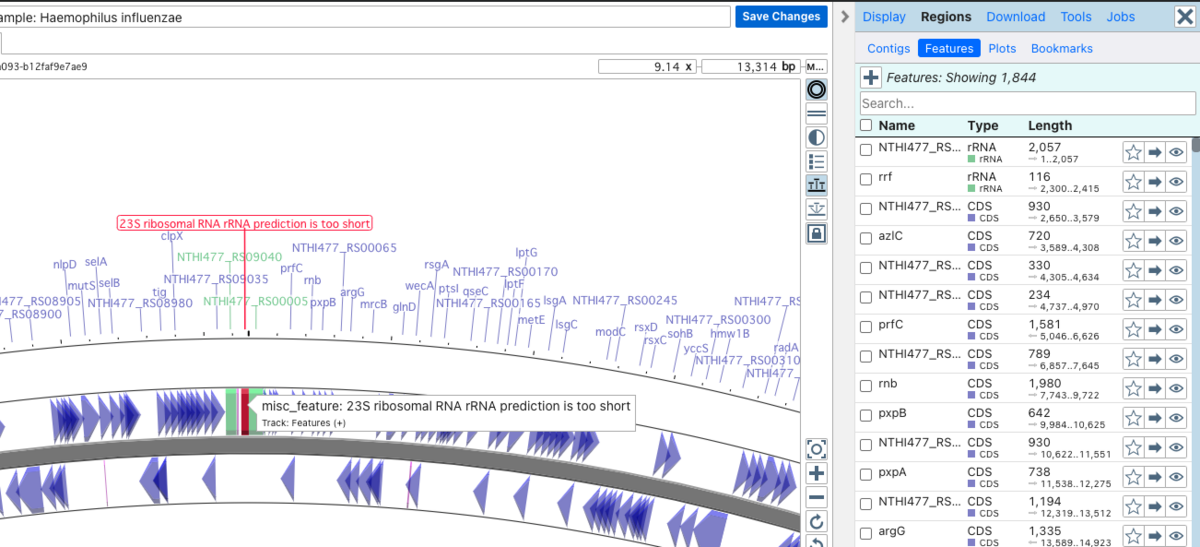
例えばORF1つの位置、向き、注釈、種類なども制御できる(Regions => Features)。

(ボタンやパラメータは全てhelpで詳しく説明されています)
Displayタブに切り替えて、新しいtrackを1つ追加した。同心円状のトラックが最外周に1つ追加された。

さらに新しいアノテーションを追加することもできる。Toolsタブを選択してツールを選ぶ。ここではCARDデータベース(Comprehensive Antibiotic Resistance Database)でAMR遺伝子のアノテーションをつけてみる。
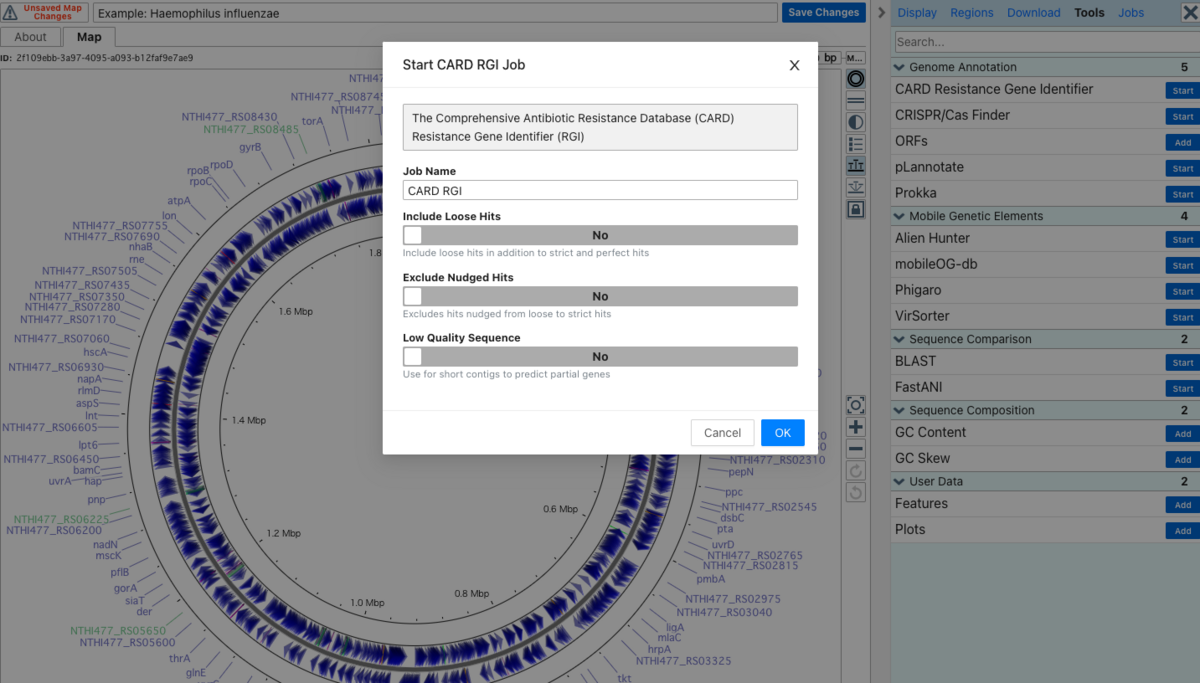
OKをクリック。
ジョブが終わるとtrackにアノテーションを追加するか聞かれる。
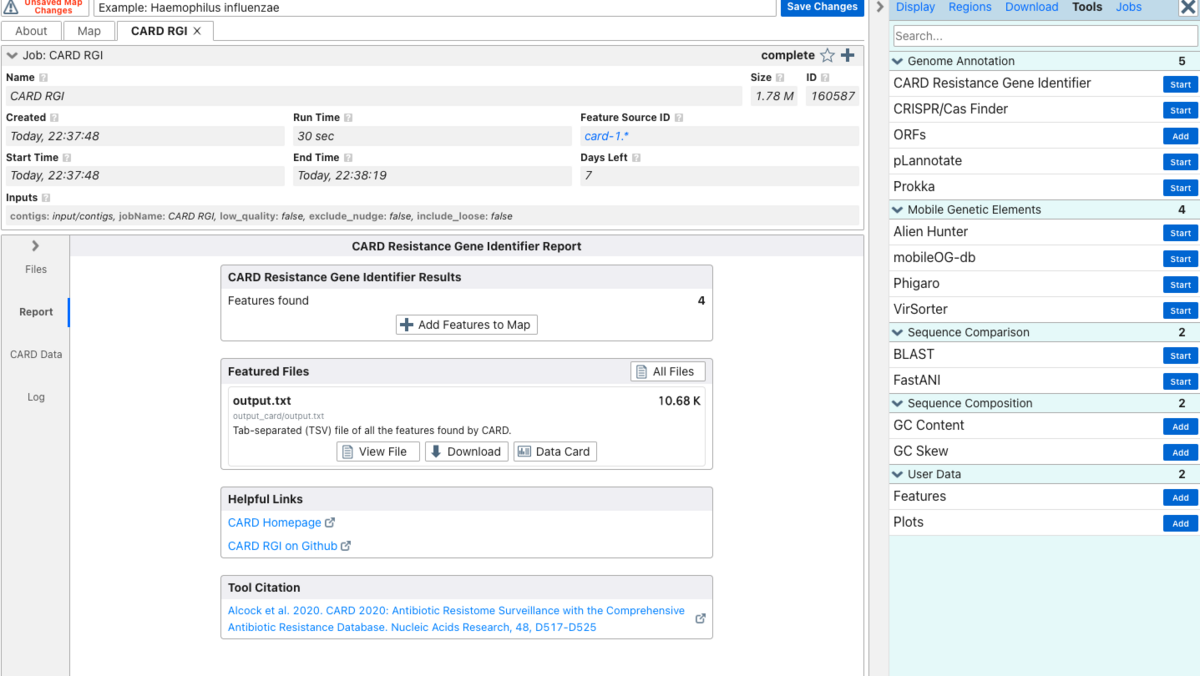
この画面から結果をダウンロードすることもできる。
どのトラックに追加するか指定してOKで決定。

アノテーションが追加される。メニューからリング間の余白も調整できる。
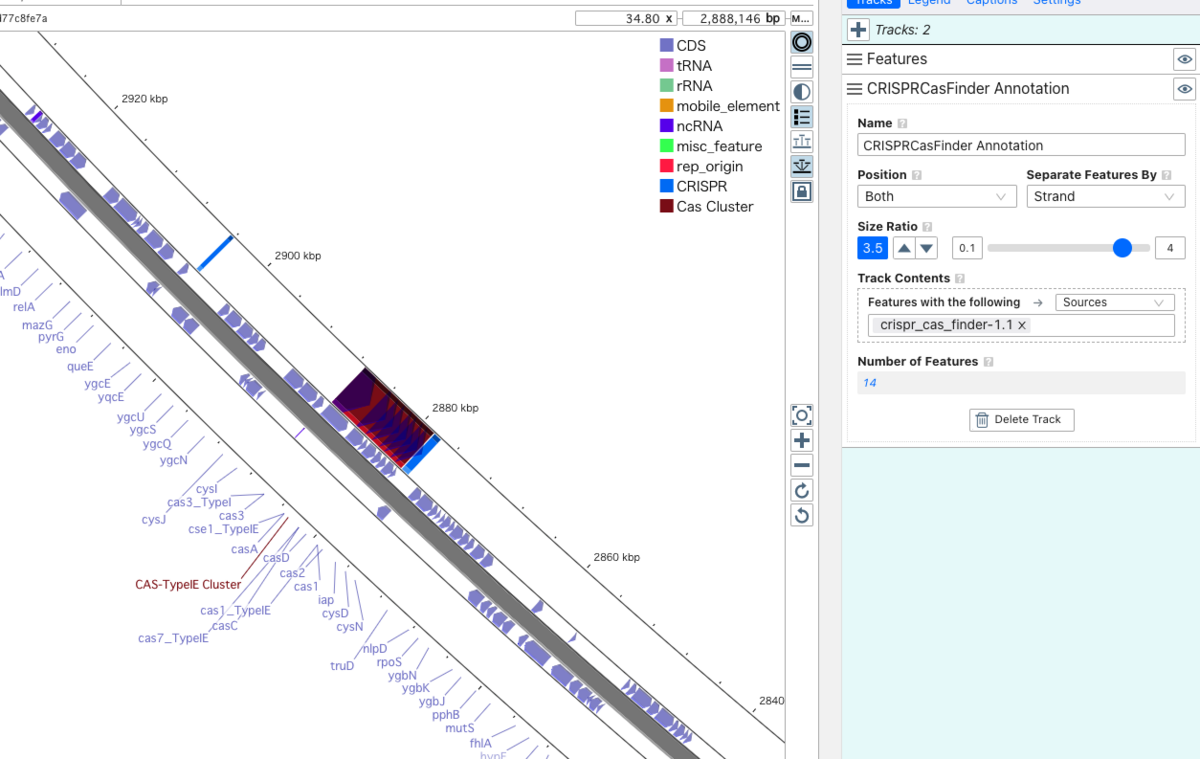
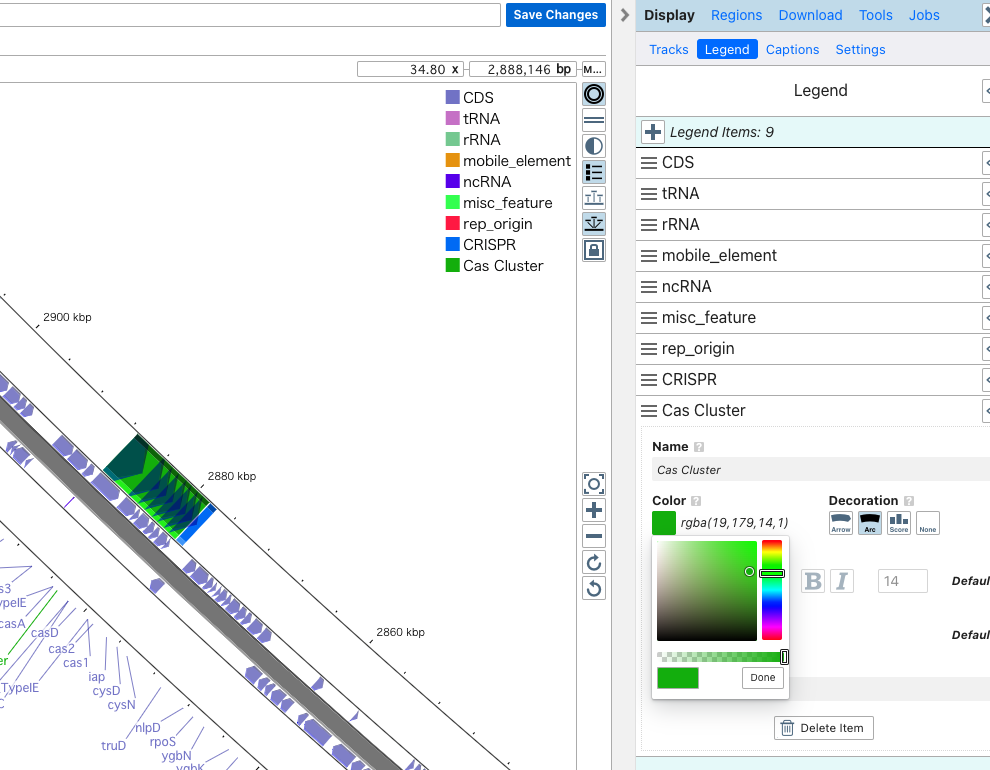
Display => Legendから新しくつけたアノテーションフィーチャーの色を緑に変更した。
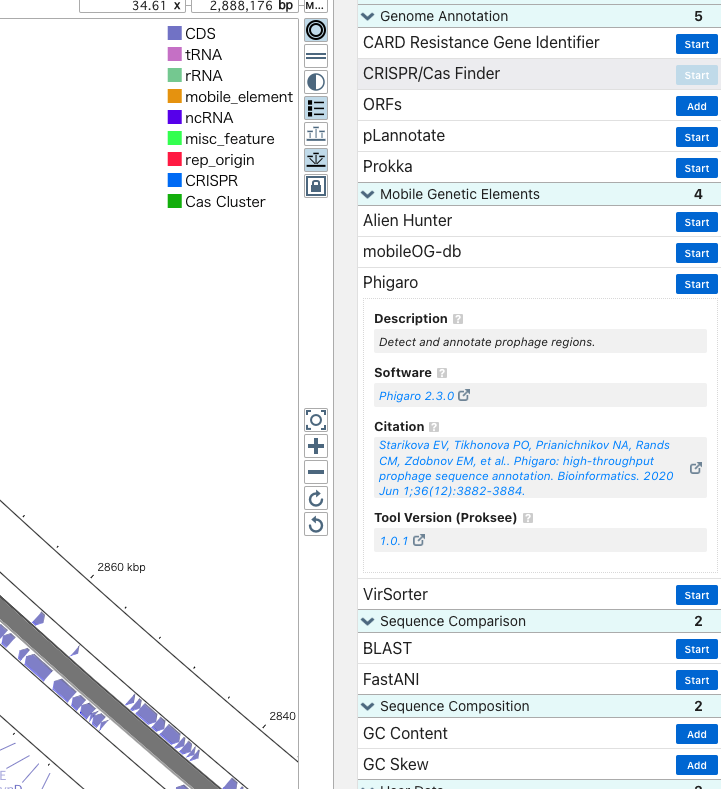
どのようなツールか分からない場合、展開すると簡単な説明が追加される。
図や配列はダウンロードから入手できる。

fastqファイルから進めるフローも少しだけ見てみる。シーケンスデータはSPAdesを用いてアセンブルされる。
exampleの結果のAssembleタブ
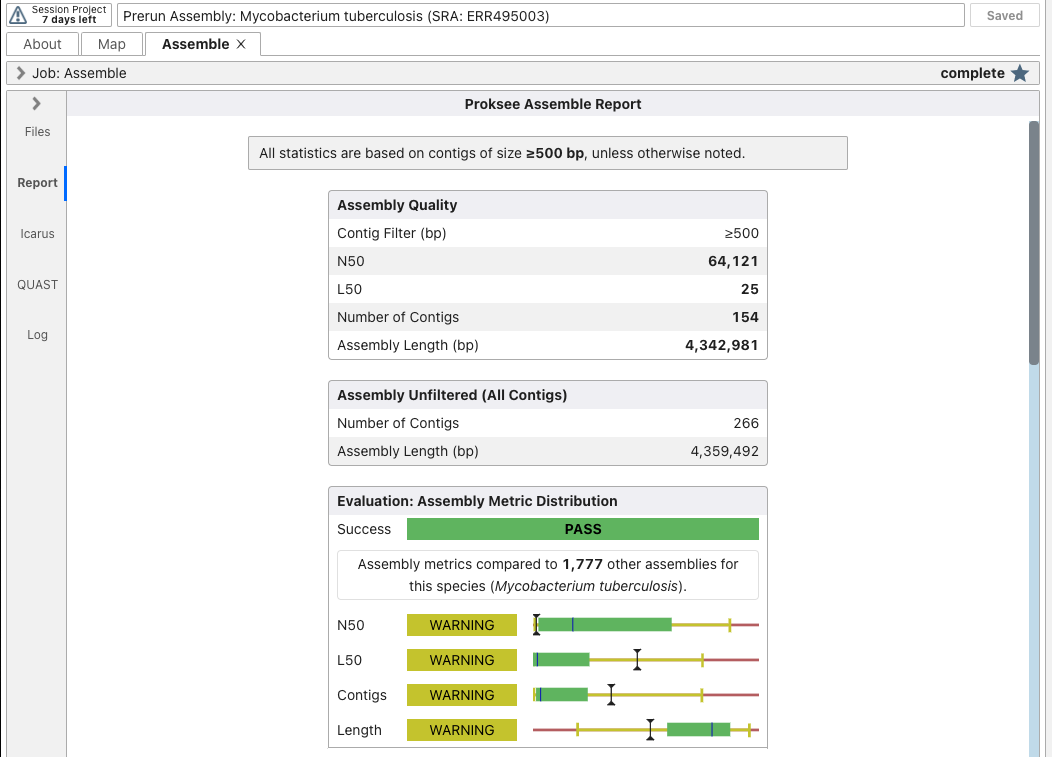
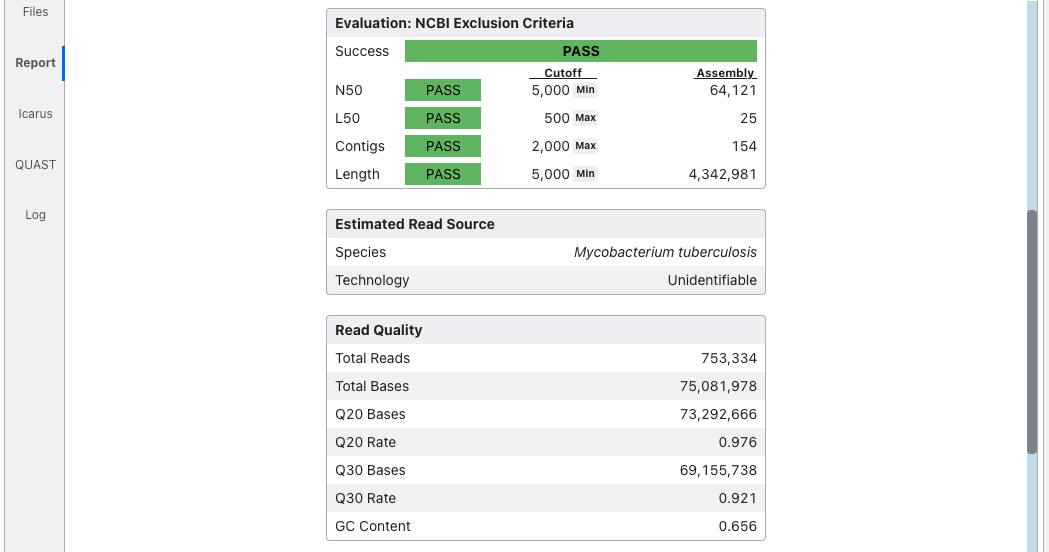
アセンブリの品質指標が表示され、PASS(緑色)しているか表示される。一般公開されているゲノム配列からのカスタムリファレンスデータベースと比較され、分類も推定される。
My projectsではプロジェクトを確認したりそのプロジェクトにアクセスする事ができる。

引用
Proksee: in-depth characterization and visualization of bacterial genomes
Jason R Grant, Eric Enns, Eric Marinier, Arnab Mandal, Emily K Herman, Chih-yu Chen, Morag Graham, Gary Van Domselaar, Paul Stothard
Nucleic Acids Research, Published: 04 May 2023
関連