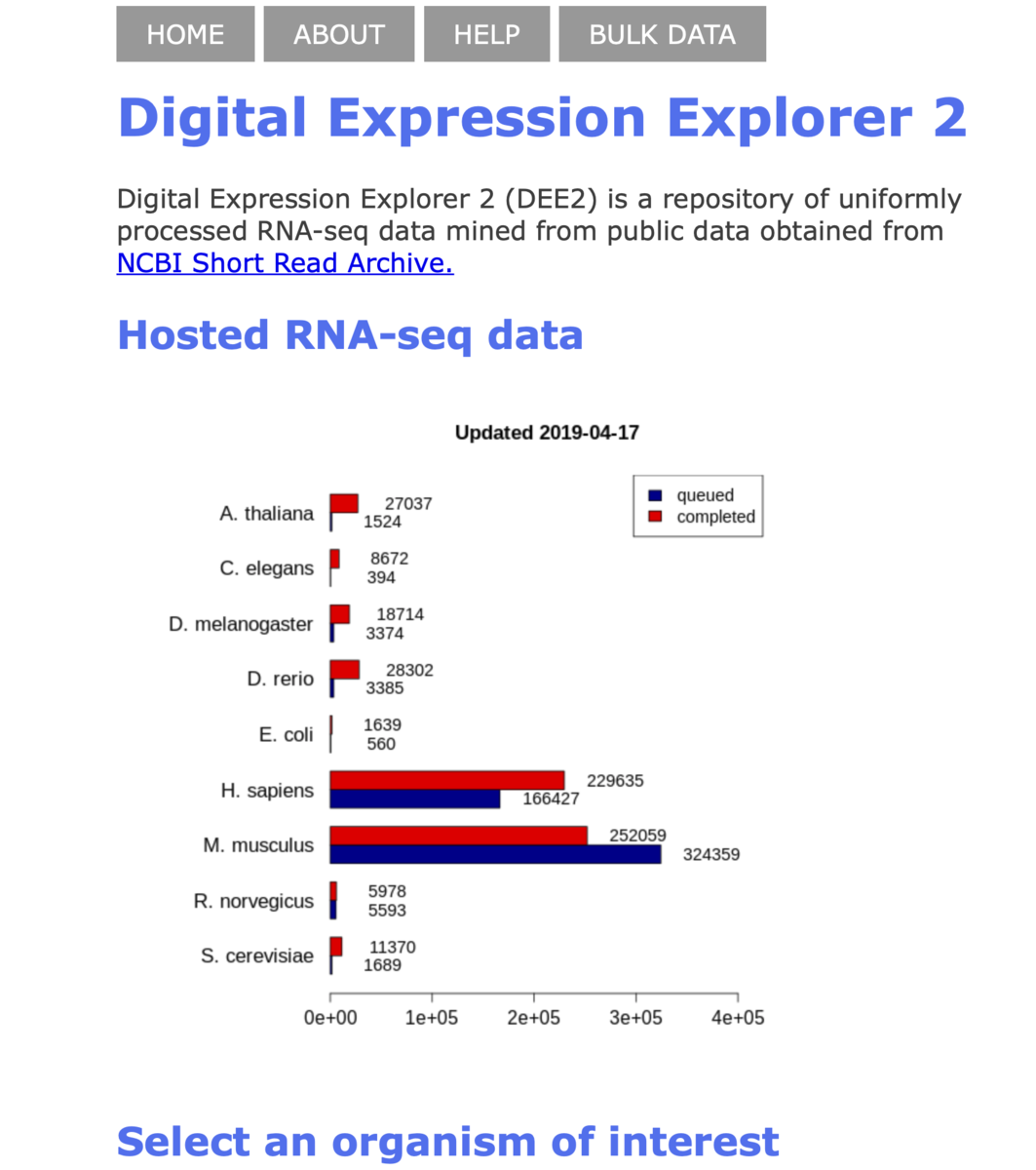
2021 1/9 ツイート追記
10年前の最初の記述以来、RNAシーケンス(RNA-seq)はトランスクリプトームにおける強力な方法となり、非常に正確な遺伝子発現の定量を可能にした[ref.1]。シークエンシングのコストが下がるにつれて、RNA seqのデータは科学文献でより一般的になりつつある。再利用と透明性の向上を目的として、rawデータおよび処理済みファイルの形でこれらのデータをGene Expression Omnibus(GEO)およびSequence Read Archive(SRA)[ref.2、3]に寄託することは当分野における標準的な慣行であり、ジャーナルにとって必須の要件である。しかし実際には、生物学者による広範な再利用を妨げるいくつかの障害がある。まず、SRAのrawシーケンスデータを処理するには、かなりの計算資源とコマンドラインの専門知識が必要になる。第二に、GEOがホストする処理済みRNA-seqデータは、さまざまなソフトウェアツールとゲノムアノテーションセットを利用するさまざまなフォーマットで作成されているため、メタアナリシスが複雑になる。科学界にとってこれらのデータの価値とそれらを生成するための多大なコストにもかかわらず、RNA-seqデータ集約の取り組みはヒトとマウスに大部分限定されているか[ref.4,5]またはクローズドソース/subscriptionサービス[ref.6]になっている。 BgeeDBは、多くの動物種に関するさまざまなライフステージ段階 (excluding disease, treatments, or genetic perturbations) でのベースラインサンプルの高品質測定に重点を置いて、アレイおよびシーケンスベースの発現データを提供している。 Expression Atlasは、有益なグラフィカルインターフェイスを備えた処理済みの発現マイクロアレイデータの最も包括的なリポジトリの1つだが、現在含まれているのは比較的少数のRNA-seqデータセットだけである[ref.8]。公共のトランスクリプトームデータの再利用を促進するために、本著者らは、多くの種類の下流分析と互換性があり、いくつかの主要な生物の一様に処理されたRNA-seqデジタル遺伝子レベルおよび転写レベルの発現データのオープンアクセスウェブベースリポジトリであるDigital Expression Explorer 2(DEE2)を開発した。
DEE2は3つの部分からなる。(i)SRAからrawデータセットをダウンロードし処理するパイプライン。 (ii)処理されたファイルが収集され、フィルタリングされ、そして編成/格納され、そしてジョブ待ち行列が生成されるデータレポジトリ。 (iii)ユーザーがメタデータを検索し、興味のあるデータセットを取得できるWebサーバー。 DEE2の編成の概略図が論文図1に提供されている。データ処理ノードはウェブサーバからSRAランアクセッション番号を要求し、SRAからrawデータを取得する。 処理されたデータはWebサーバーに送信され、検証され、DEE2リポジトリサーバーに中継される。 リポジトリサーバはさらなる検証チェックを実行し、新しいデータセットをリポジトリに組み込み、SRAdbV2 [ref.9]から対応するメタデータを収集し、未処理のジョブをキューに入れる。 リポジトリサーバーは、更新されたメタデータとジョブキュー情報をWebサーバーに送信する。 エンドユーザーは、Webブラウザ、コマンドライン、またはバルクダンプからデータを取得する。
DEE2パイプラインはコンテナ化を使用して迅速なアプリケーション展開を可能にし、さまざまなコンピュータシステムにわたる分析の再現性を保証する。 エンドユーザーは自分のハードウェア上でDockerイメージ[ref.10]を実行して、種名とSRA実行登録で指定された目的のSRAデータセットを処理することができる。 処理が完了すると、ユーザーはすぐに出力にアクセスできるようになり、DEE2リポジトリサーバーによる検証後、データセットは一般に公開されるようになる。 このように、パワーユーザーは、確立された分析パイプラインを使用することによって利益を得ると同時に、公共リソースの拡大に貢献する。 Dockerイメージに関する1つの懸念は、それらが管理者「ルート」許可なしに、例えば共有高性能コンピューティングシステムのユーザによって実行されることができないということである。 この制限に対処するために、イメージをroot権限なしで利用できるSingularity [ref.11]またはUDocker [ref.12]で使用するため変換できる。
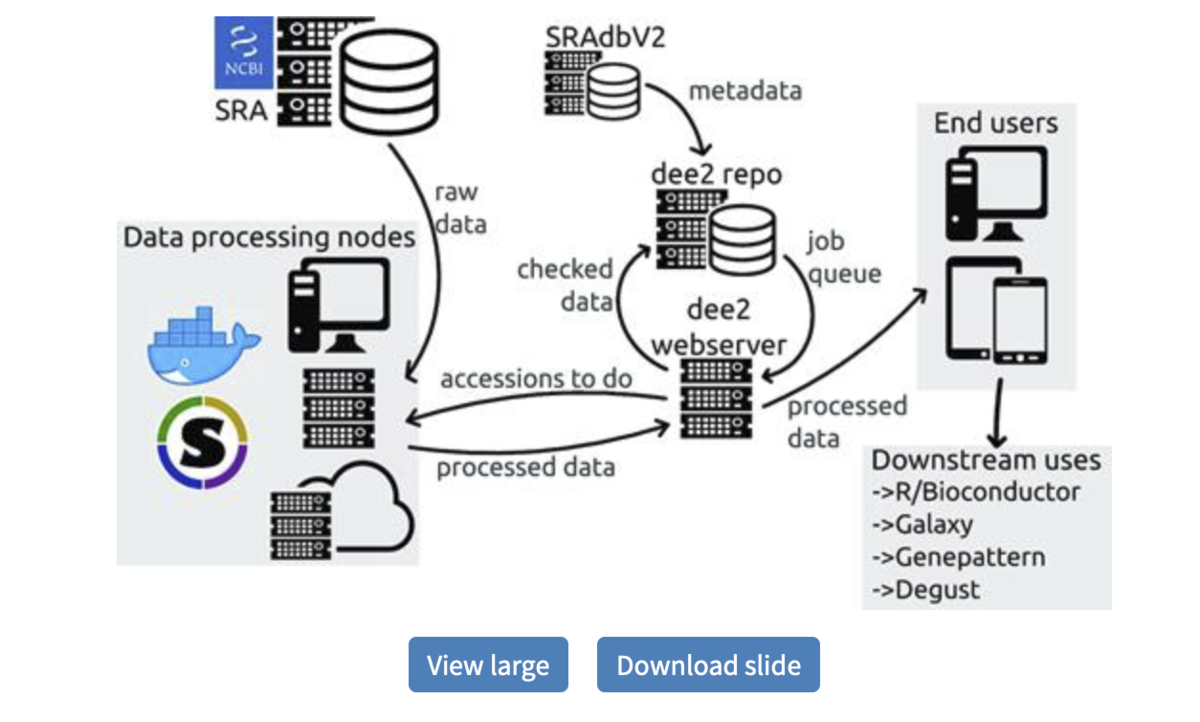
Overview of RNA-seq data processing, storage, and provision. Githubより転載
Important new update for the DEE2 database for #Arabidopsis: Over 60k SRA runs from >1863 gene expression projects now available at https://t.co/7JkBiDXucW
— Mark Ziemann (@mdziemann) January 6, 2022
Largest free resource of plant #RNAseq profiles in the world. Looking forward to see how you guys use this resource! pic.twitter.com/g64ET9Trgd
Digital expression explorer 2: a repository of uniformly processed RNA sequencing data. Paper now online. Thanks to reviewers who made suggestions to improve the paper and to the very professional @GigaScience editorial team. #genomics #RNA https://t.co/XJmHDKCiMv
— mdziemann (@mdziemann) April 4, 2019
Digital Expression Explorer: RNA-seq analysis in minutes
DEE2 data quality metrics
dee2/qc_metrics.md at master · markziemann/dee2 · GitHub
1、web版
http://dee2.io にアクセスする。
シロイヌナズナを指定し、GSE63462,SRP070529,SRR401430とタイプした。

19データセット見つかった。IDをクリックするとNCBI SRAにジャンプする。
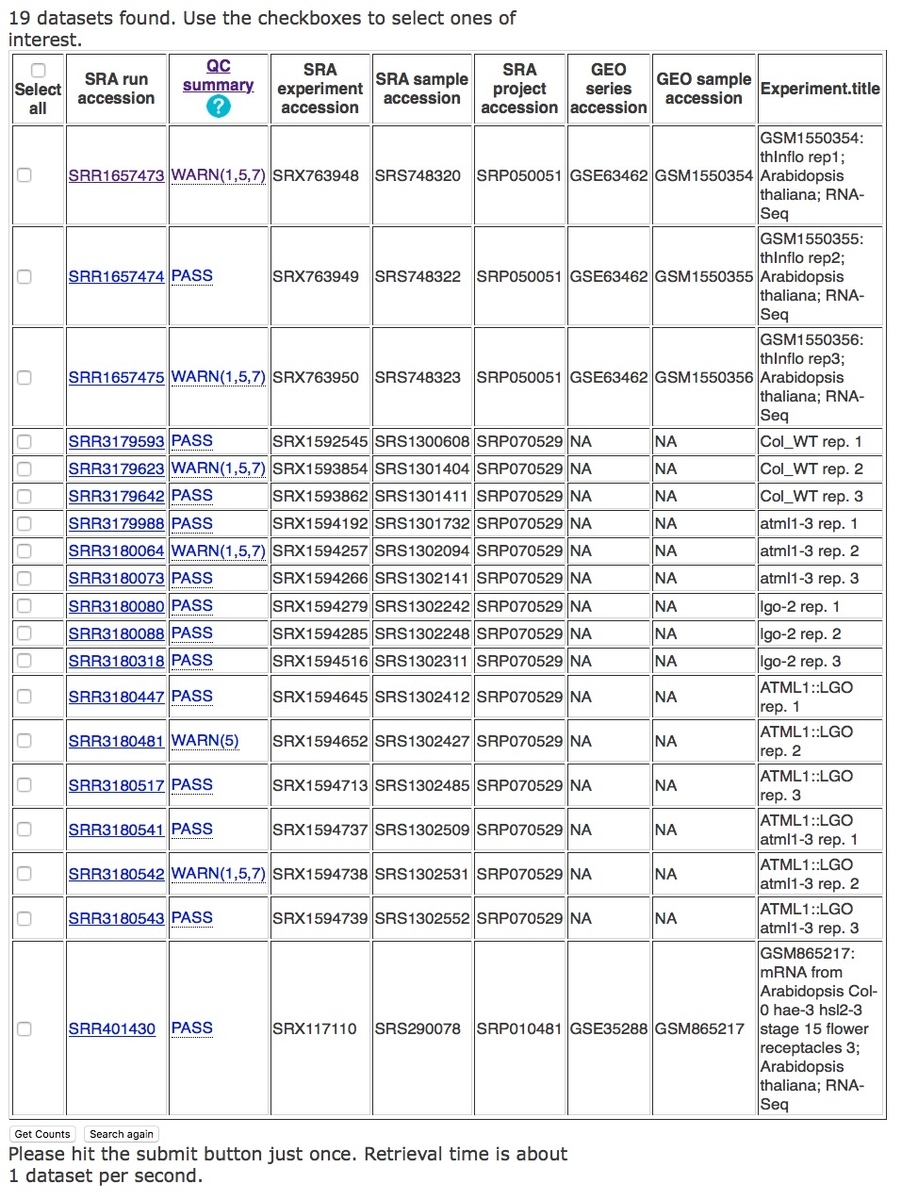
上のSelect allをクリックして19ヒット全てにチェックを入れ、下のGet Countsボタンをクリック。
QC結果、Gene counts(STAR)およびTranscripts counts(Kallisto)結果が瞬時にダウンロードされる(解析フローは論文の図2参照)。
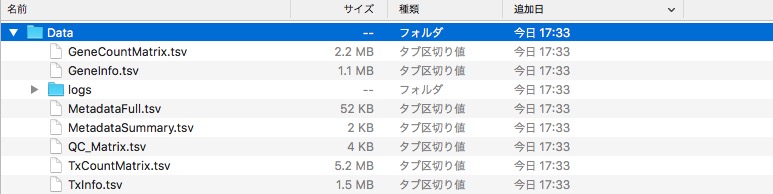
QC_Matrix.tsvをexcelで開いた。
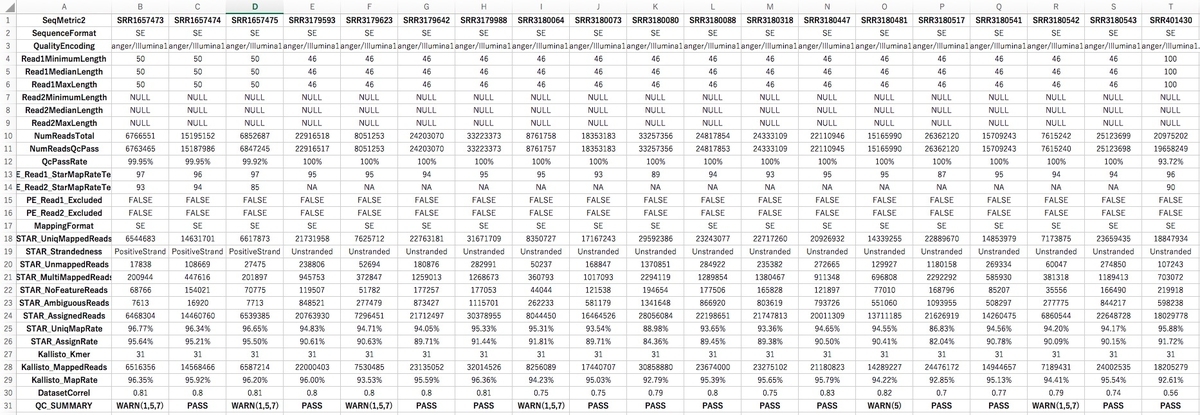
一番下の行が評価基準に従って決定されたデータの質。“pass”, “warn” 、 そして“fail”がアサインされている。 評価は複数項目にわたり、このリンク先の基準に従っている。
dee2/qc_metrics.md at master · markziemann/dee2 · GitHub
論文表2も参照。
geneレベルでの定量: GeneCountMatrix.tsv(STAR)

あっとゆう間ににリードカウントデータが得られた!
transcriptsレベルでの定量: TxCountMatrix.tsv(Kallisto)
(省略)
結果の違いは論文の引用ref.22参照(pubmed)。
あとは正規化して自由に比較できる。上に貼ったYou tube動画にあるように、オーサーらは取得したリードカウントの分析をDegustで行なうことを提案している(webベースで行うならiDEPも便利です)。
TopのSearch for keywordsからはキーワード検索もできる。
hit項目が多すぎる場合(>500)はバルクダウンロードのみ対応する。
2、コマンドライン版
コマンドライン(コンソール)でも使用できる。できることはweb版と同じだが、コマンドライン版はDEE2に登録されていないデータの解析、手持ちのデータの解析に役に立つ(*1)。web版と同じ以下のモデル生物種に対応している。
- A. thaliana
- C. elegans
- D. melanogaster
- D. rerio
- E. coli
- H. sapiens
- M. musculus
- R. norvegicus
- S. cerevisiae
特徴(Githubより)。
- Intelligent adapter detection and clipping
- Clipping of non-reference 5' bases (eg UMIs)
- Strandedness detection
- Parallel assignment of reads to genes and transcripts with STAR and Kallisto
- Thorough quality control logs
- Open source pipeline
- Distributed approach using containers link
- Ability to process own fastq files as well as those from SRA
DEE2はdockerイメージとして配布されているので、まずDEE2イメージを取得する。
docker pull mziemann/tallyup
例1、SRAのIDを指定してDEE2をランする。ここでは5つ指定。
docker run mziemann/tallyup \
ecoli SRR2637695,SRR2637696,SRR2637697,SRR2637698
ジョブが終わったらホストのカレントにコピーする("-alq"は停止したコンテナも含め最新起動のコンテナのIDのみ表示)。
docker cp $(docker ps -alq):/dee2/data/ .
data/ecoli/ディレクトリができる。出力を確認。
> ls -lh data/ecoli/
$ ls -lh data/ecoli/
total 744
drwxr-xr-x@ 9 user staff 306B 10 5 2018 SRR057750
drwxr-xr-x@ 9 user staff 306B 4 30 15:29 SRR2637695
-rw-r--r--@ 1 user staff 88K 4 30 15:29 SRR2637695.ecoli.zip
drwxr-xr-x@ 9 user staff 306B 4 30 15:34 SRR2637696
-rw-r--r--@ 1 user staff 89K 4 30 15:34 SRR2637696.ecoli.zip
drwxr-xr-x@ 9 user staff 306B 4 30 15:41 SRR2637697
-rw-r--r--@ 1 user staff 89K 4 30 15:41 SRR2637697.ecoli.zip
drwxr-xr-x@ 9 user staff 306B 4 30 15:50 SRR2637698
-rw-r--r--@ 1 user staff 90K 4 30 15:50 SRR2637698.ecoli.zip
drwxrwxrwx@ 8 user staff 272B 10 5 2018 SRR5985593_1
-rw-r--r--@ 1 user staff 29B 10 5 2018 date.txt
サブディレクトリの1つSRR2637698/の中身を確認
> ls -lh data/ecoli/SRR2637698/
$ ls -lh data/ecoli/SRR2637698
total 544
-rw-r--r--@ 1 user staff 86B 4 30 15:41 SRR2637698.attempts.txt
-rw-r--r--@ 1 user staff 0B 4 30 15:50 SRR2637698.finished
-rw-r--r--@ 1 user staff 122K 4 30 15:50 SRR2637698.ke.tsv
-rw-r--r--@ 1 user staff 30K 4 30 15:50 SRR2637698.log
-rw-r--r--@ 1 user staff 704B 4 30 15:50 SRR2637698.qc
-rw-r--r--@ 1 user staff 42K 4 30 15:50 SRR2637698.se.tsv
-rwxr-xr-x@ 1 user staff 61K 4 30 15:41 volunteer_pipeline.sh
TSVファイル
> column -t data/ecoli/SRR2637698/SRR2637698.ke.tsv |head
$ column -t data/ecoli/SRR2637698/SRR2637698.ke.tsv |head
SRR2637698_target_id SRR2637698_length SRR2637698_eff_length SRR2637698_est_counts SRR2637698_tpm
AAC73112 66 8.78165 19 267.332
AAC73113 2463 2364 503 26.2902
AAC73114 933 834 453 67.1128
AAC73115 1287 1188 584 60.7393
AAC73116 297 198 12 7.48841
AAC73117 777 678 839 152.899
AAC73118 1431 1332 558 51.7611
AAC73119 954 855 9361 1352.79
AAC73120 588 489 1423 359.558
transcripts id、raw read count、transcripts length、TPM補正値(参考)などが出力される。
例2、手持ちのシーケンシングデータを解析する。例えばヒトゲノムのデータ。
docker run -v $(pwd):/dee2/mnt mziemann/tallyup hsapiens -f sample1_R1.fq.gz,sample2_R1.fq sample1_R2.fq.gz,sample2_R2.fq
シロイヌナズナならmziemann/tallyup athalianaとする。
感想
SRAのRNA seqデータのクオリティを分析し、比較できる形で瞬時にカウントデータを取得できるデータベースです。モデル生物の研究者の方々にはたいへん実用的なデータベースだと思います。
引用
Digital expression explorer 2: a repository of uniformly processed RNA sequencing data
Mark Ziemann, Antony Kaspi, Assam El-Osta
GigaScience, Volume 8, Issue 4, April 2019
関連
*1
dataが自動でアップロードされるとあるので注意してください