2019 1/09 cocndaインストール追記 ,2/12 不要な文を削除, 4/12 dockerリンク追加
2020 4/2 インストール手順修正2022 03/28 help更新
さまざまな生態学的背景と進化の歴史を持つ原核生物ゲノムのコレクションが公開されている。このゲノムデータの大洪水は、微生物生態学と進化における重要な問題をより堅固に評価する機会を提供し、大きなゲノムデータの分析のための既存のバイオインフォマティクスアプローチを進歩させる必要性を強調している。そのような疑問の1つは、バクテリア(および他の微生物)が離散したクラスター(discrete clusters )(species)を形成するか、または水平遺伝子伝達(HGT)の頻度が高く、ゆっくりと減衰するキネティクスのために、連続的な遺伝的多様性が観察されるかどうかである。Closely relatedな少数のゲノムに基づく研究は、genetic continuum(連続性)が優先される可能性があることを示している(例えば、ref.1)。他方、HGTは種間の境界を歪めるほど頻繁ではないかもしれない、あるいは同じ種内の生物が種間の生物に比べてより頻繁に遺伝子交換を行い、それによって異なるクラスターを維持するとの研究がある(例えばref.2)。これら全ての研究に対する重要な批判は、それらが典型的に実験室の単離されたゲノムを用いて行なっているため、培養バイアスによって自然の多様性を適切に表していないかもしれないというものである。または、培養可能少数の系統由来の利用可能なゲノムしか使っていないため、堅牢な結論を出すことはできないという批判もある。(一文略)。種の定義は重要なアカデミックの練習であるだけでなく、大きな実践的な結果をもたらす。例えば、病原体の診断、国境を越えて生物を輸送することができる規制、隔離されるべき病原体、または人間や動物、植物に有益な有機物や有機物の混合物に関するコミュニケーションなど、すべて種がどのように定義されているかに根ざしている。
種の境界を評価する際の1つの基本的な作業は、2つのゲノム間の遺伝的関連性の推定である。The whole-genome average nucleotide identity(ANI)は、同じ種に属する生物が典型的にはそれらの間に≥95%のANIを示すため(ref.3,4)、近年この作業のための堅牢な方法として浮上している。 ANIは、任意の2つのゲノム間で共有されるすべてのオーソロガス遺伝子の平均ヌクレオチド同一性を表し、同一またはclosely relatedな種の株間比較で強い分解能(すなわち、80-100%のANIを示す)を示す。オルソログ遺伝子はゲノム間で大きく異なる可能性があるので、ANI測定は厳密にコアゲノムの進化的関連性を表すものではない。それにもかかわらず、それはバクテリア遺伝子プールの液体的性質を考慮し、したがって暗黙に共有機能を考慮しているので、種(ref.3)を定義するための伝統的なDNA-DNAハイブリダイゼーションをよく反映している。種を定義し、それらの進化的一意性を評価するための16S rRNA遺伝子のシーケンシングと比較して、ANIは、closely relatedなゲノム間でも高分解能を有するなどのいくつかの重要な利点を持っている。最後に、16S rRNA遺伝子などの普遍的に保存された遺伝子をコードしない(e.g., due to mis-assembly)メタゲノムまたはシングルセル技術を使用して、環境から回収されたドラフト(不完全な)ゲノム配列中のANIを推定することができ、ユニバーサル遺伝子ベースのアプローチと比較して少なくとも数百の共有遺伝子を使うことで研究および分類できる配列の数が大幅に拡大する。したがって、ANIは、DNA-DNAハイブリダイゼーションを、ゲノムの関連性の標準的な尺度として置き換える可能性について、国際的に認められている。(一文略)その強みにもかかわらず、伝統的なANI計算はアライメントベースの検索[例えばBLAST(ref.7)]に基づいており、アライメントアルゴリズムの二次的な時間複雑さのために計算上高価なままである。
ANI計算アルゴリズムはいくつかのバリエーションが提案されているが(ref.8-10)、これらは主に共有遺伝子を同定するための特定のアプローチを変更し、それらがすべてアライメントベースであるため実質的に計算を高速化しない。したがって、これらの手法および一般的に利用可能な計算資源に基づき、現在までに入手可能な数十万のオーダーの微生物ゲノムでANI値を計算することは、ほとんど不可能である。重要なことに、利用可能なゲノムデータは現存する原核生物多様性のわずかな部分であると推定され(ref.11)、決定された新しいゲノムの数は指数関数的に増加し続ける。したがって、利用可能なデータと将来のデータをスケールアップするためには、新しい計算ソリューションが必要である。
このような解決策のいくつかは、他の科学分野での「ビッグデータ」分析から概念を借りて最近提案されている。 MinHashは、World Wide Web(ref.12)で検索エンジンでほぼ重複するWeb文書を検出するために開発された、2セット間の類似性を迅速に評価する手法である。最近、この技術は、ゲノムアセンブリ(ref.13,14)やロングリードマッピング問題(ref.15)など、バイオインフォマティクスにおける新しい高速アルゴリズムの設計に成功した。 Ondov et al(ref.16)は、このテクニックを使用したANIの高速推定のためのMASHと呼ばれる最初の概念実証実装を提供した(紹介)。ANI計算についてMASHはアライメントベースよりも数桁高速であると報告されているが、ANIの計算問題に対してのMinHashの直接的採用は、不完全なドラフトゲノムについては不正確であることが判明している(ref.17) 。また、中程度に異なるゲノム
について(例えば80~90%のANIを示す時)、MASHがANIとどれくらいうまく近づけることができるかには限界があり、それは、ANIが共有された領域のみ計算するのと異なり、MASHの相同性検索が共有されていない領域についても相同性検索を行うためである。
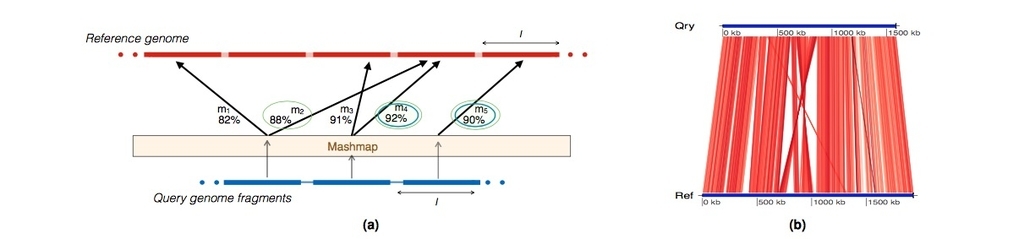
本研究では、MinHashベースのアラインメントフリーのシーケンスマッピングエンジンとしてMashmap(15)を利用した新しいアルゴリズムFastANIを開発し、ANI計算の計算上のボトルネックを軽減する。 FastANIは、80%〜100%のヌクレオチド同一性範囲に関連する完全およびドラフトクオリティのゲノムの両方について、アライメントベースのANI値と本質的に同一のANI値を提供する。したがって、FastANIにより、大量のゲノムコホートのペアワイズANI値の正確な推定、またはクエリーのドラフトゲノムと利用可能な原核生物ゲノムの完全コレクションの比較が可能になる。
Fig. 5. a. Graphical illustration of FastANI’s work-flow for computing ANI between a query genome and a reference genome.
(Preprintより転載。写真をクリックでPreprint PDFにジャンプします。)
図1にMASHとの比較あり。ANIが低い時のズレが問題だが、データセットD1の結果を見ると、fastANIの方がANIとよく相関している。データセットD3の結果もそうで、MASHはANIが高い時もズレがでる傾向にある。
Our latest: Are microbial species actually a thing? FastANI gives us a peek. More great work from @chirgjain. Great to see this published! https://t.co/2igLJex2rA
— Adam Phillippy (@aphillippy) November 30, 2018
インストール
ubuntu16.04でテストした。
依存
- Autoconf ( http://www.gnu.org/software/autoconf/ )
- either... GNU Scientific Library ( http://www.gnu.org/software/gsl/ ) or.. Boost Library ( http://www.boost.org ) (see "-with-boost" below)
- Zlib ( included with OS X and most Linuxes, http://www.zlib.net )
- C++ compiler with C++11 and openmp support
本体 GIthub リリースよりlinux向け実行ファイルのバイナリをダウンロードする。
#condaでも導入可能(mac、linux)
mamba create -n fastani -y
conda activate fastani
mamba install -c bioconda -y fastani
> fastANI -h #version 1.33
# ./fastANI -h
-----------------
fastANI is a fast alignment-free implementation for computing whole-genome Average Nucleotide Identity (ANI) between genomes
-----------------
Example usage:
$ fastANI -q genome1.fa -r genome2.fa -o output.txt
$ fastANI -q genome1.fa --rl genome_list.txt -o output.txt
SYNOPSIS
--------
./fastANI [-h] [-r <value>] [--rl <value>] [-q <value>] [--ql <value>] [-k
<value>] [-t <value>] [--fragLen <value>] [--minFraction <value>]
[--visualize] [--matrix] [-o <value>] [-v]
OPTIONS
--------
-h, --help
print this help page
-r, --ref <value>
reference genome (fasta/fastq)[.gz]
--rl, --refList <value>
a file containing list of reference genome files, one genome per line
-q, --query <value>
query genome (fasta/fastq)[.gz]
--ql, --queryList <value>
a file containing list of query genome files, one genome per line
-k, --kmer <value>
kmer size <= 16 [default : 16]
-t, --threads <value>
thread count for parallel execution [default : 1]
--fragLen <value>
fragment length [default : 3,000]
--minFraction <value>
minimum fraction of genome that must be shared for trusting ANI. If
reference and query genome size differ, smaller one among the two is
considered. [default : 0.2]
--visualize
output mappings for visualization, can be enabled for single genome to
single genome comparison only [disabled by default]
--matrix
also output ANI values as lower triangular matrix (format inspired from
phylip). If enabled, you should expect an output file with .matrix
extension [disabled by default]
-o, --output <value>
output file name
-v, --version
show version
dockerイメージ
https://hub.docker.com/r/staphb/fastani
ラン
1、シングルのクエリゲノムとリファレンスゲノム間の比較
fastANI -q query.fa -r ref.fa -o output
ジョブが終わると"output"ができる。
2、シングルのクエリゲノムと複数のリファレンスゲノム間の比較。リストファイルはゲノムのFASTAのパスを1行に1つずつ記載したものを指定する。
#リファレンスリストを作る。
cd genome/
ls *fasta > reference_list #注 実行時のカレントパスにfastaがないならフルパス指定する必要がある
fastANI -t 12 -q query.fa --rl reference_list -o output
3、複数クエリゲノムと複数のリファレンスゲノム間の比較
fastANI -t 12 --ql query_list --rl reference_list --matrix -o output
outputとoutput.matrixができる。Refseqのバクテリアゲノムを適当に100選んでAll vs All比較を行ったが、39秒しかかからなかった("-t 8"フラグをつけて実行)。
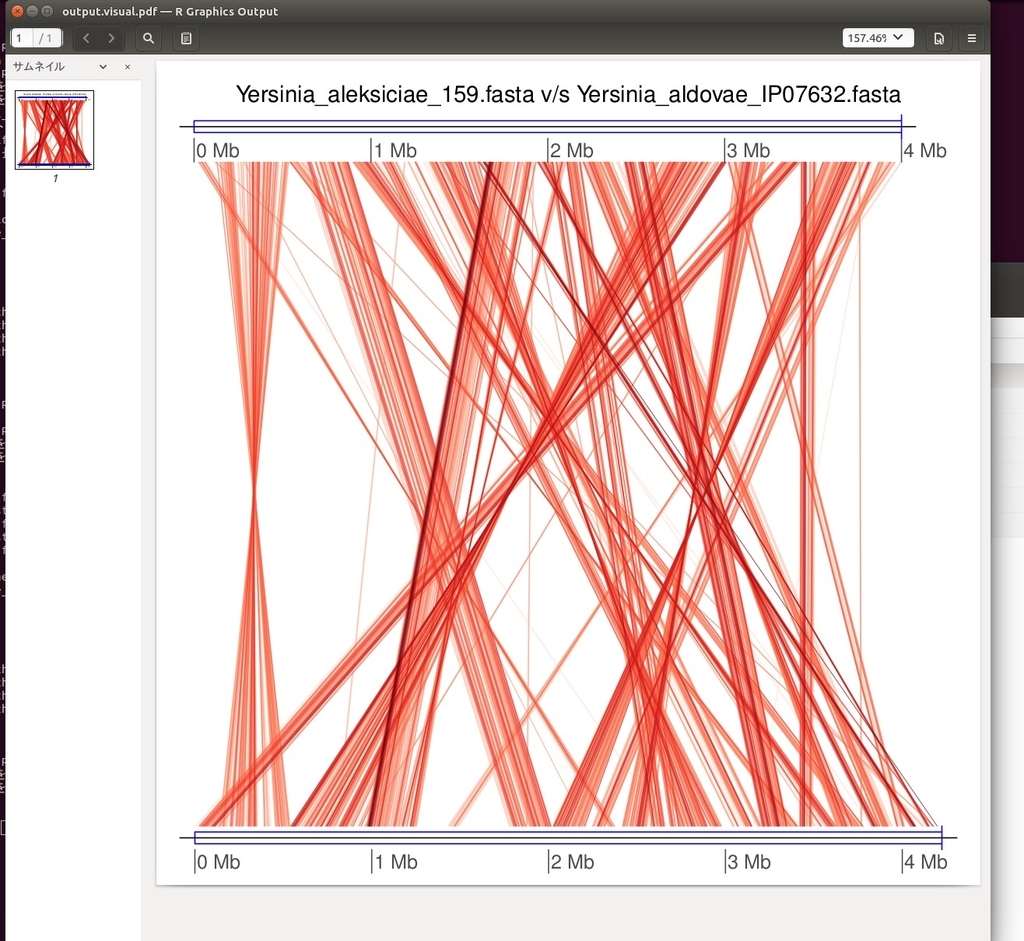
4、genoPlotRを使い2ゲノム間の比較結果を可視化する。レポジトリのRスクリプトを使う。
#genoPlotRがなければ入れておく(リンク)。R内で
> install.packages("genoPlotR", repos="http://R-Forge.R-project.org")
#ゲノム比較実行
fastANI -q query.fa -r ref.fa --visualize -o output
#outputとoutput.visualができる。できなければgenoPlotRが入っていないかもしれない。
Rscript scripts/visualize.R B_quintana.fna B_henselae.fna fastani.out.visual
出力。
引用
High-throughput ANI Analysis of 90K Prokaryotic Genomes Reveals Clear Species Boundaries
Chirag Jain, Luis M. Rodriguez-R, Adam M. Phillippy, Konstantinos T. Konstantinidis, Srinivas Aluru
bioRxiv preprint first posted online Nov. 27, 2017;
doi: http://dx.doi.org/10.1101/225342.
Preprint紹介後、2018年11月にNature Communicationsにアクセプトされています。
High throughput ANI analysis of 90K prokaryotic genomes reveals clear species boundaries
Chirag Jain, Luis M. Rodriguez-R, Adam M. Phillippy, Konstantinos T. Konstantinidis & Srinivas Aluru
Nature Communications volume 9, Article number: 5114 (2018)
Mashmap
MashmapはD-GENIESでも使われています。
fastANIを使ったクラスタリング