BMC Bioinformatics. 2010 Apr 29;11:213
COBRA(Constraint Based Reconstruction and Analysis)フレームワークに基づくゲノムスケールの代謝再構成は、生物の代謝能力を解析し、実験データを解釈するための貴重なツールである。このような再構成や解析手法の数が増えるにつれ、データの均一性や配布・利用の容易さが求められている。
生化学的、遺伝学的、ゲノム的に構造化されたゲノムスケールの代謝ネットワーク再構成の知識ベースであるBiGGについて説明する。BiGGは、複数のゲノムスケールの代謝ネットワークを標準的な命名法で1つのリソースに統合しており、異なる生物間で構成要素を比較することができるようになっている。BiGGは、モデルの内容を閲覧したり、代謝パスウェイマップを可視化したり、モデルのSBMLファイルをエクスポートして外部のソフトウェアパッケージで解析することができる。ユーザーは、BiGGからいくつかの外部データベースへのリンクをたどって、遺伝子、タンパク質、反応、代謝物、および関心のある引用文献に関する追加情報を得ることができる。
BiGGは、システム生物学のコミュニティにおいて、高品質な代謝モデルや再構成にアクセスしたいというニーズに対応している。BiGG は http://bigg.ucsd.edu から学術的に自由に利用できる。
Nucleic Acids Research, Volume 44, Issue D1, 4 January 2016
ゲノムスケールの代謝モデルは、代謝パスウェイの利用や成長表現型を予測するために使用できる、数学的に構造化された知識ベースである。さらに、実験データと統合することで、仮説を生成し、検証することができる。これらのモデルの価値を最大化するためには、高品質なモデルの集中リポジトリを確立し、確立された標準に準拠し、モデルの構成要素を関連するデータベースにリンクさせる必要がある。また、モデルを可視化するためのツールがあれば、モデルの有用性がさらに高まる。これらのニーズを満たすために、著者らは、生化学、遺伝学、ゲノムの知識ベースを完全に再設計したBiGG Models(http://bigg.ucsd.edu)を紹介する。BiGG Modelsには、75以上の高品質で手動で作成されたゲノムスケールの代謝モデルが含まれている。ウェブサイト上では、ユーザーはモデルを閲覧、検索、可視化することができる。BiGG Modelsは、ゲノムスケールのモデルをゲノムアノテーションや外部データベースに接続する。反応や代謝物の識別子は、コミュニティ標準に準拠し、モデル間での迅速な比較を可能にするために、モデル間で標準化されている。さらに、BiGG Modelsは、モデリングや解析ツールを用いてBiGG Modelsにアクセスするための包括的なアプリケーションプログラミングインターフェースを提供している。高度にキュレーションされ、標準化され、アクセス可能な代謝モデルのリソースとして、BiGG Modelsは、多様なシステムバイオロジー研究を促進し、多様な実験データの知識に基づいた解析をサポートする。
Nucleic Acids Research, Volume 48, Issue D1, 08 January 2020
BiGG Models knowledge base (http://bigg.ucsd.edu) は、高品質なゲノムスケールの代謝モデルの集中リポジトリである。過去12年間、このウェブサイトでは、ユーザーが代謝モデルを閲覧・検索できるようにしてきた。今回の更新では、各モデルをゲノムアノテーションや外部データベースに接続し、反応や代謝物の標準化を行うという当初からの取り組みを継続しながら、リポジトリの新しいコンテンツや機能を詳細に説明する。BiGG Modelsでカバーされている系統樹の部分を拡張する31の新しいモデルが追加されたことを説明する。また、多系統モデルをホストするための新しい機能についても説明する。最後に、ナレッジベースのモデルは、ゲノムスケールのモデルのためにコミュニティが開発した新しいバリデータであるMemoteを用いてベンチマークを行い、BiGG Modelsのモデルの品質と透明性が向上していることを示す。
http://bigg.ucsd.edu にアクセスする。
BiGG Modelsは、ゲノムスケールの代謝ネットワークを再構築したナリッジベースである。現在、BiGG Modelsは、108のゲノムスケールの代謝ネットワークが公開されており、BiGG識別子を用いて1つのデータベースに統合されている。BiGGモデルに含まれる遺伝子はNCBIゲノムアノテーションにマップされ、代謝産物は多くの外部データベース(KEGG、PubChemなど)にリンクされている。BiGGはこのウェブサイトで閲覧できる他、ウェブAPIを使ってアクセスすることもできる。ゲノムスケールの代謝ネットワーク再構成で査読付きジャーナルに掲載されていれば、自分のモデルをBiGGに追加することもできる。登録はトップページ右上のMemote Validator から行える(詳細についてはhelp参照)。
View Models
2021年1月現在、108生物の代謝モデルが掲載されている。
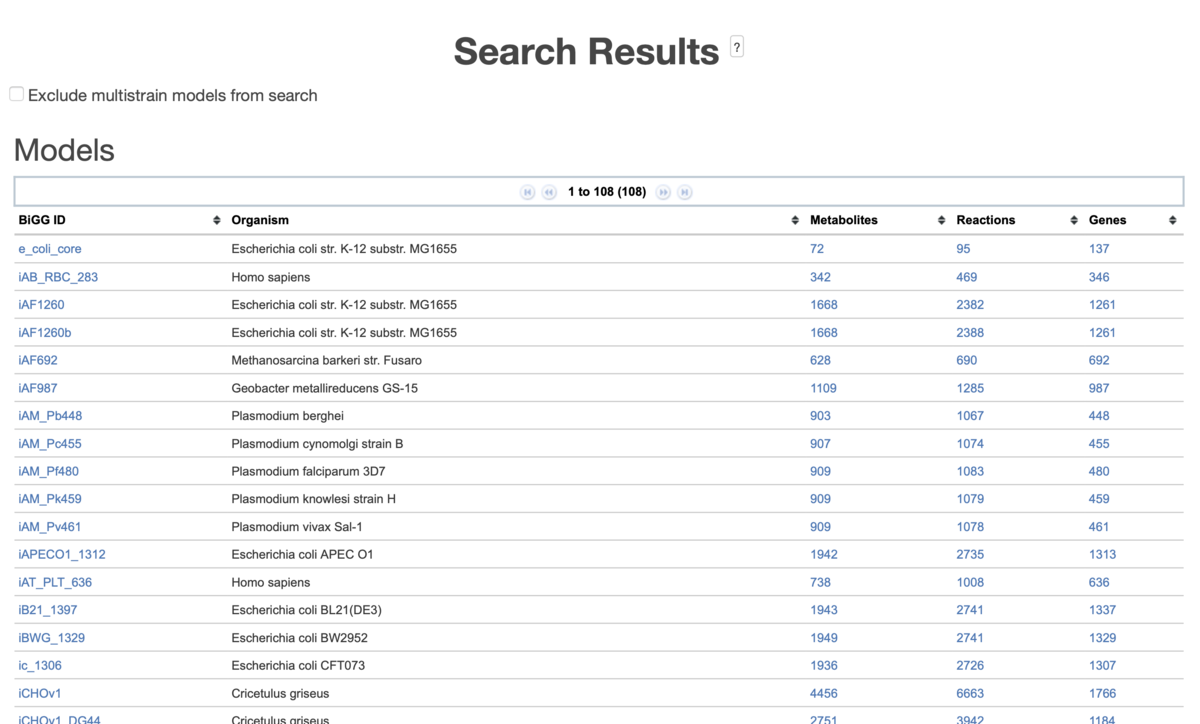
Escherichia coli str. K-12 substr. MG1655をクリックしてみた。

下には代謝モデル再構築のベースになった査読付き論文のリンクもある。
Metabolitesリンクを開いてみる。全部で72の代謝産物が登録されている。

代謝産物3-Phospho-D-glyceroyl phosphateをクリックした。その代謝産物の基本的な情報、前後の反応経路、そして右側には関連する外部リンクが表示される。

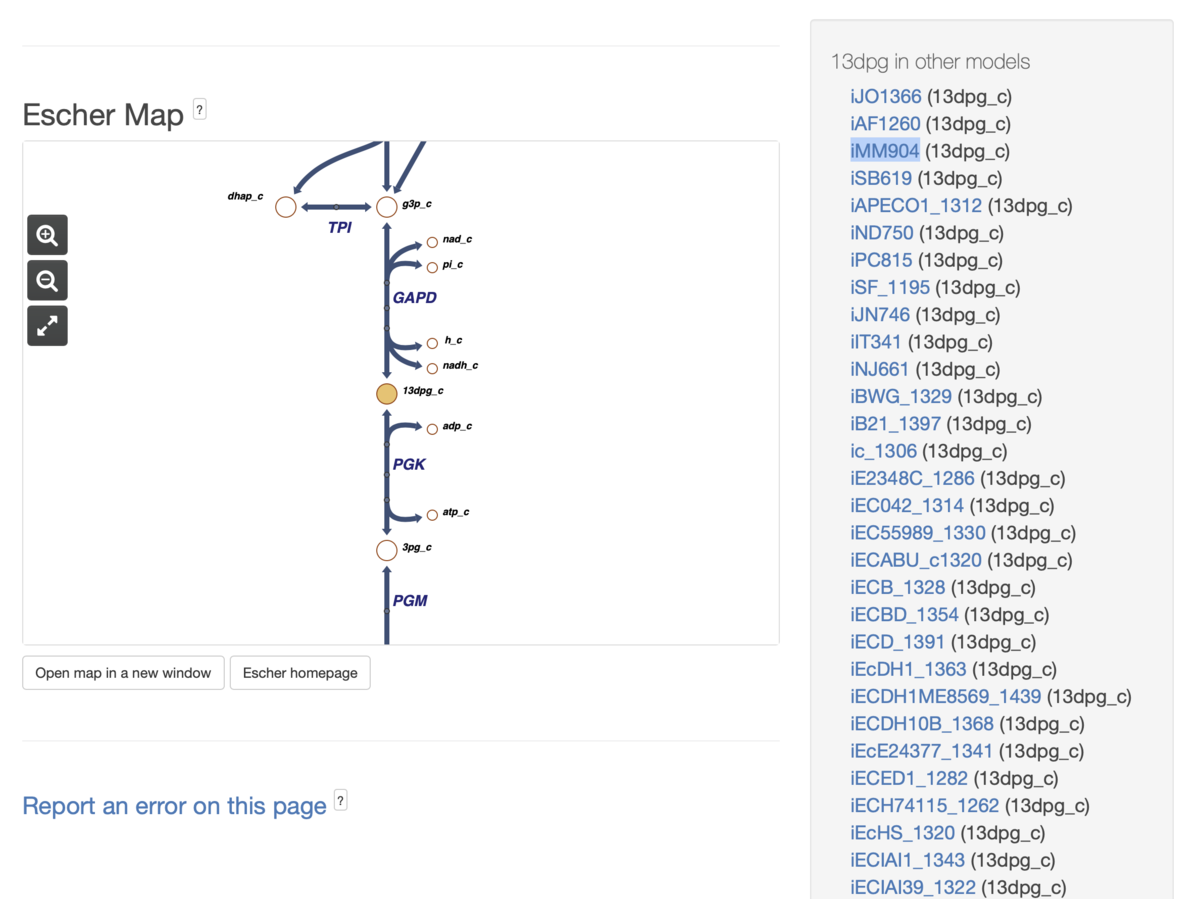
Topページからは代謝産物、または反応から検索を行うこともできる。
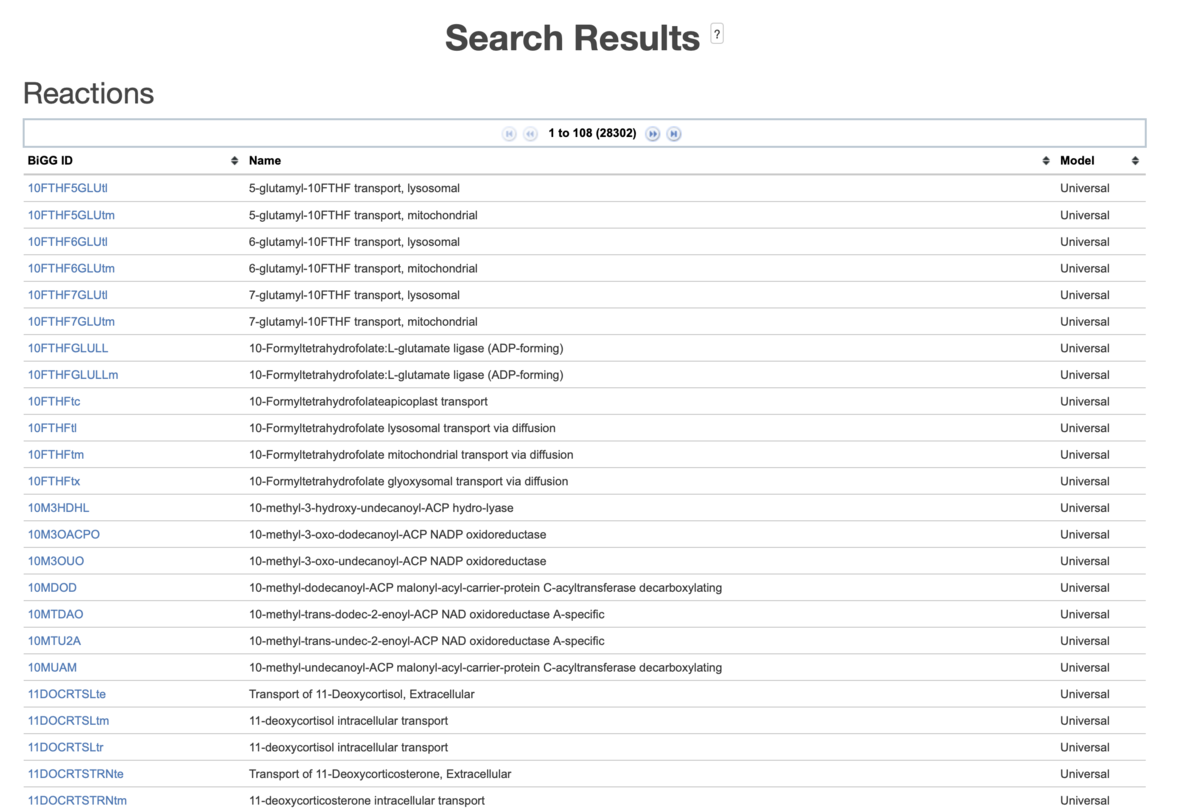
代謝産物は現在9088登録されている。

Reactionsは現在28302登録されている。
引用
BiGG Models 2020: multi-strain genome-scale models and expansion across the phylogenetic tree
Charles J Norsigian, Neha Pusarla, John Luke McConn, James T Yurkovich, Andreas Dräger, Bernhard Ø Palsson, Zachary King
Nucleic Acids Research, Volume 48, Issue D1, 08 January 2020, Pages D402–D406
BiGG Models: A platform for integrating, standardizing and sharing genome-scale models
Zachary A. King, Justin Lu, Andreas Dräger, Philip Miller, Stephen Federowicz, Joshua A. Lerman, Ali Ebrahim, Bernhard Ø Palsson, Nathan E. Lewis
Nucleic Acids Research, Volume 44, Issue D1, 4 January 2016, Pages D515–D522
BiGG: a Biochemical Genetic and Genomic knowledgebase of large scale metabolic reconstructions
Jan Schellenberger, Junyoung O Park, Tom M Conrad, Bernhard Ø Palsson
BMC Bioinformatics. 2010 Apr 29;11:213
関連
