2020 2/16 tweet追記、タイトル修正
pauvreはdarrin t schultzさんがGithubに公開されている第三世代ロングリード分析用のユーティリティツール。低クオリティなリードのフィルタリング機能も備えている。
I just updated the pauvre package to make some functions, like marginplot, work with the latest version of @matplotlib. More updates to documentation soon-to-come.
— Darrin Schultz (@conchoecia) 2020年2月15日
Here's a plot of some @PacBio IsoSeq data.https://t.co/jwAkJP6PGP #bioinformatics pic.twitter.com/XKDDqldPyH
インストール
mac os 10.12のminiconda3-4.0.5環境でテストした。
依存
- python 3.x
- matplotlib
- biopython
- pandas
- pillow
本体 Github
conda install -c bioconda -y pauvre
$ pauvre -h
usage: pauvre [-h] [-v] {marginplot,redwood,stats,synplot} ...
optional arguments:
-h, --help show this help message and exit
-v, --version Installed pauvre version
[sub-commands]:
{marginplot,redwood,stats,synplot}
marginplot plot a marginal histogram of a fastq file
redwood make a redwood plot from a bam file
stats outputs stats from a fastq file
synplot make a synteny plot from a gff file, protein
alignment, and partition file
実行方法
1、stats outputs stats from a fastq file
基本的なstatistics
pauvre marginplot --fastq input.fq.gz
Keeping reads that satisfy: length >= 0 and meanQual >= 0
# Fastq stats for SRR8182926_1.fastq.gz, reads >= 0bp
numReads: 38713
%totalNumReads: 100.00
numBasepairs: 215339875
%totalBasepairs: 100.00
meanLen: 5562.469325549557
medianLen: 4273.0
minLen: 38
maxLen: 80443
N50: 8860
L50: 7874
# Fastq stats for SRR8182926_1.fastq.gz, reads >= 1000bp
numReads: 32470
%totalNumReads: 83.87
numBasepairs: 211381859
%totalBasepairs: 98.16
meanLen: 6510.066492146597
medianLen: 5482.0
minLen: 1000
maxLen: 80443
N50: 9010
L50: 7653
この後も続く。
2、marginplot plot a marginal histogram of a fastq file
分析結果を可視化する。
pauvre marginplot -n --fastq input.fq.gz
テストラン
SRAから適当に3つ、SMSシーケンシングデータをダウンロードして分析してみる(parallel-fastq-dump紹介)。
#1 DNA sequencing of Ecoli strain H105 MinION (SRR8182926)
parallel-fastq-dump --sra-id SRR8182926 --threads 4 --gzip --outdir SRR8182926
#2 DNA sequencing of Ecoli strain H157 GridION (SRR8188010)
parallel-fastq-dump --sra-id SRR8188010 --threads 4 --gzip --outdir SRR8188010
#3 treptomyces sp. PAMC 26508 Pacbio CCS (SRR868759)
parallel-fastq-dump --sra-id SRR868759 --threads 4 --gzip --outdir SRR868759
Minion、GridION、Pacbio RS (CCSモード)のデータをダウンロードした。
可視化
#1 DNA sequencing of Ecoli strain H105 MinION (13 Nov 2018)
pauvre marginplot -n --fastq SRR8182926_1.fastq.gz

#2 DNA sequencing of Ecoli strain H157 GridION (14 Nov 2018)
pauvre marginplot -n --fastq SRR8188010.fastq.gz

#3 treptomyces sp. PAMC 26508 Pacbio CCS (27 Mar 2016)
pauvre marginplot -n --fastq SRR868759.fastq.gz
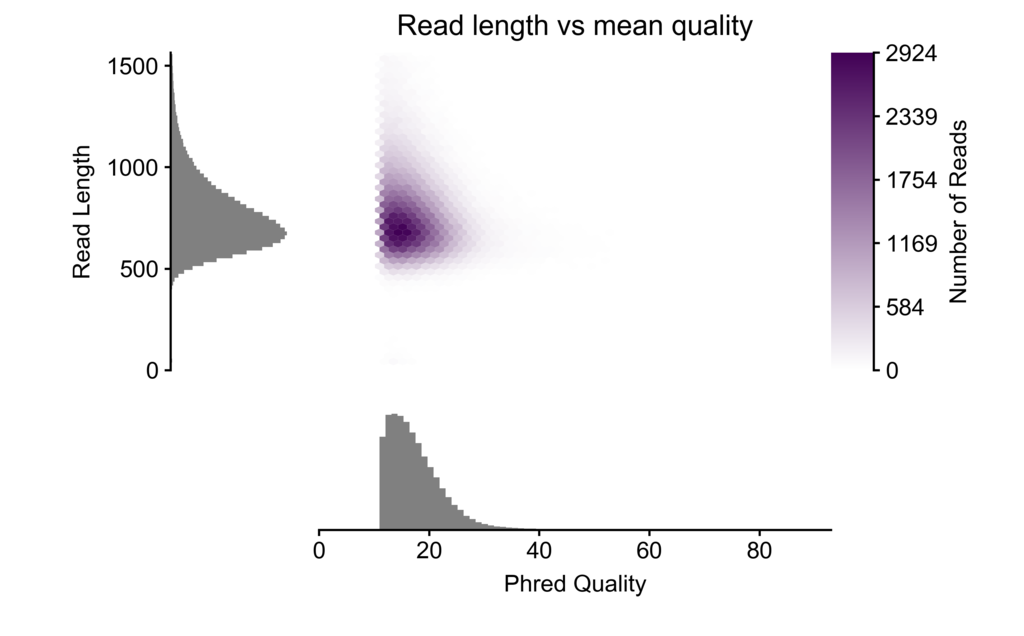 このようにシーケンス結果の外観をつかむことができる。縦軸、横軸の最大、最少はオプションで指定できます。一番下のSRR868759は、横軸の最大値は40で十分ですね。
このようにシーケンス結果の外観をつかむことができる。縦軸、横軸の最大、最少はオプションで指定できます。一番下のSRR868759は、横軸の最大値は40で十分ですね。
追記
フィルタリング機能も実装されています。Githubで確認してください。
引用