構造変異は遺伝病や進化のメカニズムにおいて重要な役割を果たしている。過去10年間、単純な構造変異を検出するために広範な研究が行われ、確立された検出方法が開発された。しかし、最近の研究では、単純な構造変異に比べて複雑な構造変異が個体に与える影響が大きい可能性があることが浮き彫りになっている。にもかかわらず、この分野には複雑な構造変異に特化した正確な検出法がまだない。したがって、非常に効率的で正確な検出法を開発することが最も重要である。このニーズに応えるため、本著者らはディープラーニング技術とコンセンサス配列を活用し、ロングリード配列データを用いたSVの検出を強化するFindCSVと呼ばれる新規手法を提案する。既存の手法と比較して、FindCSVは複雑な構造変異や単純な構造変異の検出において優れた性能を示した。FindCSVは、実データおよびシミュレーションデータにおいて、複雑で単純な構造変異を妥当な精度で検出する新しい手法である。このプログラムのソースコードはhttps://github.com/nwpuzhengyan/FindCSVにある。
Snifflesは現在最も人気のあるSV検出手法である。DeBreakとcuteSVは非常に高い再現率と精度を持つことが示されている効率的な手法である。SVcnnは本著者らが以前単純なSVを検出するのに用いた手法である。SVisionは第3世代のシーケンスデータでCSVを検出するために特別に使用された手法である。~
しかし現在のところcomplex SVs (CSVs)を正確に検出するための検出方法は不足している。実験の結果、CSV検出における2つの主要な課題が明らかになった: 1. CSVの正確な定義がないこと、2.CSVの多様な性質により、正しいブレークポイントを正確に特定し報告することが検出手法にとって困難であること。~
~ FindCSVは前のステップで得られた領域を画像に変換する。この画像表現では、各行はリードに対応し、各列は参照ゲノム上の位置に対応する。変換プロセスでは、CIGAR文字列の各文字を特定のルールに従ってピクセルに変換する。CIGAR文字列の各文字をルールに従ってピクセルに変換することで、SV候補領域内のリードのバリエーションとアラインメントパターンを視覚的に表現する5色の画像を作成する。INSは参照ゲノム上の位置を占めないため、FindCSVは、挿入長に基づいてマッチ(M)の一部を挿入(I)に置き換える。置換処理の後、領域は画像に変換され、SV画像と呼ばれる。~
FindCSVはSV候補領域のフィルタリングに畳み込みニューラルネットワークモデルを採用する。具体的には、FindCSVはフィルタリングモデルとしてLeNetモデルを選択する。LeNetモデルは3つの畳み込み層、2つのサブサンプリング層、2つの完全接続層から構成される。これらの層は入力データから特徴を抽出し、表現を学習するように設計されている。(以下略)
インストール
condaで環境を作り、pipでツールを導入した(GPU: RTX 3090)。
依存
1. python3
2. pysam
3. cigar
4. numpy
5. pyfaidx
6. copy
7. time
8. argparse
9. PIL
10. pytorch
11. torchvision
12. os
13. swalign
git clone https://github.com/nwpuzhengyan/FindCSV.git
cd FindCSV/
mamba create -n findCSV python=3.11 -y
conda activate findCSV
pip install pysam cigar numpy pyfaidx TIME-python argparse pillow torch torchvision os-sys swalign pycopy-copy
> python FindCSV.py -h
$ python FindCSV.py -h
usage: FindCSV.py [-h] bam fasta
positional arguments:
bam bam file
options:
-h, --help show this help message and exit
実行方法
ロングードのマッピングのbamとfastaファイルを指定する(アライナーはNGMLR、Minimap、Minimap2が想定されている)。bam.baiファイルも必要。fastaファイルは解凍されている必要がある。
python FindCSV.py input.bam reference.fasta
出力例(ref. : A.thaliana、SRAのONTデータの1つをダウンロードし、minnimap2の-x map-ontでマッピングして使用)
> head -n 20 FindCSV_result.vcf

FindCSVはSV領域を画像に変換し、その画像をSV_into_imageフォルダに格納する。各画像の名前は染色体名とSVの開始位置と終了位置からなる。
SV_image/

1_14866598-14867705.png

各リードが画像の1行を占める。黄色と青のピクセルはマッチ、黒のピクセルは欠失、赤のピクセルは挿入、緑のピクセルはミスマッチを表す。
1_13993090-13993201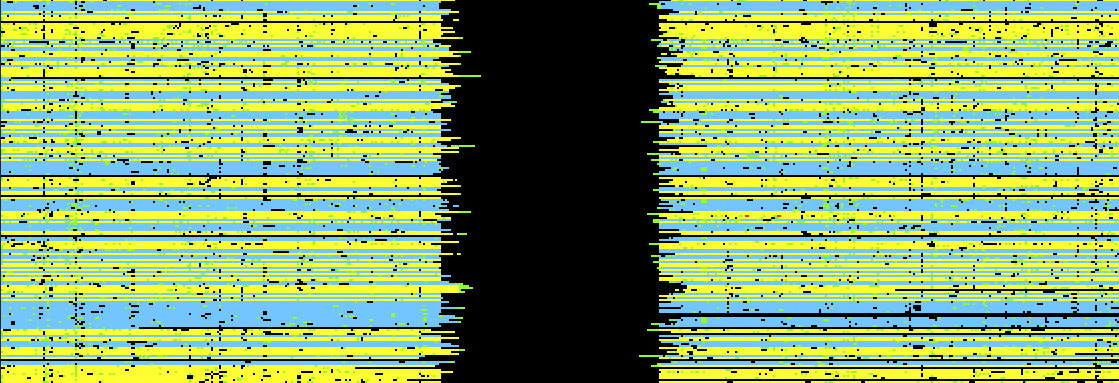
IGV
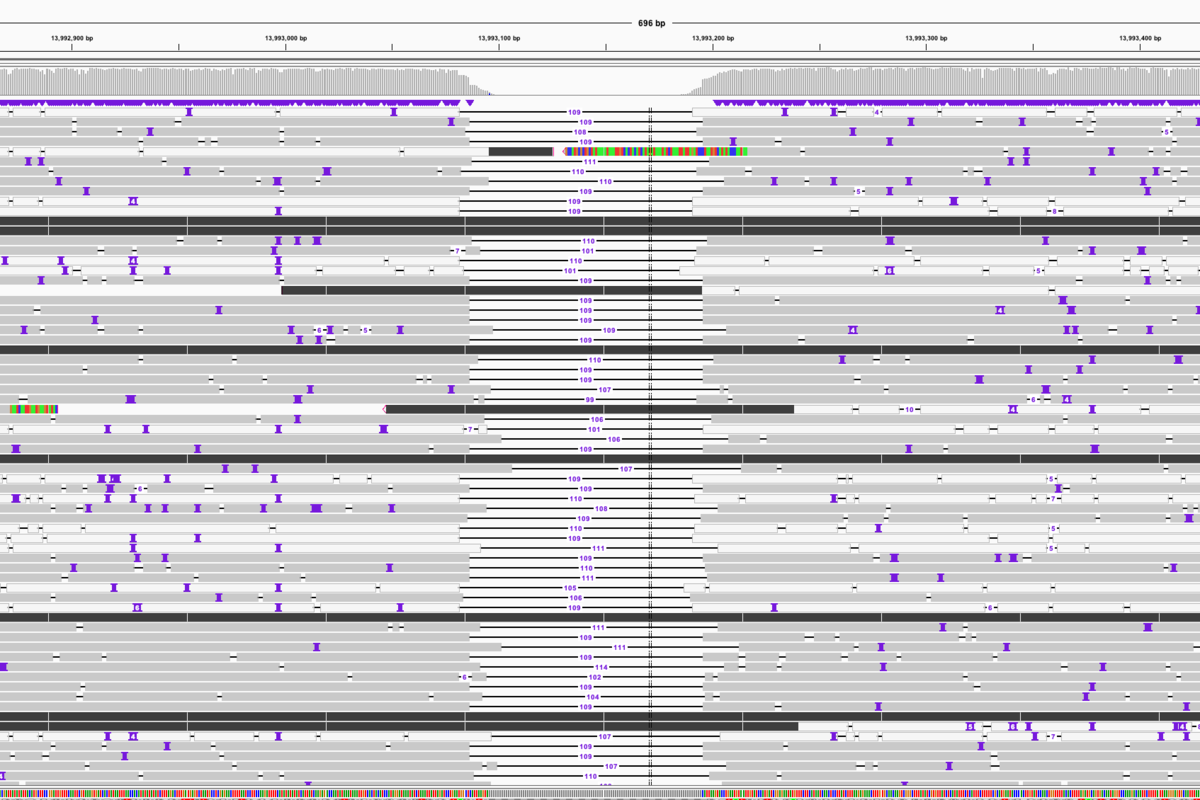
(少しずれているのはキャプチャの誤差による)
引用
FindCSV: a long-read based method for detecting complex structural variations
Yan Zheng & Xuequn Shang
BMC Bioinformatics volume 25, Article number: 315 (2024)