イルミナのショートリードのような第二世代のDNAシークエンシング技術は、ヒトゲノムのリシークエンシングを日常的なものにした(ref.1)。ヒトゲノムにおける最も豊富な変異タイプであるSNVとsmall indel変異の両方は、30〜40×の全ゲノムイルミナシークエンシングを使用して確実に検出することができる(ref.2; 3)。それにもかかわらず、ショートリードシークエンシング技術を用いたヒトゲノムのシークエンシングには多くの制限がある。第一に、ヒトは各常染色体の2コピー(母方および父方)を有する二倍体生物である。ハプロタイプ、または個々の染色体上に存在する対立遺伝子の配列は、複数のヘテロ接合型変異にわたるようなリードの重複を利用して計算により組み立てることができる(ref.4-6)。しかし、ヒトゲノムのヘテロ接合性が低いため(ref.7)、ショートリードライブラリ(長さ200〜500 bp)のペアエンドシークエンシングから得られたイルミナのリードは、通常1つの変異ポジションしかカバーせず、長い塩基配列を提供しない。第二に、ゲノムの約3.6%は、ショートリードを一意にマッピングすることができず、したがってSNVを検出することができない。これらは長く非常に類似した重複配列からなる。それらの領域は、PMS2やSTRCなどの多くの疾患関連遺伝子のコード領域とオーバーラップしている(ref.8)。
Pacific Biosciences(PacBio)やOxford Nanopore(ONT)などの第3世代の1分子シークエンシング(SMS)技術は、長いシーケンスリードを生成する。 PacBio1分子リアルタイム(SMRT)テクノロジの平均リード長は10〜30キロベースである(ref.9)。これらのロングリードは、ハプロタイプ決定および構造変化検出を含むショートリード技術で制限があった事象を克服する可能性を有する。実際、SMSデータは、ヒトゲノムの新規構築(ref.10-11)、ヒトゲノムの複雑な構造変異(ref.12)およびハプロタイプ構築(ref.10, 13)の同定に首尾よく使用されてきた。しかし、イルミナのようなショートリードシーケンシング技術と比較して、SMSリードの1塩基当たりのエラー率は10%を超える(主にindelエラーによる)(ref.14)。この高いエラー率は、SNVなどの小さな配列変異、特にヘテロ接合変異の検出を困難にする。
SMS技術のコストの減少およびヒトゲノムシーケンシングへの使用の増加に伴って、SMSデータのための正確なバリアントコール方法は多くの点で価値があり得る。Genome in a Bottle(GIAB)コンソーシアム(ref.15,16)によって発展した、ヒトゲノムバリアントコールの現在のベンチマークは、ショートリードシーケンシングデータに基づいており、リファレンスヒトゲノム配列の〜90.8%をカバーしている。これらの信頼性の高いバリアントコールは、新しいバリアントコール方法およびシーケンシング技術を開発するために非常に貴重である。しかしながら、これらのバリアントコールセットは、ショートリードを用いてコールしやすい(easy-to-call)ゲノムの領域にバイアスしている(ref.17)。ロングリードSMSデータを使用した正確なSNVコールは、ショートリードSNVコールの独立した検証を提供することでfalse positivesを減らし、体系的なエラーやアーティファクトについての理解を深めることができる。さらに、SMSリードを用いたSNVコーリングは、セグメント重複を含むゲノムのrepetitive領域における高信頼性バリアントコールの生成を可能にし得る。ショートリードシークエンシング技術ではアクセスできないrepetitive領域のバリアントをコールする能力も、重複遺伝子における疾患原因変異検出のためのSMS技術の使用をadvanceさせることができる(ref.18)。
SMSリードからのHaplotype-resolved SNV detectionはまた、ハプロタイプを用いたリードの分離を介した構造変異(SV)のような他の種類のヒト遺伝性変異の発見も可能にし得る。ハドルストン et al(ref.19)は2つのhaploid全ゲノムPacBioデータから何千ものSVを同定するためにアセンブリベースのアプローチ、SMRT-SVを使用した。その89%は1000ゲノムプロジェクト(ref.20)によって報告されていなかった。しかしながら、SMRT-SVを用いたSV検出感度は、二倍体ゲノムにおいてわずか41%であった。 Chaisson et alは、複数のシークエンシング技術を用いてヒトゲノムの高密度全ゲノムハプロタイピングを行い、haplotype-separatedされたSMSリードの各グループのSVをうまくコールすることができた(ref.21)。
ショートリードデータ解析用に開発されたGATK HaplotyperCaller(ref.22)やFreebayes(ref.23)などのバリアントコーリングツールは、次の2つの理由で、PacBioデータを使用したSNV検出には適していない。(i)ショートリードのエラー率は低く(<0.5)、これらの方法はSMSリードの高いindelエラー率をモデル化していないため、真のSNVをエラーと区別することを困難にし、そして(ii)これらの方法はshort windows(典型的には数百塩基)でリードを分析し、SMSリードに存在するハプロタイプ情報をレバレッジするようには設計されていない。シーケンシングエラーは分離する可能性が低いが、真のバリアントはそれが生じるハプロタイプに由来するリードで分離するので、このハプロタイプ情報は真のバリアントをエラーと区別するのに非常に貴重であり得る。最近、ロングリードからのバリアントコールおよびディープラーニングベースのバリアントコーリング方法のためのいくつかの方法が開発された(ref.24-26)。しかしながら、これらの方法のSMSデータからSNVをコールする精度は、現在、イルミナの全ゲノムシーケンシング(WGS)を用いたものよりもはるかに低い(ref.25, 26)。
本著者らはSNV検出とハプロタイピングを共同で実行するためにロングSMSリードを利用する二倍体SNVコール方法、Longshotについて説明する。このために、Longshotは我々(本著者ら)のリードベースのハプロタイプフェージング方法HapCUT2(ref.13)を使用する。 SMSリードの高いエラー率を克服するために、それは、ローカルアラインメントにおける不確実性を平均し、遺伝子型尤度を計算するために使用することができる正確なbase quality valuesを推定するためにペア隠れマルコフモデルを利用する。我々(本著者ら)は、シミュレーションデータおよびPacBio SMRTおよびOxford Nanoporeシーケンシング技術を使用してシーケンシング(ref.15, 16, 27)した複数のヒト個体について、Longshotのベンチマークを行った。
Figure 1: Overview of the Longshot algorithm. Preprintより転載
Longshotに関するツイート
インストール
ubuntu16.04でテストした(ホストOS macos10.14)。
依存
Longshot has been tested using Ubuntu 16.04 and 18.04, CentOS 6.6, Manjaro Linux 17.1.11, and Mac OS 10.14.2 Mojave. It should work on any linux-based system that has Rust and Cargo installed.
- rust >= 1.30.0
- zlib >= 1.2.11
- xz >= 5.2.3
- clangdev >= 7.0.1
- gcc >= 7.3.0
- libc-dev
- make
- various rust dependencies (automatically managed by cargo)
#install dependencies
sudo apt-get update && sudo apt-get install cargo zlib1g-dev xz-utils libclang-dev clang build-essential curl git
biocondaでも間も無く導入できるようになるとアナウンスされている。現在でもレシピをダウンロードしてローカルでビルドできる。手順はGithubで確認してください。
git clone https://github.com/pjedge/longshot
cd longshot
cargo install --path .
# add cargo binaries to path
export PATH=$PATH:~/.cargo/bin/
> ~/.cargo/bin/longshot -h
# ~/.cargo/bin/longshot -h
Longshot 0.3.2
Peter Edge <edge.peterj@gmail.com>
SNV caller for Third-Generation Sequencing reads
USAGE:
longshot [FLAGS] [OPTIONS] --bam <BAM> --ref <FASTA> --out <VCF>
FLAGS:
-A, --auto_max_cov Automatically calculate mean coverage for region and set max coverage to mean_coverage +
5*sqrt(mean_coverage). (SLOWER)
-S, --stable_alignment Use numerically-stable (logspace) pair HMM forward algorithm. Is significantly slower but
may be more accurate. Tests have shown this not to be necessary for highly error prone
reads (PacBio CLR).
-F, --force_overwrite If output files (VCF or variant debug directory) exist, delete and overwrite them.
-x, --max_alignment Use max scoring alignment algorithm rather than pair HMM forward algorithm.
-n, --no_haps Don't call HapCUT2 to phase variants.
-h, --help Prints help information
-V, --version Prints version information
OPTIONS:
-b, --bam <BAM> sorted, indexed BAM file with error-prone reads
-f, --ref <FASTA> indexed FASTA reference that BAM file is aligned to
-o, --out <VCF> output VCF file with called variants.
-r, --region <string> Region in format <chrom> or <chrom:start-stop> in which to call variants
(1-based, inclusive).
-p, --hap_bam_prefix <BAM> Write haplotype-separated reads to 3 bam files using this prefix:
<prefix>.hap1.bam, <prefix>.hap2.bam, <prefix>.unassigned.bam
-c, --min_cov <int> Minimum coverage (of reads passing filters) to consider position as a
potential SNV. [default: 6]
-C, --max_cov <int> Maximum coverage (of reads passing filters) to consider position as a
potential SNV. [default: 8000]
-q, --min_mapq <int> Minimum mapping quality to use a read. [default: 30]
-a, --min_allele_qual <float> Minimum estimated quality (Phred-scaled) of allele observation on read to
use for genotyping/haplotyping. [default: 7.0]
-y, --hap_assignment_qual <float> Minimum quality (Phred-scaled) of read->haplotype assignment (for read
separation). [default: 20.0]
-Q, --potential_snv_cutoff <float> Consider a site as a potential SNV if the original PHRED-scaled QUAL
score for 0/0 genotype is below this amount (a larger value considers
more potential SNV sites). [default: 20.0]
-e, --min_alt_count <int> Require a potential SNV to have at least this many alternate allele
observations. [default: 3]
-E, --min_alt_frac <float> Require a potential SNV to have at least this fraction of alternate
allele observations. [default: 0.125]
-L, --hap_converge_delta <float> Terminate the haplotype/genotype iteration when the relative change in
log-likelihood falls below this amount. Setting a larger value results in
faster termination but potentially less accurate results. [default:
0.0001]
-l, --anchor_length <int> Length of indel-free anchor sequence on the left and right side of read
realignment window. [default: 6]
-m, --max_snvs <int> Cut off variant clusters after this many variants. 2^m haplotypes must be
aligned against per read for a variant cluster of size m. [default: 3]
-W, --max_window <int> Maximum "padding" bases on either side of variant realignment window
[default: 50]
-I, --max_cigar_indel <int> Throw away a read-variant during allelotyping if there is a CIGAR indel
(I/D/N) longer than this amount in its window. [default: 20]
-B, --band_width <Band width> Minimum width of alignment band. Band will increase in size if sequences
are different lengths. [default: 20]
-D, --density_params <string> Parameters to flag a variant as part of a "dense cluster". Format
<n>:<l>:<gq>. If there are at least n variants within l base pairs with
genotype quality >=gq, then these variants are flagged as "dn" [default:
10:500:50]
-s, --sample_id <string> Specify a sample ID to write to the output VCF [default: SAMPLE]
--hom_snv_rate <float> Specify the homozygous SNV Rate for genotype prior estimation [default:
0.0005]
--het_snv_rate <float> Specify the heterozygous SNV Rate for genotype prior estimation [default:
0.001]
--ts_tv_ratio <float> Specify the transition/transversion rate for genotype grior estimation
[default: 0.5]
-P, --strand_bias_pvalue_cutoff <float> Remove a variant if the allele observations are biased toward one strand
(forward or reverse) according to Fisher's exact test. Use this cutoff
for the two-tailed P-value. [default: 0.01]
-d, --variant_debug_dir <path> write out current information about variants at each step of algorithm to
files in this directory
テストラン
ランにはロングリードマッピングのbam、リファレンスのfastaが必要。
cd longshot/example_data/
longshot --bam pacbio_reads_30x.bam --ref genome.fa --out longshot_output.vcf
出力(VCF4.2)
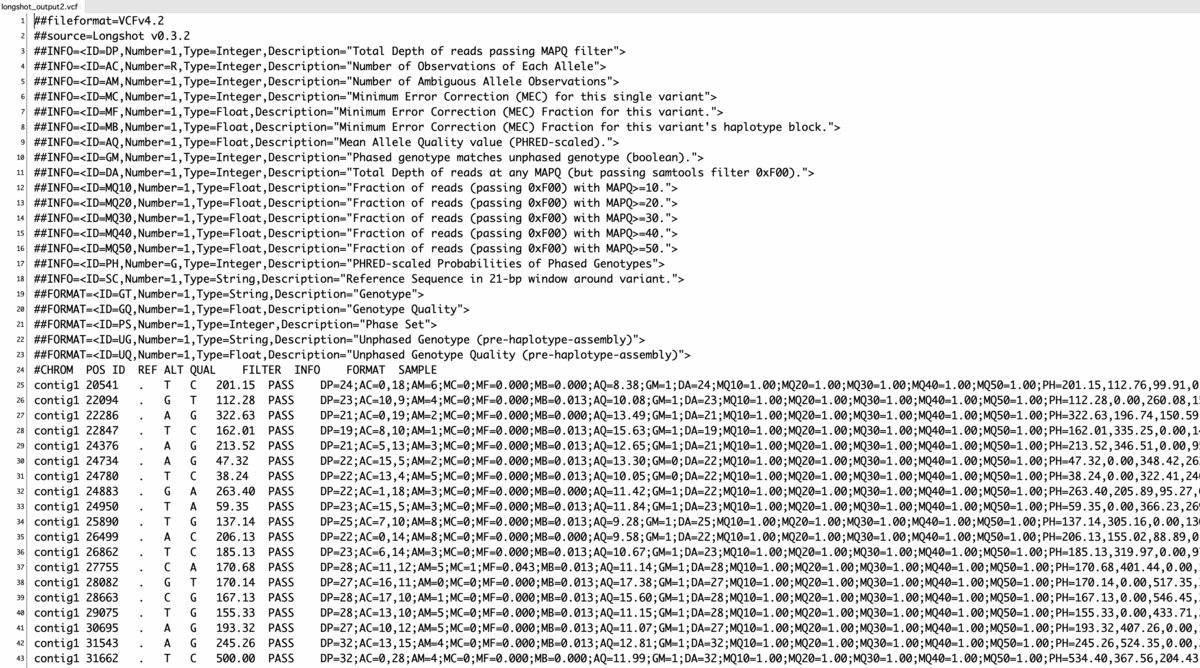
他の実行例
chr1に限定してバリアントコールを行う(*1)。auto_max_coverageも設定する。
longshot -A -r chr1 --bam pacbio.bam --ref ref.fa --out output.vcf
- -A Automatically calculate mean coverage for region and set max coverage to mean_coverage + 5*sqrt(mean_coverage). (SLOWER)
chr1:1000000-1500000に限定してバリアントコールを行う。ハプロタイプ分離したリードをreads.hap1.bam、reads.hap2.bam、reads.unassigned.bamの3つに分けて出力する。
longshot -r chr1:1000000-1500000 -y 30 -p reads --bam pacbio.bam --ref ref.fa --out output.vcf
- -y <float> Minimum quality (Phred-scaled) of read->haplotype assignment (for read
separation). [default: 20.0] - -p <BAM> Write haplotype-separated reads to 3 bam files using this prefix:
<prefix>.hap1.bam, <prefix>.hap2.bam, <prefix>.unassigned.bam
vcfの他に、 reads.hap1.bam、 reads.hap2.bam、reads.unassigned.bamが出力される。
実行前にGiithubの"important considerations"も目を通しておいて下さい。
引用
Longshot: accurate variant calling in diploid genomes using single-molecule long read sequencing
Peter Edge, Vikas Bansal
bioRxiv preprint first posted online Mar. 1, 2019
*1
chromosomeごとに分けてランすることが推奨されている。
