パスウェイ解析はオミックス研究で広く用いられている。パスウェイベースのデータ統合と可視化は、解析の重要な要素である。このニーズに応えるために、本著者らは、Pathviewという新しいRパッケージを開発した。Pathviewは、多種多様な生物学的データを分子パスウェイグラフにマッピングし、統合し、レンダリングする。パスウェイの視覚化とデータ統合を、特別なコンピューティングスキルやリソースを持たない科学者も含めて、すべての科学者が利用できるようにするために、Pathview Webサーバーを開発した。Pathview Webは、直感的なグラフィカルウェブインターフェイスとユーザー中心のデザインを特徴としている。このサーバーは、Pathviewのコア機能を拡張するだけでなく、オフラインのRパッケージでは利用できない多くの便利な機能を提供する。重要なのは、複数のオミックスデータの通常のパスウェイ解析と統合パスウェイ解析の両方に対応する包括的なワークフローを提供することである。また、プログラムによるアクセスやサードパーティのソフトウェアやワークフローへの便利な統合のためのRESTful APIも提供している。Pathview Webは、https://pathview.uncc.edu/ で公開されており、自由に利用することができる。
help
https://pathview.uncc.edu/tutorial?instruction_flag=T
Introduction
https://pathview.uncc.edu/overview#kegg_view
https://pathview.uncc.edu/homeにアクセスする。
1、タブまたはカンマ区切りのデータマトリックスファイル;Gene Dataファイルを用意する必要がある。このファイルは、1列目は遺伝子ID(ENTREZ Gene, Symbol, RefSeq, GenBank Accession Number, UNIPROT, Enzyme Accession Number、KEGGなど)、1列目はサンプルIDとする。2列目、2行目以降は発現量のvalue。
Gene Dataの例(Example 1)

準備できたらアップロードする。

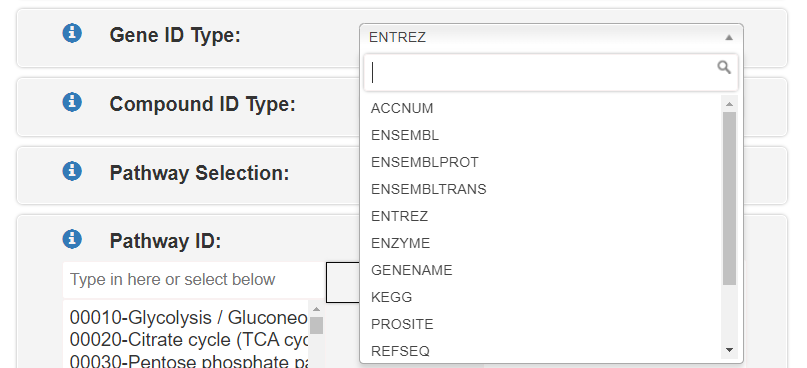
続いてGene ID TypeでGene IDの種類を指定する。上の例はENTREZ。
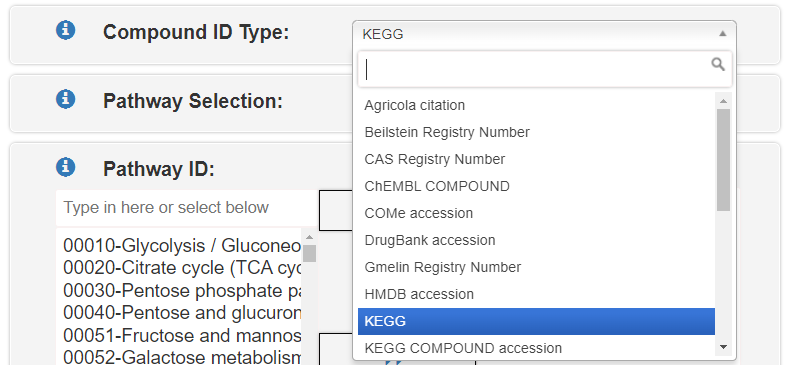
2、任意でタブまたはカンマ区切りのデータマトリックスファイル;Compound Dataを用意すれば、パスウェイの酵素だけでなく、代謝化合物もマッピングされる。また、Compound Dataも提供した場合、各データセットに対して個別にパスウェイ解析が行われ、その結果がグローバル統計値/P値にまとめられる(論文図2B)。Compound Dataのデータマトリックスファイルの1列目は代謝物、薬物、低分子などの化合物IDで、1列目はサンプルIDになる。

続いてCompound ID TypeでCompound IDの種類を指定する。
Speciesを選択する(KEGG pathway IDのorg部分、ヒトはhsa)

submitボタンをクリックすると解析がスタートする。
出力 (example1)
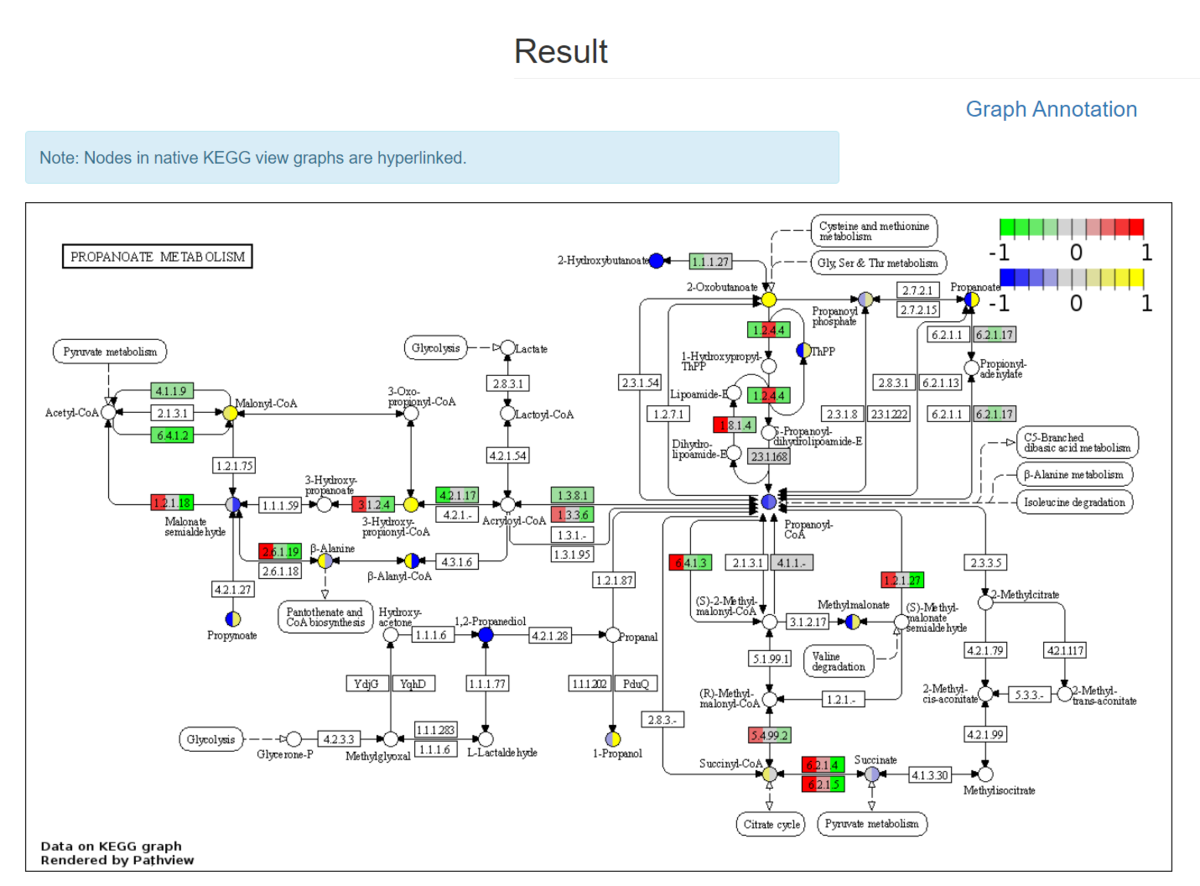
ユーザーのデータがマッピングされたすべてのノード(分子、遺伝子/タンパク質/酵素、代謝物/化合物)は、グラフ上で色付けされ、ハイライトされる。上の図では、青い〇が代謝化合物で、緑のボックスは酵素タンパク質になる。これらのノードは、KEGGの参照ページにハイパーリンクされている。
引用
Pathview Web: user friendly pathway visualization and data integration
Weijun Luo, Gaurav Pant, Yeshvant K. Bhavnasi, Steven G. Blanchard, Jr, Cory Brouwer
Nucleic Acids Research, Volume 45, Issue W1, 3 July 2017, Pages W501–W508