ClinQCは、SangerおよびNGSシーケンシングデータの品質管理、フィルタリングおよびトリミングの統合パイプライン。臨床研究で数百から数千のサンプル/患者に対する サンガーのシーケンシングとNGSデータを分析し、サンプル/患者ごとに統一された分析レポートを出力する。一度に大量のサンプル(illumina、サンガー、pacbioなど)を解析して、結果を統合して出力できるのが本ツールの特徴といえる。
まず、ClinQCは、入力ファイルをFASTQフォーマットに変換し、アダプターとPCRプライマーを除去する。 次に、バーコード化されたサンプルを分離し、重複をフィルタリング、コンタミネーションおよび低品質のシーケンスを生成し、QCレポートを生成する。
マニュアル
https://sourceforge.net/p/clinqc/wiki/ClinQC_Manual/
ダウンロード
依存
- Python 2.7.9
- Biopython 1.60 or higher
- Bioperl 1.6 or higher
- Perl 5.10 or higher
- Java 1.7 or higher
- AlienTrimmer ftp://ftp.pasteur.fr/pub/gensoft/projects/AlienTrimmer/
- TraceTuner https://sourceforge.net/projects/tracetuner/
- FASTQC http://www.bioinformatics.babraham.ac.uk/projects/fastqc/
- PRINSEQ http://sourceforge.net/projects/prinseq/files/standalone/
Dockerイメージでも提供されている。ここではDockerイメージをダウンロードしてランする。
Dockerイメージのダウンロード
https://sourceforge.net/p/clinqc/wiki/Virtual_Machine/
分かりにくいがリンク先のウィンドウ内左上 "Download ClinQC VM"をクリック。VirtualBoxを持ってない人は、ClunQCのマニュアルに従いOracle VM VirtualBoxと Extension Packを導入しておく。
立ち上げる(起動前に、余裕があればメモリとCPUの数は増やしておく)。

cd ~/ClinQC_v1.0
ラン
configファイルに従い解析が行われる。コンフィグファイルとして、ClinQCOptions_Sanger、ClinQCOptions_NGS、ClinQCOptions_NGS_pacbioが用意されている。
また、サンプルの情報が記載されたターゲットファイルもランには必要となる。テストではsanger_target_file.txtとなっている。テストのsanger、illumina、pacbioそれぞれに保存されている。下のようなフォーマットとなる。

右端から2番目のカラムにサンガーシーケンスデータのパスが記載されている。
テストのサンガーシーケンスデータを解析する。
./clinqc --option-file ClinQCOptions_Sanger
テストでは2検体の解析となる。ME9は5、MEN1は36のサンガーシーケンスの統合結果となる。

ME9のクオリティフィルタリング前。クオリティはつかない。

クオリティフィルタリング後。
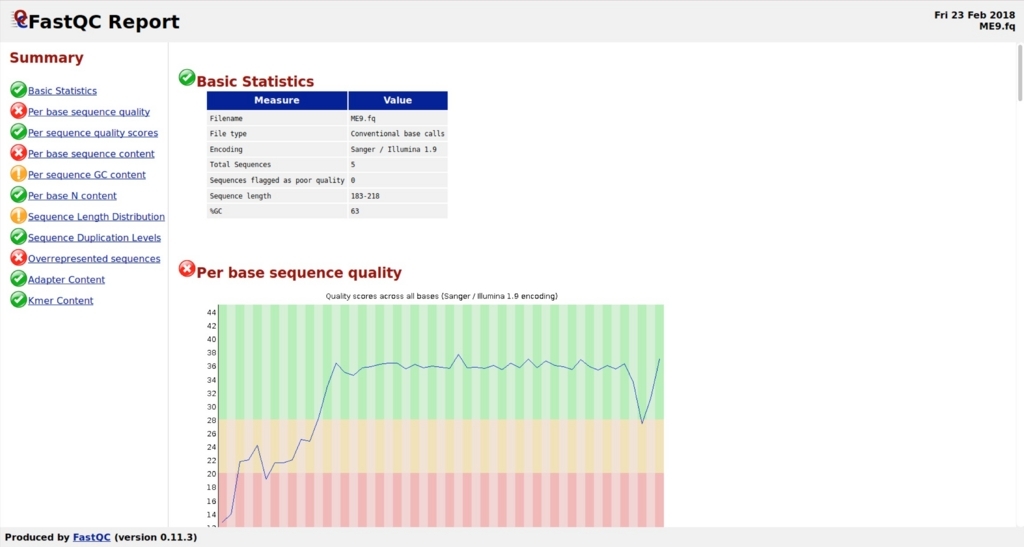
fastqで配列も出力される。

引用
ClinQC: a tool for quality control and cleaning of Sanger and NGS data in clinical research.
BMC Bioinformatics. 2016 Feb 2;17:56.
Pandey RV, Pabinger S, Kriegner A, Weinhäusel A.