伝統的なウイルス同定法は単離および培養に依存しており、時間がかかるだけでなく、多くのウイルスおよび宿主が培地で増えないため実行不可能なことも多い。 2004年に登場したNGSの技術により、ウイルスとその存在量を迅速に測定することが可能になった。ウィルスをNGSデータから分析、同定するには、準備したウィルスデータベースとリードを比較するのが理想である。MetaPhlAn2<該当記事>は、リードを100万を超えるマーカー遺伝子のデータベースにアライメントしてバクテリア、ウイルスを同定する。しかしながら、このような戦略は、クエリの配列が既知マーカー遺伝子とヒットしない新規ウィルスでは機能しない。コンティグに組み立てて長い配列で taxonomic analysis を行うとより正確な結果が得られる傾向にあるので( 論文より Roux et al、2017 )、contigからvirusを検出する方法もあるが、それにはメタゲノムデータからウィルスゲノムを正確にアセンブルする必要がある。highly polymorphicなvirusゲノムのアセンブルではキメラの可能性が増大するので、正確な分類定量は簡単ではない(精度はアセンブルツールに大きく依存する)。
FastViromeExplorerは、メタゲノムデータから迅速かつ正確にウイルス検出と定量を行うスタンドアロンのパイプライン。 RNAseqのアライメントと定量のために開発された擬似アライメントベースのkallisto(紹介)を使い、ショートリードをウイルスデータベースにマッピングする。次に、FastViromeExplorerは最小のカバレッジ基準に基づいてアライメント結果をフィルタリングし、ウイルスの種類と存在量をアノテーションとともに出力する( kallistoを使い、k -mer(デフォルト31 bp)が正確に一致するデータを検索している)。
インストール
依存
- java1.8
javaの複数バージョン切り替えはこの辺り(リンク)を参考にしてください。java以外のsamtoolsやkallistoなどの依存はダウンロードに含まれています。
プロジェクトページ
https://code.vt.edu/saima5/FastViromeExplorer
git clone git@code.vt.edu:saima5/FastViromeExplorer.git
cd FastViromeExplorer/
cd FastViromeExplorer/tools-mac/
#samtools(1.5)とkallisto(0.43.1)のパスを通す
sudo cp kallisto /usr/local/bin/
sudo cp samtools /usr/local/bin/
source ~/.bash_profile
#またはこのディレクトリにパスを通す
echo export PATH=\$PATH:`pwd`\ >> ~/.bash_profile && source ~/.bash_profile
cd FastViromeExplorer_roor_dir/ #ルートに戻る
javac -d bin src/*.java
java -cp bin FastViromeExplorer #ヘルプ
> java -cp bin FastViromeExplorer
$ java -cp bin FastViromeExplorer
Usage:
java -cp bin FastViromeExplorer -1 $read1File -2 $read2File -i $indexFile -o $outputDirectory
-1: input .fastq file for read sequences (paired-end 1), mandatory field.
-2: input .fastq file for read sequences (paired-end 2).
-i: kallisto index file, mandatory field.
-db: reference database file in fasta/fa format.
-o: output directory. Default option is the project directory.
-l: virus list containing all viruses present in the reference database along with their length.
-cr: the value of ratio criteria, default: 0.3.
-co: the value of coverage criteria, default: 0.1.
-cn: the value of number of reads criteria, default: 10.
-salmon: use salmon instead of kallisto, default: false. To use salmon pass '-salmon true' as parameter.
-reportRatio: default: false. To get ratio pass '-reportRatio true' as parameter.
ランにはkallistoのindexファイルが必要で、ある程度のvirusについてはオーサーが準備してくれている。公式のリンク先から"ncbi-virus-kallisto-index-k31.idx"をダウンロードする。4GBある。また、そこにはファージやeukaryotic-virusのkallistoインデックスファイルもある。必要に応じてダウンロードする。公式リンク→
https://code.vt.edu/saima5/FastViromeExplorer
ラン
付属のペアエンドデータをランする。
mkdir test-output
java -cp bin FastViromeExplorer -1 test/reads_1.fq -2 test/reads_2.fq -i test/testset-kallisto-index.idx -o test-output
- -1 input .fastq file or .fastq.gz file for read sequences (paired-end 1), mandatory field.
- -2 input .fastq file or .fastq.gz file for read sequences (paired-end 2).
- -i kallisto index file, mandatory field.
- -o output directory, default option is the project directory.
テストではNCBI Reseqに登録された100程度のvirusを調べているだけなので、5-10秒以内に解析は終わる。出力のabundanceファイルを確認する。
less test-output/FastViromeExplorer-final-sorted-abundance.tsv
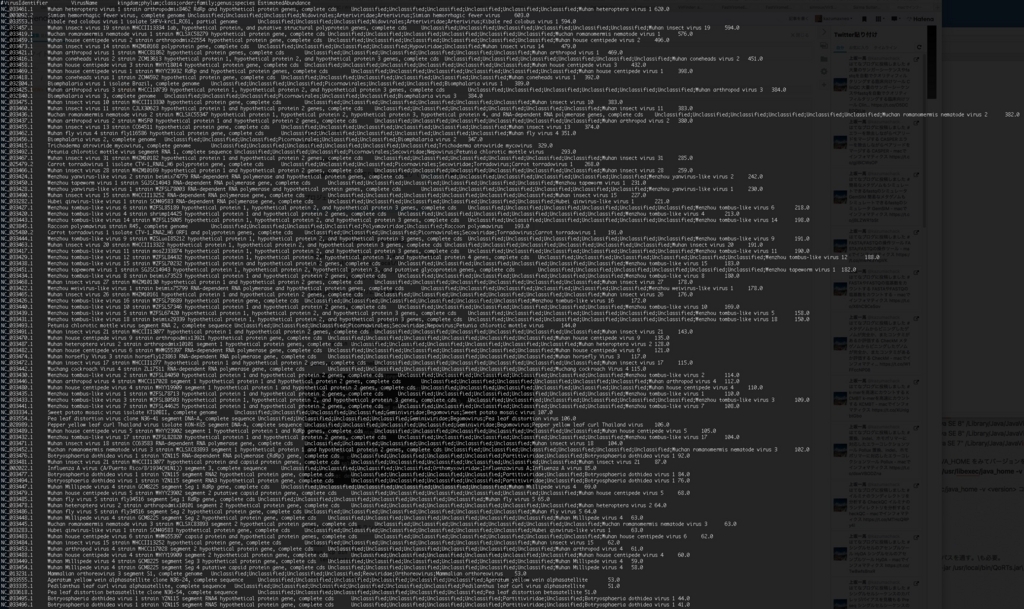
右端のカラムにestimate abundanceの数値が記載されている。
公式ページでは、IMG/VRデータベース(pubmedリンク)のindexを使ってランする方法や、カスタムデータベースを使う方法(virusのfastaファイルとテキストファイルからビルドする)も記載されています。
引用
FastViromeExplorer: a pipeline for virus and phage identification and abundance profiling in metagenomics data.
Tithi SS1, Aylward FO2, Jensen RV2, Zhang L.
PeerJ. 2018 Jan 12;6:e4227.