葉緑体ゲノムの完全長配列は、種の進化の歴史に関する豊富な情報を提供している。次世代シークエンシング技術の進展に伴い、葉緑体ゲノムの完全配列の数は飛躍的に増加すると予想されており、ゲノム配列をアノテーションするための強力な計算機ツールが急務となっている。
著者らは、ウェブサーバCPGAVASを開発した。このサーバーは、葉緑体ゲノムの全塩基配列を入力として受け付ける。まず、Blastx, Blastn, protein2genome, est2genomeプログラムの結果を統合し、最も類似した完全長のタンパク質、cDNA、rRNA配列の同定とマッピングに基づいて、タンパク質をコードする遺伝子とrRNA遺伝子を予測する。第二に、tRNAscan、ARAGORN、vmatchを用いて、tRNA遺伝子とインバーテッドリピート(IR)をそれぞれ同定する。第三に、アノテーションされたゲノムのサマリー統計量を計算する。第四に、公開可能な環状マップを生成する。第五に、GenBankにサブミットするためのSequinファイルを作成することができる。最後に、与えられた遺伝子と種のリストに対して、タンパク質とmRNAの配列を抽出することができる。GFF3形式のアノテーション結果は、互換性のあるアノテーション編集ツールを使用して編集することができる。編集したアノテーションはCPGAVASにアップロードして、更新や再解析を繰り返すことができる。既知の葉緑体ゲノム配列をテストセットとして用いた結果、CPGAVASは他のアプリケーションであるDOGMAと比較しても遜色のない性能を有していること、また、いくつかの優れた機能を有していることを示した。
CPGAVASは、葉緑体ゲノムの配列を半自動で完全にアノテーションし、アノテーション結果を可視化、編集、解析することができます。葉緑体ゲノムを研究する研究者には欠かせないツールとなるでしょう。本ソフトウェアは、http://www.herbalgenomics.org/cpgavas から自由にアクセスできる。
help
http://47.90.241.85:16019/analyzer/documents
tutorial PDF
http://47.90.241.85:16019/documents/cc.pdf
mirror1:http://47.96.249.172:16019/analyzer/home にアクセスする。
mirro2:http://47.90.241.85:16019/analyzer/home
chloroplastのFASTAファイルを指定する。ここではexample dataを使う。メールアドレスを記載しておくとラン後にメールが届く。

追加パラメータ

Submitボタンを押して実行する。
出力
結果を表示するにはランIDを入力する。

GFF3形式の遺伝子モデル、png形式の環状マップ、FASTA形式の予測されたDNAやタンパク質の配列、解析レポートテーブルが出力される。
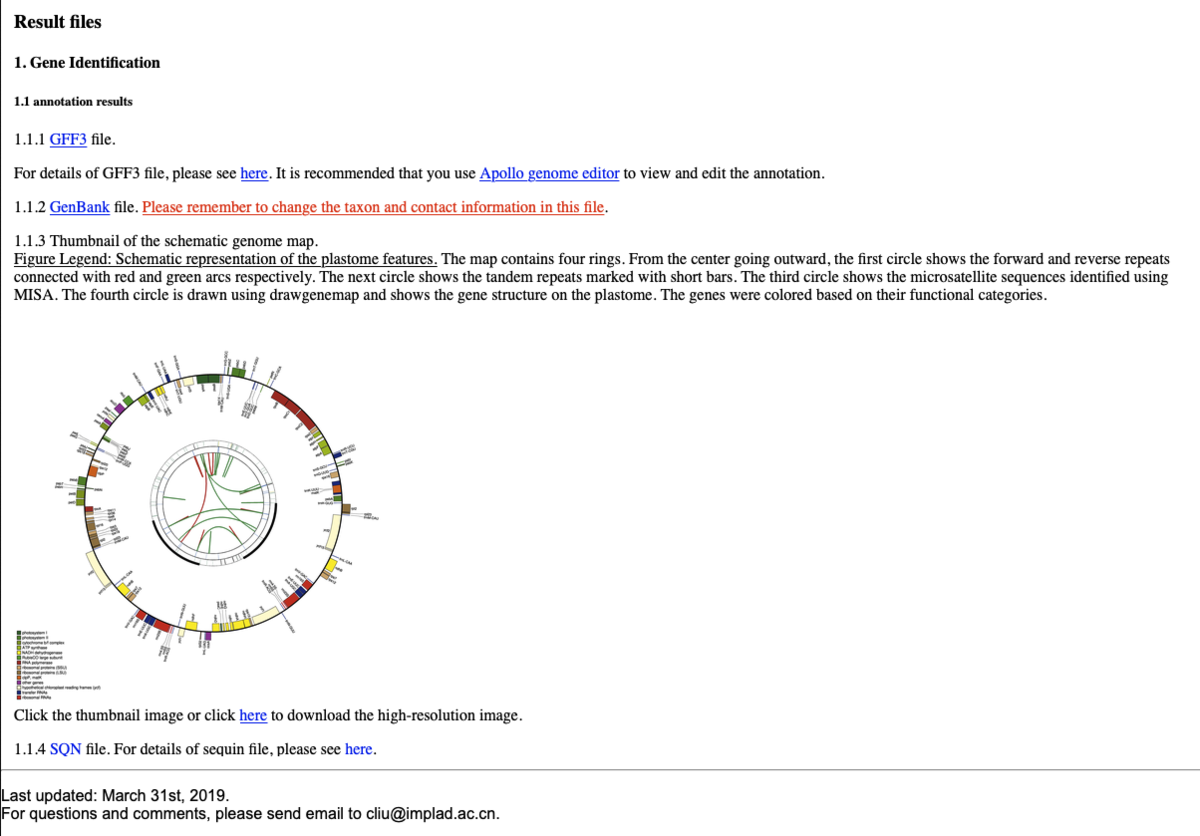
クリックすると拡大(SVG)

また、NCBI GenBank sumission用にsequinのSQNファイルもダウンロードできる。
結果の視覚化、および編集にはApollo genome editorの使用が推奨されている。
http://47.90.241.85:16019/analyzer/view
引用
CpGAVAS, an integrated web server for the annotation, visualization, analysis, and GenBank submission of completely sequenced chloroplast genome sequences
Chang Liu, Linchun Shi, Yingjie Zhu, Haimei Chen, Jianhui Zhang, Xiaohan Lin & Xiaojun Guan
BMC Genomics volume 13, Article number: 715 (2012)
関連
