生物の分子進化の歴史を理解するには、通常、近縁種や系統のゲノム領域を視覚的に比較する必要がある。このタスクを達成するためのアプリケーションはすでにいくつか存在するが、それらは古すぎたり、限定的すぎたり、あるいは複雑すぎたりして、ほとんどのユーザーのニーズには合わない。GenoFigは、原核生物のゲノム領域を視覚化するためのグラフィカルなアプリケーションであり、可能な限り使いやすく、様々なニーズに適応できる柔軟性を意図している。GenoFigは、正規表現を用いて、GenBankファイルから抽出されたアノテーションを、配列間で一貫した方法でパーソナライズされた形で表現することができる。また、配列間の相同領域の表示を最適化するユニークなオプションや、配列のGCパーセンテージやGC-skew表現のような、より古典的な機能も提供する。要約すると、GenoFigは、原核生物における特定のゲノム領域の進化を探索し、出版に耐えうる図を作成するための、シンプルで、無料で、高度に設定可能なツールである。Genofigは、GPL 3.0ライセンスのもと、https://forgemia.inra.fr/public-pgba/genofigで利用できる。
インストール
ubuntu22でcondaで環境を作ってテストした。レポジトリではリリース1.1のWindows版とMacOS版のコンパイル済みバージョンのダウンロードリンクも用意されている。
git clone https://forgemia.inra.fr/public-pgba/genofig.git
cd genofig/
#linux
mamba env create -f extras/requirements.yml
conda activate genofig
extras/SETUP.sh #link
#windows (WSLではない)
mamba env create -f extras/requirements_windows.yml
#パスを通す
export PATH="'$(pwd)':$PATH"
#or,
echo 'export PATH="'$(pwd)':$PATH"' >> ~/.bashrc && source ~/.bashrc
> Genofig
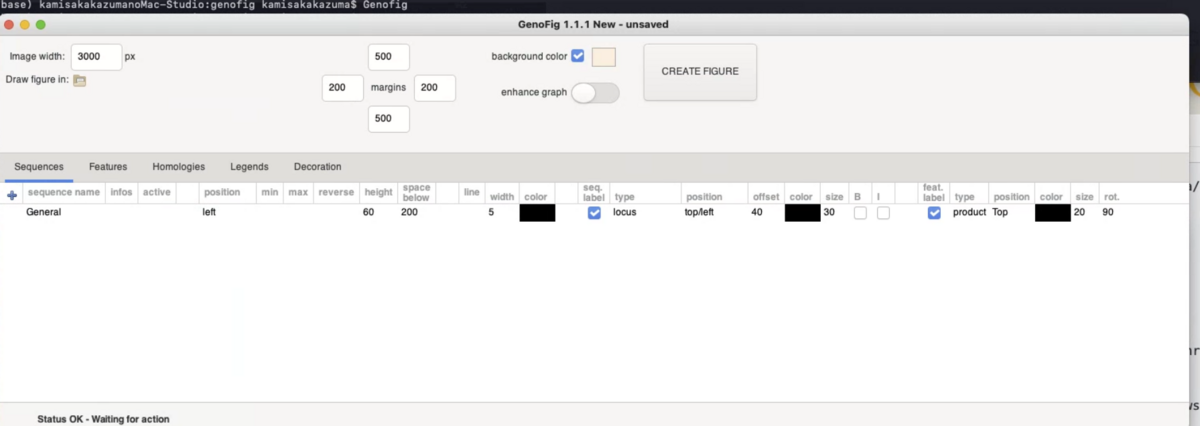
GenoFigはGenBankフォーマットのアノテーションを使う。GenoFigは小さなゲノム領域(せいぜい数百Kbp)を比較するように設計されているので、ゲノム全体を読み込むには向いていない。数個の遺伝子や遺伝子クラスターなどを可視化する。
チュートリアルではNCBI nucleotideから遺伝子フィーチャーを可視化して、比較したい領域をGenBankでダウンロードしている。
GenBankは左端のプラスのボタンから読み込む。

CREATE FIGURE ボタンを押すと視覚化されてSVG形式で保存される。

アノテーションがついているCDSは灰色のボックスでプロットされる。例外として、アノテーションが hypothetical proteinsは白色のボックスでプロットされる。
特定のCDSだけ色を変更できる。Featureタブに移動し、プラスマークで新しいfeatureを追加、type列で色を変更したいCDSのアノテーション名(同じ名前だと一括して適用される)を入力し、productを選択、さらに右の方のcolorで色を指定する。

視覚化すると色が変更された。フィルターは正規表現をサポートしているので、柔軟に特定のfeatureだけ強調したりできる。

レポジトリで紹介されている代表的な機能を見ていく。
配列タブでreverseにチェックを入れると全ての矢印の向きが反転する(全体が反転するのではなく、個々のfeatureが180度回転する)。

SequencesタブのBとIをチェックして左端のラベルを太字と斜体にする。

Sequencesタブの左端のラベルの色を緑にして、さらにtypeをorganismにして生物名がラベルされるようにする。

Sequencesタブの左端のラベルのサイズを20に下げる。

Sequencesタブには、他にもラベルの位置やサイズ、幅などを細かく調整できるようになっている。
ラベルがはみ出さないように、上のパネルで左側のマージンを400に増やす。

2つ目の配列を追加した。

ラベルが被っている。ほとんどのフィーチャーは同じなので、Sequencesタブの右端近くのfeat.label列のGeneralのチェックをはずし、最初の配列にだけチェックを入れる。

一番上のgenbank配列にのみアノテーションが表示された。
Featuresタブの右端の方のlabel列のチェックを特定のフィーチャー以外は外した。

Homologies パネルでRun blastnを実行し、それから描画した。

配列間の相同なCDSがシンテニーで示される(すべての配列間でBLASTnが実行されるため配列数が多い時は注意)。画像では、上下は同じ配列のため、全領域にシンテニーが見つかっている。
レジェンドタブで凡例が表示されるようにする。サイズはスケール係数で、場所はpositonで指定できる。

カスタムした設定はsaveから保存できるが、 そのApplicationメニューが正しく表示されないという問題が発生しているらしい。試した時は表示されなかった(M1 macでrosetta2使用)。
レポジトリでは情報が整理されてずっと綺麗な作図がされています。確認して下さい。
引用
GenoFig: a user-friendly application for the visualisation and comparison of genomic regions
Maxime Branger, Sébastien O Leclercq
Bioinformatics, Published: 13 June 2024
関連
・遺伝子クラスターを比較してインタラクティブな図で視覚化する clinker
・