系統(種、属、高次のクレード)における遺伝子の非消耗性は、多くのゲノムシーケンスプロジェクトがパンゲノームレベルに移行するにつれて重要性を増している。
ほとんどの解析では、遺伝子を、調査対象となったすべてのゲノムに存在する「コア遺伝子」と、調査対象となった一部のゲノムにしか存在しない「ディスパーサブル(使い捨て)遺伝子」に分類している。コア遺伝子とディスパーサブル遺伝子の分類は、解析対象ゲノムにおける存在/非存在の任意のカットオフ値に基づいていることが多い。
QUODでは、遺伝子をコアまたはディスペンサブルに分類する代わりに、各遺伝子にディスペンサブルスコアを付与する。QUODは、様々な環境条件に対する系統特異的な適応の背景となっている可能性のある、ディスパーサブル可能な遺伝子の候補を特定するのに役立つ。
インストール
python3.9の環境(2020年頃)を作ってテストした(* 最近のバージョンだとエラーが発生する)。
依存
- argparse, pandas, numpy (optional: matplotlib for visualization)
mamba create -n QUOD python=3.9 -y
conda activate QUOD
mamba install pandas numpy matplotlib -y
mamba install -c bioconda samtools -y
#本体
git clone https://github.com/ksielemann/QUOD.git
cd QUOD/
> python QUOD.py
'--input_dir' was not set'
python3 QUOD.py
--input_dir <FULL_PATH_TO_FOLDER_INPUT_BAM_FILES> (file names = sample names)
--bam_is_sorted <PREVENTS_EXTRA_SORTING_OF_BAM_FILES> (optional argument)
--gff <FULL_PATH_TO_REFERENCE_ANNOTATION_FILE>
--output_dir <FULL_PATH_TO_OUPUT_FOLDER>
--min_cov_per_genome <INTEGER> (default = 10, optional argument)
--visualize (optional argument)
REQUIREMENTS: os, glob, sys, argparse, pandas, numpy (optional: matplotlib for visualization)
テストラン
実行するには、調査対象の同種の別系統の株のゲノムのシークエンシングリードを、dispensable genesを調査したい株のリファレンスゲノム配列にマッピングする。これらのマッピングから得られたBAMファイルをQUODにかけ非常に稀な遺伝子を検出する。
Arabidopsis thaliana Nordborgセットからランダムに選ばれた4つのアクセッションのゲノムリードが用意されている。
https://pub.uni-bielefeld.de/record/2946079からbamとgff3をダウンロードする。
wget https://pub.uni-bielefeld.de/download/2946079/2946082/AthNd1_v2c_chr1_1mb.gff3
wget https://pub.uni-bielefeld.de/download/2946079/2946083/SRR1945627.bam.gz
wget https://pub.uni-bielefeld.de/download/2946079/2946084/SRR1945757.bam.gz
gunzip SRR1945627.bam.gz SRR1945757.bam.gz
mkdir bam_dir
samtools sort SRR1945627.bam > bam_dir/SRR1945627.bam
samtools sort SRR1945757.bam > bam_dir/SRR1945757.bam
#準備ができたら実行する
python3 QUOD.py --in bam/ --bam_is_sorted --gff AthNd1_v2c_chr1_1mb.gff3 --out output_QUOD/ --visualize
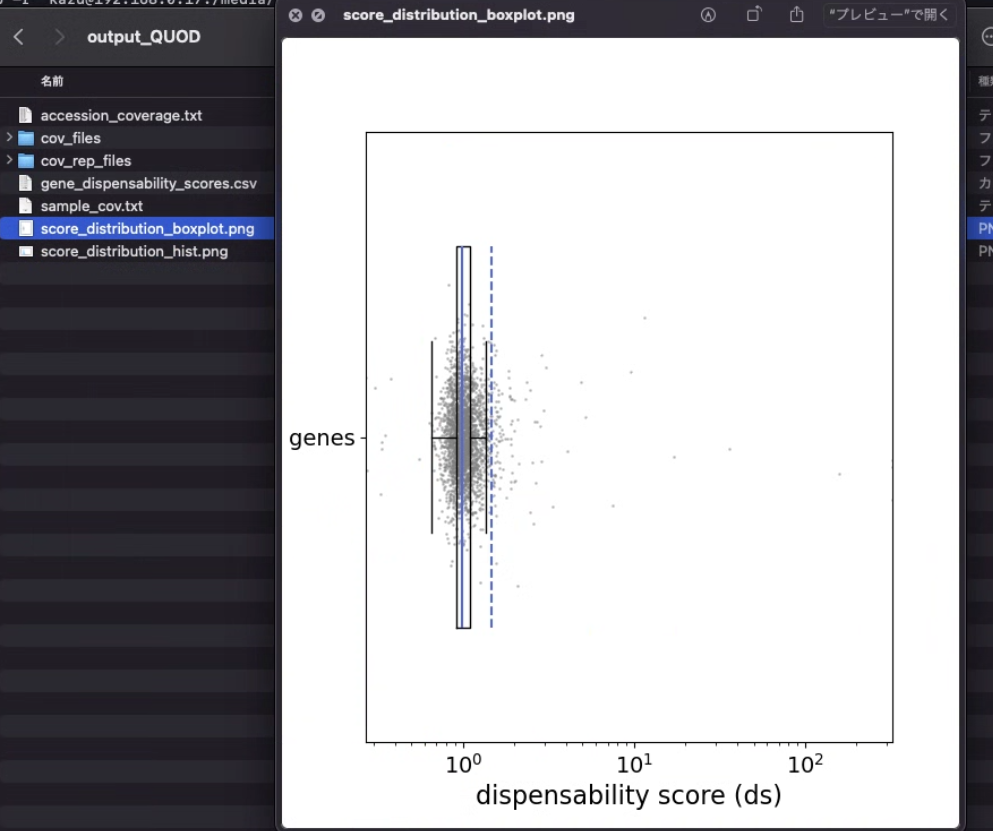
各遺伝子について、リードマッピングの正規化カバレッジに基づく遺伝子特異的dispensability scoreが計算される。
出力例

score_distribution_hist.png

gene_dispensability_scores.csv

引用
Reference-based QUantification Of gene Dispensability (QUOD)
Katharina Sielemann, Bernd Weisshaar & Boas Pucker
Plant Methods volume 17, Article number: 18 (2021)
関連