オーソロガス遺伝子は、タンパク質または機能的RNA分子をコードする核酸のセクションであり、単一の祖先遺伝子から派生し、その後に種分化により分岐している[ref.1、2]。対照的に、パラロガスな遺伝子は、単一の種内の重複から生じたものである。 OrthoDB [ref.3]、Eggnog [ref.4]、InParanoid [ref.5]、およびOrthologous Matrix(OMA)プロジェクト[ref.6]を含む、所定のオルソログを探索する多数のリポジトリが利用可能である。オーソロガス遺伝子は、アセンブリまたはアノテーションの完全性を評価し、遺伝子機能を予測/推測するために、および2つ以上の種間の系統解析の前駆体として、新たにアノテーション付けされたゲノムから新たに同定することもできる[ref.7、8、9]。オルソログを予測するための多くのツールと方法が開発されてきた。たとえば、タンパク質のペアワイズBasic Local Alignment Search Tool(BLAST)[ref.10]からの相互のベストヒットを介して開発されている。 :InParanoid [ref.11]またはOrthoMCL [ef.12]。大規模な遺伝子ファミリー、低品質のアノテーション、および/またはアセンブリーはそれぞれ、オルソログ予測の精度に寄与する要因として特定されている[ref.13]。オルソログの予測は、DAGchainerツール[ref.14]などによって、連続したチェーンに含まれる予測を特定することにより、さらに洗練されている。
オルソログを使用して、シンテニーの証拠を提供できる。2つの個体または種の間の染色体上の遺伝子座の順序の保存である。シンテニック領域の視覚化は、genome expansions[ref.15]や染色体転座[ref.16]などの進化過程の検出と表示に役立つ。さらに、水平方向の遺伝子導入を特定するために、シンテニーの欠如が使用されてきた[ref.17]。ゲノムアセンブリの汚染または不正確さも、たとえばシンテニーのレベルが低い場合や、そうでなければclosely relatedな単離株の染色体リアレンジメントが豊富な場合に検出される場合がある。これらのプロセスを検出する他の方法には、Dot Plots[ref.18]、またはMummer [ref.19]やThreaded Blockset Aligner(TBA)[ref.20]などのグローバルアラインメント検索ツールがある。ただし、これらの方法は本質的に遺伝子中心ではなくゲノムであるため、種全体の遺伝子含有量の変化を特定したり、誤ったオロソログまたはゲノムアセンブリを生物学的変異と区別するための追加作業が必要である。
Syntenyの視覚化は、Sybil / Sybillite [ref.21]などの一連のソフトウェアスイートおよびツールに実装されている。Sybil/ Sybilliteは、オーソロガス遺伝子のクラスターに基づいていくつかのゲノムを検索および視覚化するコマンドラインおよびWebツールである。別の一般的なシンテニー視覚化ツールはCircos [ref.22]で、ゲノムをサークルとして描き、保存または相互作用の領域間に弧を描く。要件、データ入力、および必要な視覚化の種類の違いにより、比較ゲノムでの使用には依然として追加のツールが必要だが、既存のツールでは多くの場合、新機能の追加の開発と保守とエラー修正が必要である。
ここでは、Synteny Imager(Synima)という名前のPerlベースのツールを使用して、2つ以上のゲノム間で予測されるオルソログのチェーンを視覚化する。 Synimaは、DAGchainer出力ファイルに含まれるオルソログデータを読み取り、PDFで各ゲノムの染色体と遺伝子間の位置と関係を視覚化するRscriptを生成及び 起動する。染色体および/または最大3つの別々の遺伝子カテゴリーは、最初の実行からコマンドラインで指定するか、Synima構成ファイルで指定することにより、Synimaの1回の実行でオプションで強調表示できる。 Synimaはhttps://github.com/rhysf/Synimaから無料で入手できる。 Synimaは、さまざまなプロジェクト[ref.16、23、24、25]で正常に使用されたコードに取って代わるもので、種間および種内でのゲノムの類似性と進化的変化の定量と提示を促進する。Synimaは、それぞれ1,720〜1,830万塩基の最大12ゲノムを含む一連のデータセットで開発およびテストされている。
Newest version of Synima has options to predict orthogroups with Orthofinder. pic.twitter.com/Gy9qPd96AB
— Rhys Farrer (@RhysFarrer1) November 1, 2018
インストール
依存
BioPerlはCPANからダウンロードしてビルドする(or apt-get install bioperl
)。legacy blastのformatdb(現在のmakeblastdb)とblastallコマンドにもパスが通っている必要がある。"conda install -c bioconda -y blast-legacy"で入れるか、biocondaのプリビルドバイナリをfetchして来てもいい(2.2.6 link)。
apt update
apt install bioperl
apt install Math::Round
#Rがないなら導入
apt install r-base
本体 Github
git clone git@github.com:rhysf/Synima.git
cd Synima/
$ perl SynIma.pl
Usage: perl SynIma.pl -c <config.txt> or -a <aligncoords> -b <aligncoords.spans>
Commands: -c Config.txt [/Users/kazu/Documents/Synima-master/SynIma-output/config.txt]
-a Aligncoords
-b Aligncoords.spans
Optional: -e Genome FASTA filename extension (e.g. /Users/kazu/Documents/Synima-master/genome1/genome1.genome.fa etc.) [genome.fa]
-t Aligncoords.spans 2
-u Aligncoords.spans 3
-k Gene IDs 1 (1 per line)
-l Gene IDs 2 (1 per line)
-o Gene IDs 3 (1 per line)
-r Run full program (y) or just create config (n) [y]
-v Verbose output (y/n) [n]
Plot Opts: -i Width of figure in pixels [1100]
-j Height of figure in pixels (num of genomes * 100)
-g Fill in chromosome/contig synteny (c) or gene synteny (g) [c]
-z Plot individual genes (y/n) [n]
-x Order of genomes from bottom to top seperated by comma
-n Genome labels from bottom to top seperated by comma
-w number of lines for left hand margin [12]
Notes: Config.txt will be made automatically if not present, and read automatically if it is.
Config.txt specifies order of genomes, chromosomes, colours, and other plot options.
Config.txt can be manually edited after creation.
Default genome labels will be as they appear in aligncoords
Order of genomes must have names as they appear in aligncoords
Aligncoords.spans and Gene ID files will be highlighted according to the config
Citation: When publishing work that uses Synima please cite:
Farrer RA (2017), BMC Bioinformatics 18:507
> perl util/Create_full_repo_sequence_databases.pl -h
$ perl util/Create_full_repo_sequence_databases.pl -h
Usage: perl util/Create_full_repo_sequence_databases.pl -r <Repo_spec.txt>
Optional: -f Feature wanted from GFF [mRNA]
-s Seperator in GFF description for gene names (" ; etc) [;]
-d GFF description part number with the parent/gene info [0]
-m Remove additional comments in column [ID=]
Notes: Will copy all transcripts and specified features from GFF into primary fasta files
>perl ../util/Blast_grid_all_vs_all.pl -h
$ perl ../util/Blast_grid_all_vs_all.pl -h
Usage: perl ../util/Blast_grid_all_vs_all.pl -r <Repo_spec>
Optional: -t Type of alignment (PEP/CDS) [PEP]
-c Number of best matches to capture between species [5] # only single best hit
-s Number of top hits to capture in self-searches for paralogs [1000]
-e E-value cutoff [1e-20]
-o Blast cmds outfile [blast.$type.cmds]
-g Run commands on the grid (y/n) [n]
-p Platform (UGER, LSF, GridEngine) [UGER]
-q Queue name [short]
Note: BLAST legacy (formatdb and blastall) need to be in PATH (use BLAST)
> perl util/Blast_all_vs_all_repo_to_Orthofinder.pl
$ perl util/Blast_all_vs_all_repo_to_Orthofinder.pl
Usage: perl ../util/Blast_all_vs_all_repo_to_Orthofinder.pl -r <Repo_spec>
Optional: -t Type (PEP/CDS) [PEP]
-o Out directory [Orthofinder_outdir]
> perl util/Blast_all_vs_all_repo_to_RBH.pl
$ perl util/Blast_all_vs_all_repo_to_RBH.pl
Usage: perl ../util/Blast_all_vs_all_repo_to_RBH.pl -r <repo_spec>
Optional: -t Type (PEP/CDS) [PEP]
-o Out directory [RBH_outdir]
-g Run commands on the grid (y/n) [n]
-p Platform (UGER, LSF, GridEngine) [UGER]
-q Queue name [short]
> perl util/Orthologs_to_summary.pl -h
$ perl util/Orthologs_to_summary.pl -h
Usage: perl util/Orthologs_to_summary.pl -o <ortholog file (E.g. PEP.RBH.OrthoClusters, all_orthomcl.out, Orthogroups.csv)>
Optional -t Type of clustering (OMCL, RBH, Orthofinder) [OMCL]
-d Outdir from Blast_all_vs_all_repo_to_OrthoMCL.pl (if used) [OMCL_outdir]
-r Repo Spec [./Repo_spec.txt]
-p Repo Spec CDS/PEP file [./Repo_spec.txt.all.PEP]
> perl util/DAGchainer_from_gene_clusters.pl
$ perl util/DAGchainer_from_gene_clusters.pl
Usage: perl util/DAGchainer_from_gene_clusters.pl -r <repo_spec> -c <Ortholog cluster data (E.g. ORTHOMCLBLASTFILE.clusters)>
Optional: -z File containing a list of genomes to restrict the analysis to []
-i Minimum number of paired genes in a single dagchain [4]
-o Cmds outdir [dagchainer_rundir]
-l Cmds outfile [cluster_cmds]
-g Run commands on the grid (y/n) [n]
-p Platform (UGER, LSF, GridEngine) [UGER]
-q Queue (hour, short, long) [short]
-v Verbose (y/n) [n]
テストラン
cd examples/
perl ../SynIma.pl -a Repo_spec.txt.dagchainer.aligncoords -b Repo_spec.txt.dagchainer.aligncoords.spans
- -a Aligncoords
- -b Aligncoords.spans
ディレクトリSynIma-outputができる。

config.txt.pdf

実行方法 (full example run)
1、Repo_spec.txtを指定してCreate_full_repo_sequence_databases.plを実行。
cd examples/
perl ../util/Create_full_repo_sequence_databases.pl -r ./Repo_spec.txt
- -r Repo_spec.txt []
Repo_spec.txtには以下のような情報が記載されている。
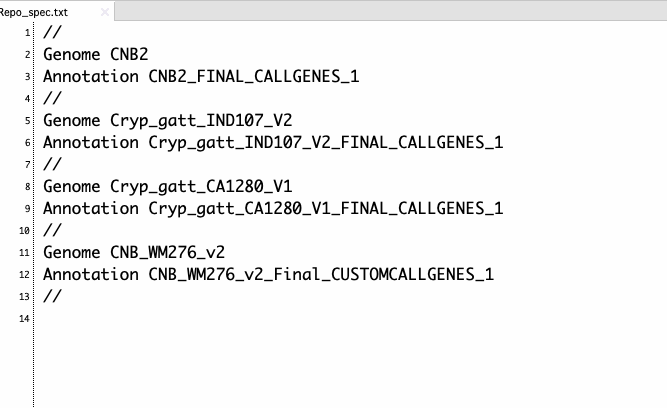
CNB2はgenome IDで、カレントに該当するgenome ID名のディレクトリがないといけない。上では4ゲノム比較するので、CNB2のほか、Cryp_gatt_IND107_V2、Cryp_gatt_CA1280_V1、CNB_WM276_v2のディレクトリも存在している必要があ
る。4つのディレクトリそれぞれには以下のようなファイルが入っている。
CNB2(他も同様)

写真にあるように、ディレクトリ内にはゲノム配列とテキストのアノテーション行に記載したannotation-id名のファイル、CNB2なら3行目で指定したCNB2_FINAL_CALLGENES_1~が含まれなければならない。すなわち、以下のようなファイル構成になる。

- ゲノム配列 - [genome-id].genome.faという名前にする必要がある。CNB2な CNB2.genome.fa。
アノテーションファイル(CNB2_FINAL_CALLGENES_1~)は以下の3つ。
- アノテーションのGFF3ファイル - [annotation-id].annotation.gff3
- コード領域の塩基配列cdsファイル - [annotation-id].annotation.cds
- タンパク質配列のPEPファイル - [annotation-id].annotation.pep (prptein.fasta)
GFF3にはcdsとpepと同じid名でgeneとmRNA のfeaturesが記載されていなければならない(詳細はGithubのREADME参照)。注意点として、別のゲノムで同じヘッダー名をしようしてはならない。よってヘッダーにchr1とかつけない方が良い。
2、run all vs all BLAST hits using Blast_grid_all_vs_all.pl
All versus allのblast解析を実行する。全タンパク質を使うため、時間がかかる(*1)。
perl ../util/Blast_grid_all_vs_all.pl -r ./Repo_spec.txt
ランが終わるとそれぞれのgenome_idディレクトリに~RBH_blast_[PEP/CDS]ができる。
3、 run OrthoMCL or reciprocal best hits (RBH) on the BLAST output
using Blast_all_vs_all_repo_to_OrthoMCL.pl or Blast_all_vs_all_repo_to_RBH.pl
respectively.
#ALTERNATIVELY 1
perl ../util/Blast_all_vs_all_repo_to_RBH.pl -r ./Repo_spec.txt
#ALTERNATIVELY 2
perl ../util/Blast_all_vs_all_repo_to_Orthofinder.pl -r ./Repo_spec.txt
4、summarise the OrthoMCL output (OMCL_outdir/all_orthomcl.out)
perl ../util/Orthologs_to_summary.pl -o all_orthomcl.out
- -o Ortholog file (E.g. PEP.RBH.OrthoClusters, all_orthomcl.out, Orthogroups.csv) []
(*2)
5、 run DAGChainer on the ortholog summary using DAGchainer
perl ../util/DAGchainer_from_gene_clusters.pl -r ./Repo_spec.txt \
-c GENE_CLUSTERS_SUMMARIES.OMCL/GENE_CLUSTERS_SUMMARIES.clusters
6、SynIma.pl
perl ../SynIma.pl -a Repo_spec.txt.dagchainer.aligncoords \
-b Repo_spec.txt.dagchainer.aligncoords.spans
引用
Synima: a Synteny imaging tool for annotated genome assemblies
Rhys A. Farrer
BMC Bioinformatics volume 18, Article number: 507 (2017)
*1
この4セットのexamleデータでも数時間かかる。
*2
コードを見る限り-oがオプションとして記載されていない。バグ修正する必要がある。
関連