ハイスループットシークエンシングおよび分析ツールの最近の爆発により、培養不可能な生物を含む生命のツリーを横切るほぼ全ての種のシークエンシングがより容易かつ安価になった。しかしながら、これらのゲノムの質と完全性は、リピート領域をアセンブリするチャレンジと可変または不十分なシーケンシングカバレッジ[ref.1]のために変化する可能性がある。microbial dark matter project [ref.2 link]、 Human Microbiome Project [ref.3]または1000 fungal genomes project(http://1000.fungalgenomes.org)などの大規模シーケンシングプロジェクトは、何千もの微生物ゲノムアセンブリを生み出してきた。ドラフトデータの迅速な作成と公開により、病理学、進化、および酵素またはpathwayの発見の研究に広く使用される重要かつ有用なデータセットとして貢献できる。ドラフトゲノムの質と完成度が様々であると、遺伝子量、 transposable element の量、ゲノムサイズに関して推論に影響を与える可能性がある。それから推論することができる情報の質のコンテキストを提供するためにゲノムの完全性を定量化する必要がある。この研究はまた、系統特異的な遺伝子の喪失が、特に真菌において進化における重要な推進力であるという観察によって動機付けられており[ref.4, 5]、欠けている遺伝子のパターンについて描かれた結論の正確さは類似の品質ゲノム間の比較を必要とする。
ゲノムの品質と完全性を評価するためのアプローチは、ほぼ100の異なる測定基準を使用して提案されています[ref.6]。残念なことに、これらの測定基準のほとんどは一般に非モデル種には適用できない。なぜならそれらは高価であるか、または多数のものを取得するには実行不可能であるかなりの量の追加の高品質データ(例えばフォスミド、リファレンスゲノム、optical maps)を必要とする。現在のところ、アセンブリの欠落データ量を事前知識なしに見積もることを試みる方法はほとんどない。最も一般的なアプローチの1つであるCEGMAは、248のシングルコピーマーカー遺伝子セットの存在により完全性を推定する[ref.7、8]。 CEGMAは数多くの研究で使用されてきたが、重要な問題はマーカーが6つのモデル真核生物種から選択されただけであり、より遠い系統がサンプリングされるためこれらのマーカーの遍在性と検出は矛盾することである。 CEGMAは最近サポートが中止されており、著者は代替ツールの使用を推奨している(http://www.acgt.me/blog/2015/5/18/goodbye-cegma-hello-busco)。この概念は最近BUSCOでクレードに焦点を絞ったタンパク質コーディング遺伝子マーカーのセットで再検討され更新された[ref.9]。 246のシングルコピー真菌遺伝子ファミリーの別のセットがFUNYBASEによって提案された[ref.10]。後者は一連の保存された真菌遺伝子を提供するが、そのツールはゲノムの完全性を評価するために明確に開発されていない。さらに、FUNYBASEデータベースは2010年に作成されたが、多様な真菌ゲノムのより幅広いサンプリングが利用可能になった[ref.11 link]。
完全性を評価するための独立したマーカーのデータセットを構築するためには、典型的には、シングルコピーオルソログ遺伝子が選択される。マルチコピー遺伝子ファミリーはこれらの選択において系統的に除外されているが、それらの有用性、ならびに代替の非タンパク質コード遺伝子マーカーは、ゲノムの完全性を評価する際に十分に検討されていない。ゲノムアセンブリの2つの要約統計量は、品質と完全性を評価するために頻繁に使用される。アセンブリの断片化レベルを記述するN50およびL50統計[ref.12]は、アセンブリのscaffoldsまたはcontigsの長さに基づいて計算される(一部略)。
本研究では、真菌界に焦点を当てた。真菌のゲノムサイズは、数メガベース(Mb)から1000 Mb近くまでさまざまである[ref.11]。この論文の主な動機は真菌ゲノムのためのアセンブリの完成度の現実的な推定を提供することである。精度は、不完全なアセンブリによって人工的に断片化されているように見えたり、系統によっては急速に進化する遺伝子座によって失われたように見えたりする可能性がある遺伝子について、正確に識別する能力に依存する。遺伝子の内容からゲノムの完全性を計算する際には、遺伝子の性質、進化の軌跡、および損失の可能性を考慮する必要がある。本著者らは、マーカーの新規セットを提案し、FGMP(Fungal Genome Mapping Project)と呼ばれるゲノムアセンブリにおけるそれらの存在を評価するためのパイプラインを構築した。FGMPの多段階アプローチは、同定可能な真菌タンパク質と高度に保存された非コード領域を統合することによって以前のアプローチを拡張する。選択されたタンパク質マーカーは、シングルコピーマーカーとマルチコピーマーカーの両方を含み、以前に公表されたデータセットとのオーバーラップは50%のみで、完全性を評価するための異なる次元の配列進化を提供する。真菌ゲノムの高度に保存された非コード領域は、本著者らが開発しFGMPのゲノム完全性評価に組み込んだ新規のリソース源である。最後に、アセンブリの必要性を回避する、シーケンシングリードにおいてマーカーを検索するために、rarefaction analysisに結合されたmultisampling approachを使用する。したがって、研究者は、計算コストが高くなる可能性があるアセンブリを試みる前に、FGMPを使用することで手元にある一連のリードの品質を迅速に評価できる。最後に、様々な範囲のクオリティのゲノムアセンブリからなる246の真菌種についての最新の方法と本ツールを並べて比較した。 NCBIアセンブリアーカイブに1つ以上のアセンブリがリリースされ記録されている57の真菌種において、アセンブリの改善/低下を捉えた。本ツールのモジュール構造は、より複雑なパイプラインに簡単に組み込むことができ、ゲノムの完全性推定のための貴重なツールになる。
FGMPの典型的な実行は3つのステップから成る。 第一に、生の遺伝子モデル(タンパク質)セットがクエリのアセンブリから生成され、それはその後のステップでさらにフィルタリングされ高信頼性遺伝子になる。 第二に、高度に保存された非コード真菌DNA elements(> 200ヌクレオチド)の存在が推定される。 第三に、possibleミスアセンブリまたは崩壊した重複領域を追跡するため、遍在性マルチコピータンパク質ファミリーのコピー数を決定する。 FGMPのワークフローを論文図1に示し、その方法論を次のセクションでさらに詳しく説明する。 FGMPはメタゲノムアセンブリの完全性を評価することを意図していない。 入力データは単一の種に属すると期待され、バクテリアコンタミネーションは事前に除去する必要がある。
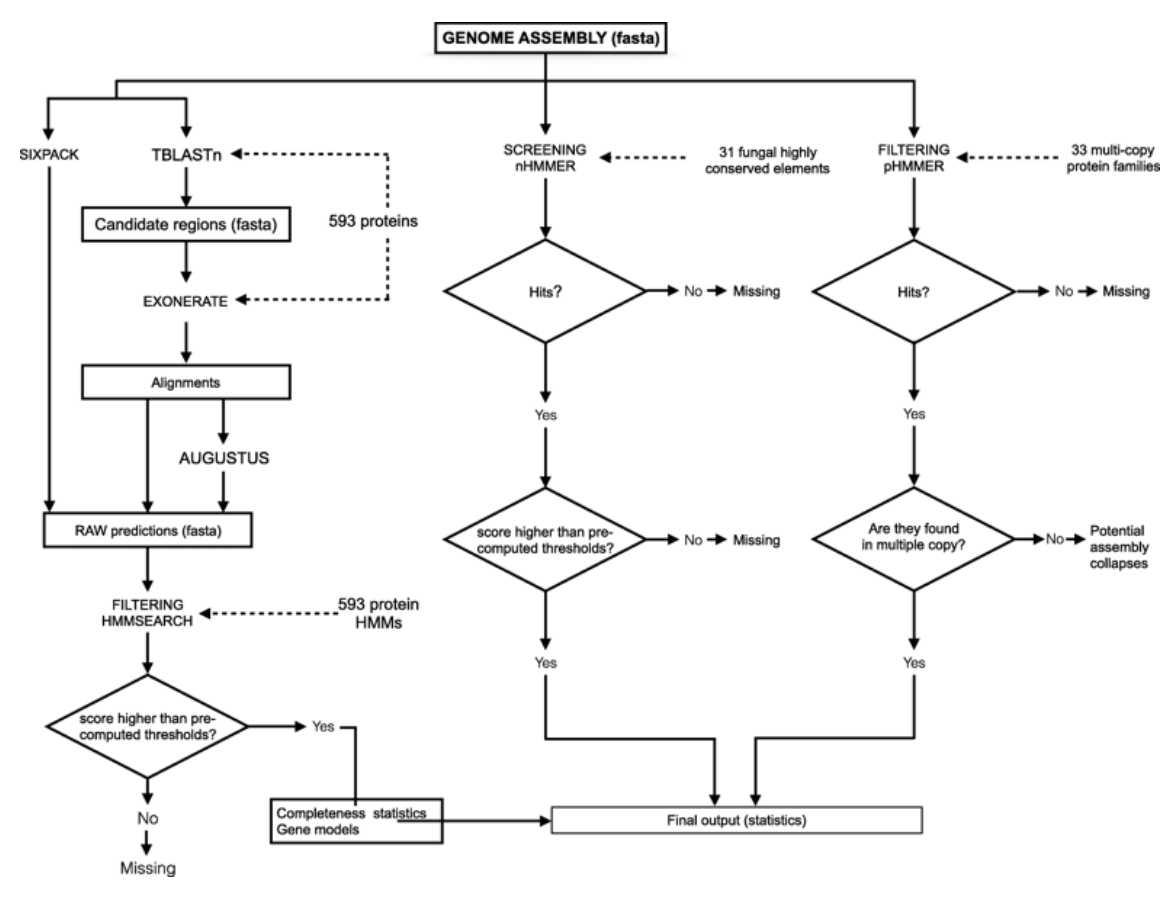
The FGMP workflow. 論文より転載。
インストール
依存
System requirements
- Perl 5 (tested with the version 20)
- BioPerl-1.6.924 http://bioperl.org
- HMMER v3.0 http://hmmer.org/
- NCBI BLASTALL (tested using version 2.2.31+) ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/2.2.31/
- Exonerate (tested using version 2.2.0) https://www.ebi.ac.uk/about/vertebrate-genomics/software/exonerate
- Augustus (tested using version 3.0.3) http://augustus.gobics.de/
本体 Github
#Anaconda環境ならcondaで
conda install -c bioconda fgmp
condaではperlのライブラリの一部が@INC配列に認識されなかった。cpanmで手早く導入(-lでパス指定)。
cpanm IO/All.pm
本体のfgmpも認識されない。git cloneしてsrc/fgmp.plを直接叩く。
git clone https://github.com/stajichlab/FGMP.git
cd FGMP/src/
> perl fgmp.pl
# perl fgmp.pl
fgmp
SOFTWARE:
fgmp - 1.0.2
USAGE
fgmp [options] -g < genome_fasta_file >
DESCRIPTION
REQUIRES
fgmp requieres the installations of the following softwares
- hmmer (HMMER 3.0)
- NCBI blast+
- Exonerate
- BioPerl xxx
- IO::All
- Emboss sixpack & csplit
ENVIRONMENT VARIABLES
You can specific the path where the fgmp can find the default files
with the shell variable "fgmp".
o Using a Bourne-SHell
export FGMP="path"
export FGMPTMP="path"
export PERL5LIB="$PERL5LIB:$FGMP/lib"
COMMAND-LINE OPTIONS
Available options and a short description are listed here;
-g, --genome genome in fasta format
-p, --protein protein seeds
-o, --output output file prefix
-d, --blastdb blast database for the genome sequence
-c, --cutoff_file profiles cutoff file
-m, --mark_file completeness markers
-r, --reads reads
--fuces_hmm fungal Ultra Conserved Elements (hmms)
--fuces_prefix fungal Ultra Conserved Elements (names - one per line please!)
--multicopies default: multicopy genes from 1FKG data
-t, --tag tag to use OMA for fgmp, FUNY (Funybase) or CEG (cegma)
-T, --threads Specify the number of processor threads to use
-v, --verbose show progress
-q, quiet suppress show log
-h, --help show this help
--tmp keep temporary files
-augTraingCutoff specify the num of genes for augustus training
--nsampleSize Specify the # of samples for "SEARCH IN READS" module
--nsampleSize Specify the sample size for the "SEARCH IN READS" module
BUGS:
Please report bugs to 'ousmanecis@gmail.com'.
AUTHORS:
fgmp has been developped by Ousmane H. Cisse and Jason E. Stajich.
テストラン
ドラフトゲノムのfastaを指定する。
perl fgmp.pl -g ../sample/sample_test.dna > fgmp_report.out
パスが違ったり割と色々なエラーが起きる。修正できたら追記します。
引用
FGMP: assessing fungal genome completeness
Ousmane H. Cissé, Jason E. Stajich
BMC Bioinformatics 2019 20:184
関連