バクテリア、菌類、植物は、それぞれが関与する多様な生物間相互作用において、地球全体で独自の特定の生態学的役割を持つ特殊な代謝産物の非常に多様性を生み出している。この多様な特殊な代謝は、医薬品、農業、製造業で広く使用されている天然物の豊富な供給源である。最も特殊な代謝パスウェイの遺伝子がいわゆる生合成遺伝子クラスター(BGC)に物理的にクラスター化されている細菌および真菌では、ゲノム配列の急速な蓄積が天然産物発見のプロセスに革命をもたらした:実際、ゲノムマイニングが新規分子の発見方法として支配的になった(ref.1–4)。このゲノムマイニングプロセスでは、ゲノムシーケンスでBGCがコンピューターで識別され、機能解析(たとえば、メタボロームデータ、化学構造予測、変異体ライブラリー、および/またはheterologous expressionを使用)によって分子にリンクされる。このゲノムマイニング手順の多くのシーケンスベースの側面は、2010年に開始されたantiSMASHフレームワークによって促進され(ref.5)、それ以来継続的な開発が行われている(ref.6,7)。ゲノムマイニング手順には2つの主な目的がある:(i)重要な既知化合物の生合成遺伝子を見つけて、工業株での発酵によるheterologous productionを可能にすること、および(ii)生合成遺伝子クラスターの多様性によって導かれる新しい天然物のケミストリを識別すること。全体として、この開発は「遺伝子クラスター革命」と呼ばれている(ref.1)。
近年、微生物だけでなく植物の生合成パスウェイも頻繁に染色体上にクラスター化されていることが明らかになっている:最初の環状ヒドロキサム酸2,4-dihydroxy-1,4-benzoxazin-3-one(DIBOA)とアベナシン遺伝子クラスター(ref.8,9)の発見後、約30の植物BGCが発見された(ref.10,11)。(一部略)
真核生物のゲノムシーケンス(ref.13)におけるさまざまな技術開発により、最終的に大規模で完全な植物ゲノムシーケンスが実現可能になった:ほぼ100種の高品質の植物ゲノムシーケンスがすでに公開されており、ほぼ完全なゲノムをわずかな時間と各10〜5万米ドルでシーケンスできる。したがって、ゲノムマイニングは植物の天然産物の研究においても重要な方法論になる可能性があり、したがって、植物の天然産物の研究コミュニティが独自の「遺伝子クラスター革命」を持つ現実的な機会が存在している。当然、これを実現するために必要な重要な技術は、植物BGCの識別と分析のために特別に設計された計算フレームワークである。重要なのは、細菌および真菌のゲノムマイニングに使用できるツールは、植物には不十分な事である(ref.14)。(i)植物の生合成パスウェイには、細菌および真菌には見られない独自の酵素ファミリーが含まれる。 (ii)すべての植物生合成パスウェイがクラスター化されているわけではないため(アントシアニン(ref.15)など)、生合成遺伝子の同定はBGC同定と同等ではない; (iii)植物ゲノムの遺伝子間距離はより大きく、はるかに変動しやすい(ref.16–19)。 (iv)植物ゲノムにはその産物がパスウェイを構成しない遺伝子クラスター(タンデム配列など)が含まれる。 (v)いくつかの植物のパスウェイは複数のBGCに splitされている(ref.20,21)。
ここでは、これらの各課題に対処するように設計された、植物用のantiSMASH(または略して「plantiSMASH」)を紹介する。植物生合成パスウェイに関与することが知られている酵素ファミリーのプロファイルHidden Markov Models(pHMM)の包括的なライブラリーと、同じファミリーに属する予測タンパク質配列のCD-HITクラスタリングと組み合わせることで、複数の遺伝子座をコードするゲノム遺伝子座の効率的な識別が可能になる。さらに、これらの候補BGC内の遺伝子発現パターンの分析と同様に、比較ゲノム解析により、1つのパスウェイで一緒に機能する遺伝子をエンコードする可能性について各遺伝子座を評価できる。最後に、候補BGCとゲノム全体の他の遺伝子との共発現解析により、複数の遺伝子座でエンコードされた生合成パスウェイを特定できる。この新しいフレームワークを活用するために、植物界全体のBGC多様性の初期分析を提供する。これは、多様な種の多くの複雑な生合成遺伝子座の存在を示している。
Help
http://plantismash.secondarymetabolites.org/help.html#news
. @satriaphd and #hernandosuarez wrote a nice book chapter / protocol on how to use plantiSMASH for identifying plant gene clusters: https://t.co/hkG2fudmeI
— Marnix Medema (@marnixmedema) June 5, 2018
Our #plantiSMASH poster at #BioSB17https://t.co/d4POsLHFIK pic.twitter.com/RI0xGfwag4
— Satria A Kautsar (@satriaphd) April 5, 2017
http://plantismash.secondarymetabolites.org/download.html
public webサービスの使い方
http://plantismash.secondarymetabolites.org
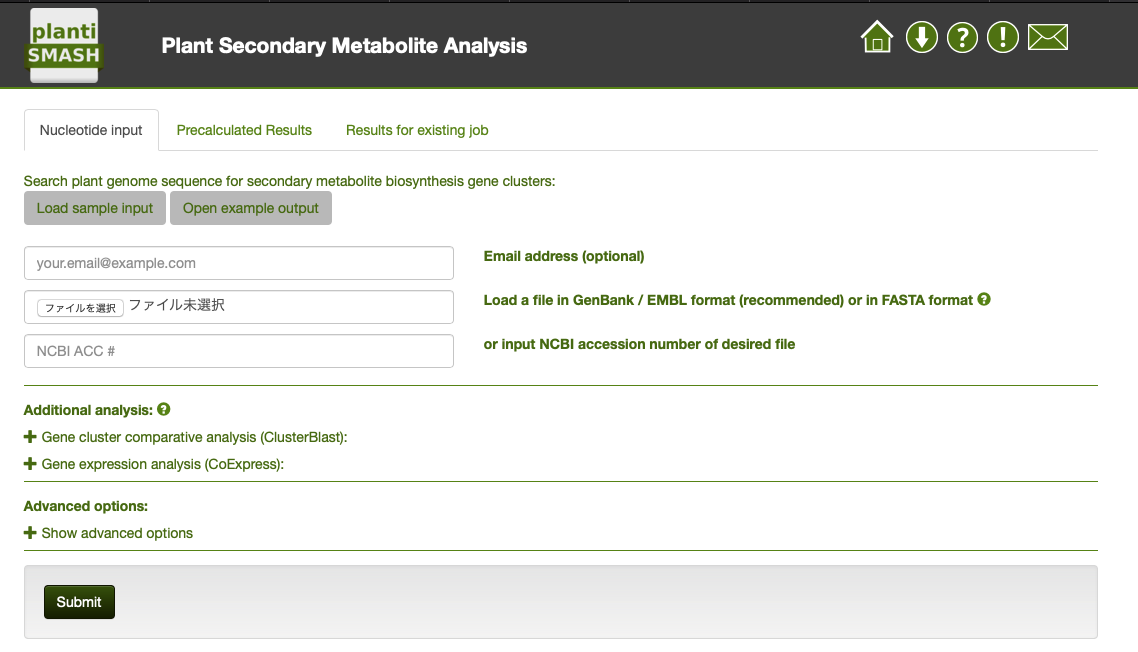
ゲノムのFASTAかGenBank EMBL フォーマットのゲノムファイルを指定する。publicデータベースのaccession IDを使うなら一番下のウィンドウに記載する。

ここではLoad sampleをクリックしてシロイヌナズナのchr5を使う。
Adbancedからパラメータやオプションの解析を実行するかどうか選択できる。またGene expression analysis (CoExpress):からはGEOのco-expressionデータを指定したり、手持ちのCSVファイルもアップ可能。


submitボタンを押して実行する。
結果。検出された推定遺伝子クラスターの一覧が表示される。テストデータでは合計10クラスター検出されている。

様々なクラスターが検出されていることが分かる。1つ開いてみる。

Cluster2。生合成パスウェイと予測された遺伝子は割り当てられた酵素タンパク質ドメインプロファイルによって色分けされる。下に凡例がある。

クリックすると詳細が表示される。

アミノ酸配列のコピー、染色体上の位置の確認、blastp解析など実行できる。
showボタンを押すと遺伝子リストが表示される。

やや分かりにくいが、戻るときは①②...の左側のOverviewの文字をクリックする。

Co expressionデータを読み込んでおくと追加で階層的クラスタリングなど実行できる。Co expressionデータの中に欠損値(一部だけmissingがある行)がある場合は埋める必要があったり、フォーマットは厳密でなくてはならない。詳細はFAQ参照。
http://plantismash.secondarymetabolites.org/help.html#faq
引用
plantiSMASH: automated identification, annotation and expression analysis of plant biosynthetic gene clusters
Satria A. Kautsar, Hernando G. Suarez Duran, Kai Blin, Anne Osbourn, Marnix H. Medema
Nucleic Acids Res. 2017 Jul 3; 45(Web Server issue): W55–W63
関連