ハイスループットなオミクス手法により、メタボロミクスデータを含む大規模データセットが増加しており、解析が難しいことが少なくなくなってきている。複数のサンプリング条件(例:異なる時間や処理)におけるメタボロミクスデータをパスウェイの文脈でマッピング・調査し、研究者がそれらのデータセットを扱い解析できるよう支援するために、PathwayNexus を開発した。PathwayNexus はマッピング結果を行列形式で提示し、化合物とパスウェイの関係を容易に把握できるようにする。さらに、ランキング、ソート、クラスタリング、パスウェイビュー、その他の解析ツールといった機能も提供する。その主たる目的は、大量のパスウェイ群をユーザーの特定の関心に合致する、より小さく関連性の高い部分集合へと要約することである。ここで提示する手法は PathwayNexus に実装されており、Vanted 用のオープンソースのアドオンとして www.cls.uni-konstanz.de/software/pathway-nexus から入手できる。
Overview
Software | Life Science Informatics - Prof. Dr. Falk Schreiber
インストール
依存
https://cls.uni-konstanz.de/software/pathway-nexus/overview/にアクセスする。
中央のDownload PathwayNexus ボタンからプログラムの.jarファイルをダウンロードする。
PathwayNexusを使うにはVANTEDが必要。VANTEDのadd-onとして使う。
https://cls.uni-konstanz.de/software/vanted/download/
ここではMac版を選択した。
vanted-2.8.8.dmgをダブルクリックして展開する。許可

VANTEDが立ち上がったら別にダウンロードしたPathwayNexusの.jarファイルをドラッグ&ドロップする。

このようにVANTED のウィンドウにドラッグ&ドロップするか、あるいは、Edit > Preferences > Addon Manager > Install / Configure Add-ons > Install Add-on から手動でインストールする。
実行方法
PathwayNexus をインストールしたら、画面右のパネルのボタンからPathwayNexusに切り替える。

それからPathwayNexusボタン下の「Load Experiment Data」ボタンをクリックして読み込むファイルを選択する。ここではサンプルデータを使う(解凍したCSVを指定)。読み込みには数秒くらいかかるので他のボタンに触らず待つ。
デモデータが読み込まれた。

読み込んだデモデータは以下のようなCSV(メタボロミクス解析の定量データ)。

各行が1種類の代謝物(metabolite)を表す。ファイルフォーマットは以下で説明されている。
https://cls.uni-konstanz.de/software/pathway-nexus/learn/file-format/
データの読み込みが完了したらマトリクスの操作を行うことができる(解説:
https://cls.uni-konstanz.de/software/pathway-nexus/learn/matrix-panel-and-tools/)
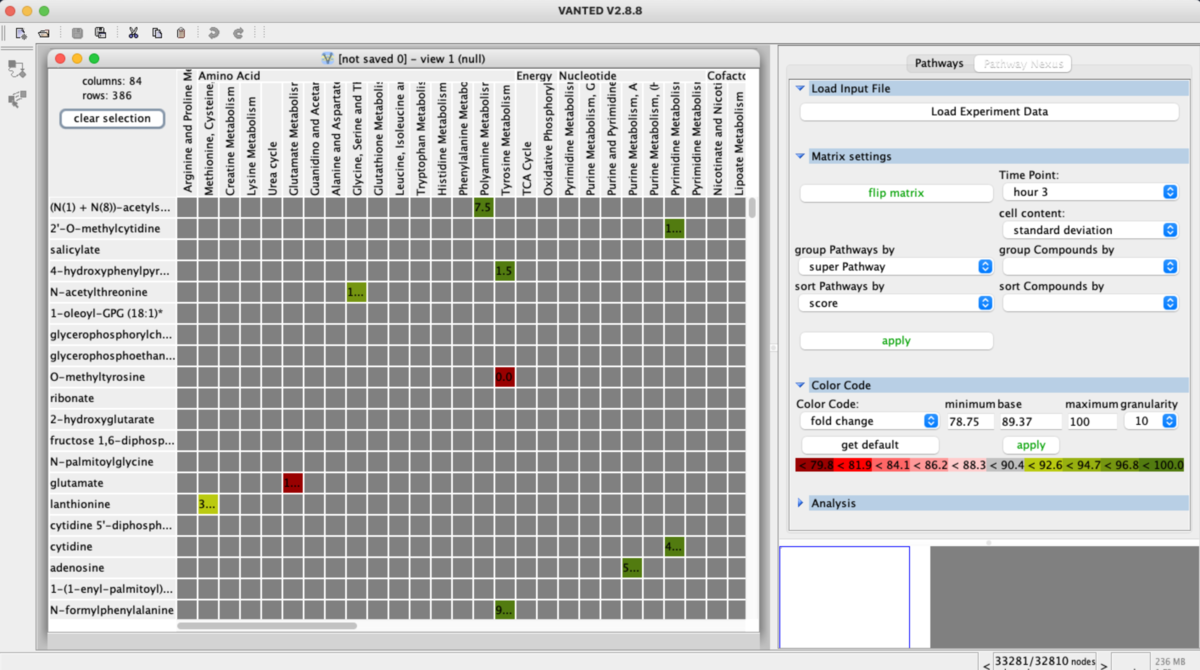
代謝物(行)とそれらが属する経路(列)がマトリクス状に配置される。各セルには実験から得られた数値(どれを表示するかは選択可能)が入り、その値に応じて色分けされる。灰色は空を表す。
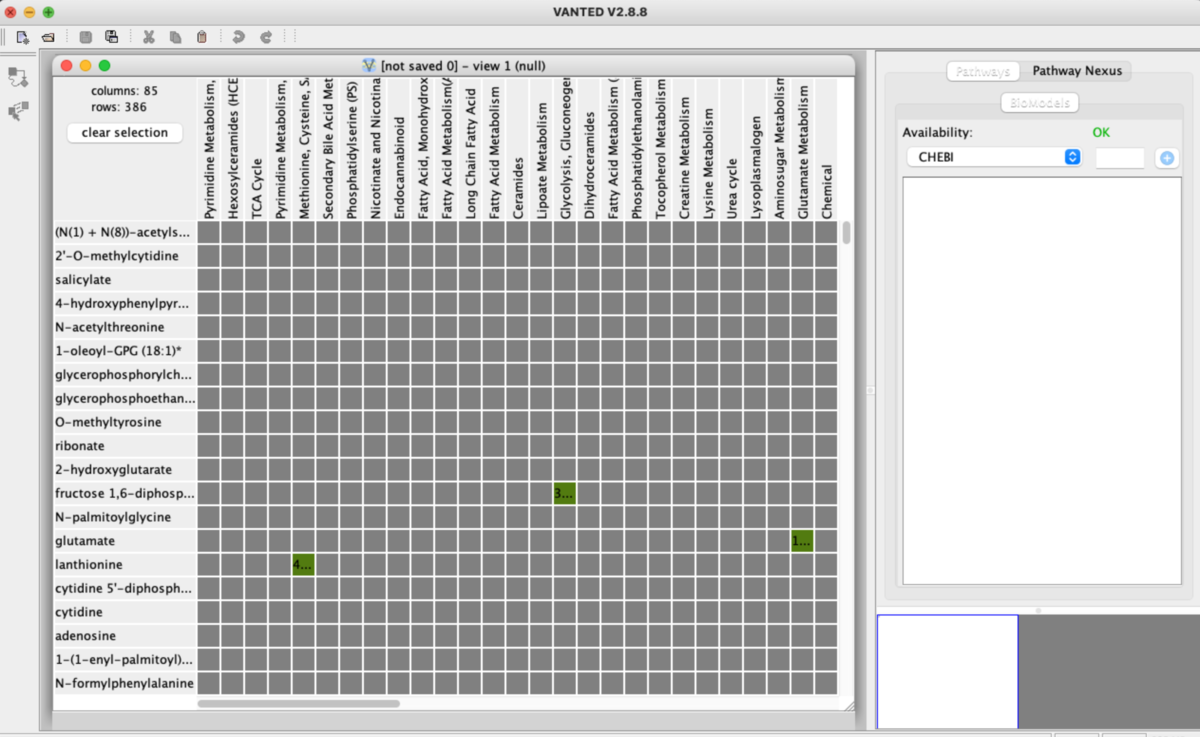
(デフォルトでは、代謝物は行、経路は列、並べ替え可能)
マウスホイールで拡大縮小できる。小さくした。

セルをクリックすると、その行と列が十字状にハイライトされ、該当する代謝物と経路が選択される。
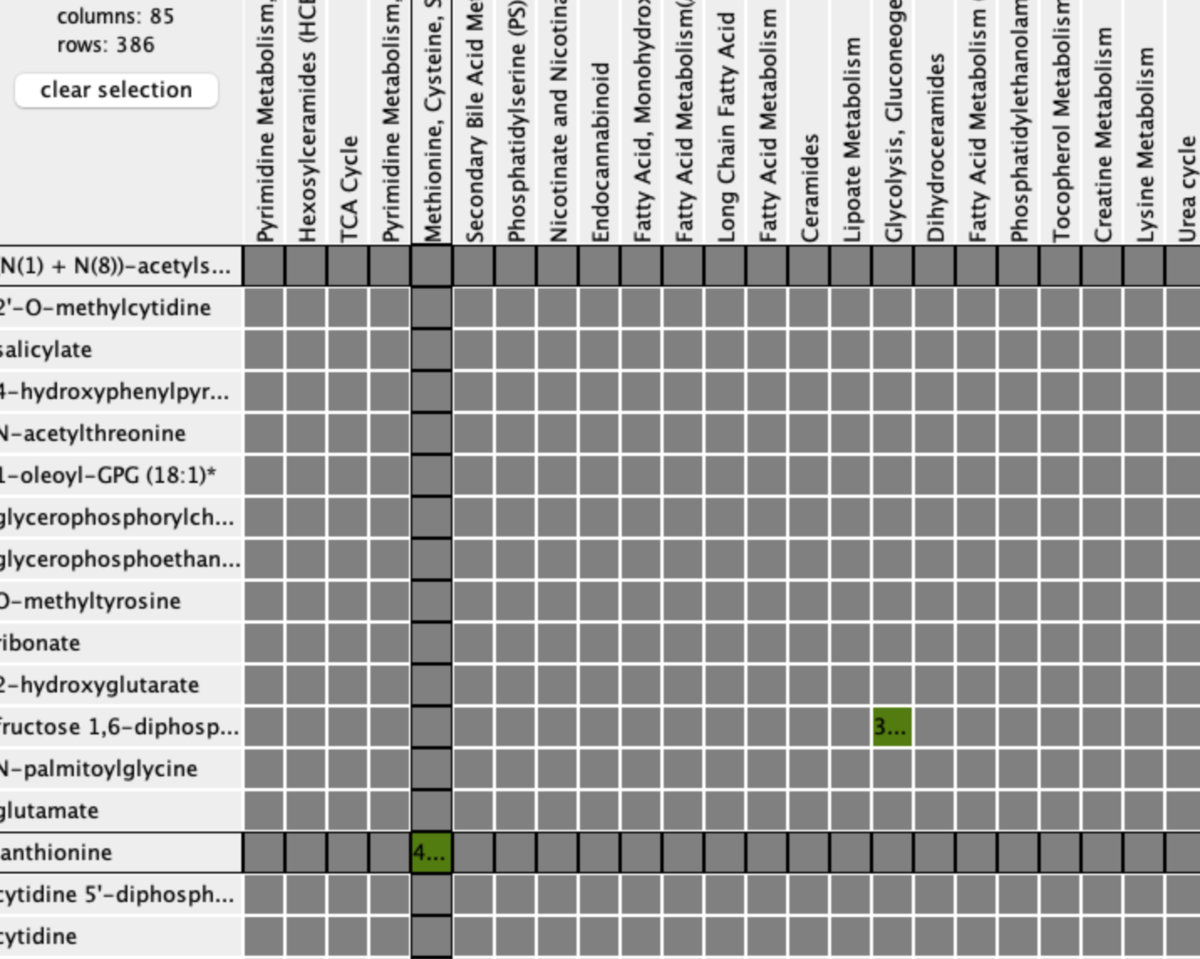
クリックするたびに累積で選択されていく。もう一度クリックすると選択が解除される。

すべてまとめて選択を解除するには、左上のclear selectionをクリックする。
右側のツールタブからマトリクスの操作ができる。Matrix Settings => super Pathwayをクリックし、applyボタンを押した。数秒待つとパスウェイの大分類ごとに個々のパスウェイが分類され、パスウェイ名の上に大分類の注釈が付いた。
右側のツールタブからSort Pathway byを選択すると行と列それぞれを並べ替えが可能。
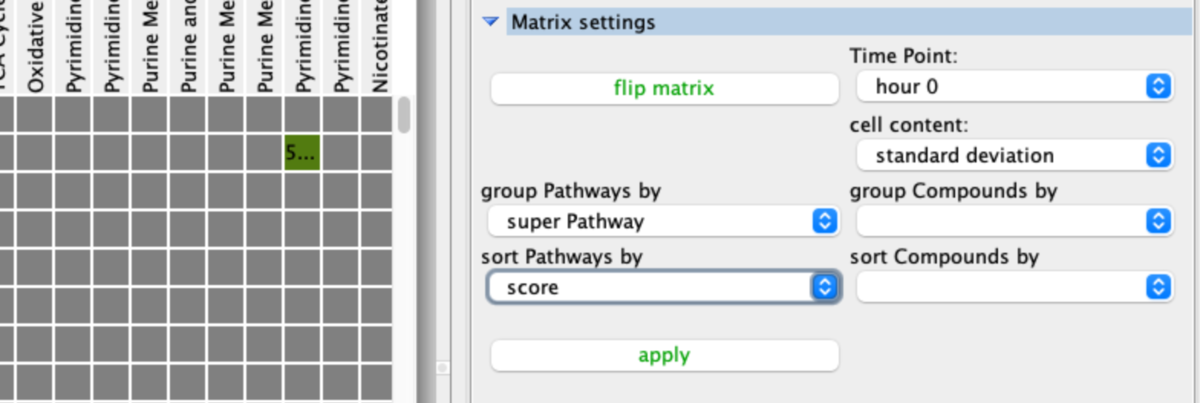
右側のツールタブからTime pointを3に(* デモデータでは複数の時間を選べる)、セルの色をstandard deviationからfold changeにした。

Time point 3(上の画像ではtime point 10)
代謝物またはパスウェイを右クリックするとコンテキストメニューが開く。

コンテキストメニューのassociate compounds機能は、その名の通り、例えばpathway(列)を右クリックしてassociate compoundsを選択すると、そのpathwayで検出されている化合物が選択状態になる。化合物側(行)でも同じことができる(=> associate pathways)。

関心のある物質やパスウェイを特定している場合は、パスウェイを右クリックしてネットワークビューにアクセスできる。
興味があるpathwayや化合物が見つかったら、化合物かpathwayを右クリックしてnetwork viewを選択する。しばらく待つとpathway adapterのウィンドウが開く。

先ほど選択していたpathway中で検出されている化合物が読み込まれている。
get data from DBをクリックして、KEGGからネットワーク越しにpathwayの情報を取得した。

右下のnetwork viewをクリックすると、pathway図がダウンロードされて表示される。

進捗が右下に表示される。1分程度かかる。
アプリのウィンドウとして表示されるので、隠れないように元のパネルは最小化しておく。しばらく待つと表示された。

検出されている代謝物(化合物)はリファレンスカラーから、色が量に応じて変化している。

化合物のノードはドラッグして移動できる。図自体はマウスホイールで拡大縮小できる。

ドラッグして囲むことで複数選択して、まとめて移動することもできる。

ノードもエッジもダブルクリックして編集できるようになっている。

例えばアノテーションを編集したり、外見を変えたりできる。
コメント
簡単にだけ紹介しました。興味ある方はダウンロードして使ってみてください。
論文より
- VANTED 内で新しいパスウェイを作成できる(しばしば不自然に見える古典的な経路から逸脱することが可能)。また KEGG、BioModelsDB などのデータベースからパスウェイをダウンロードすることもできる。データはスプレッドシートとして読み込まれ、代謝物(または経路上にマッピング可能な他の実体)に関する情報、任意のデータベース識別子、および代謝物に割り当てられた実験データ(例:濃度、フォールドチェンジ、P 値など。経時的または異なる処理条件下)を提供する。ユーザーはマッピングのための情報を追加でき、追加された情報は物質と経路をそれぞれ一方の軸に配置したマトリクスに表示される。セルには、色分けされた値と物質に関する情報が示される(図1参照)。
引用
PathwayNexus: a tool for interactive metabolic data analysis Open Access
Philipp Eberhard, Martin Kern, Michael Aichem, Hanna Borlinghaus, Karsten Klein, Johannes Delp, Ilinca Suciu, Benjamin Moser, Daniel Dietrich, Marcel Leist, Falk Schreiber
Bioinformatics, Volume 40, Issue 6, June 2024.