2020 11/7 タイトル修正
2020 11/8 感想追加
2020 11/11 誤字修正, タイトル修正(”主に植物”を削除)
構造的特徴に基づくコンピュータプログラムを用いたLTR要素の同定は効率的であるが(10,11)、多数の偽陽性(4)に悩まされている。最近、インタクトなLTRレトロトランスポゾンの正確なde novo同定のために、LTR_retrieverソフトウェアが開発された(4)。このツールは、入力品質に関係なく LTR の偽陽性を排除し、非常に低い偽発見率で超高感度・高精度を実証している(4)。植物ゲノムからインタクトな LTR 要素を検索している間に、ドラフトゲノムと比較して、より多くの完成したゲノムアセンブリからより多くのインタクトな要素が同定されることが観察された。例えば、イネの標準ゲノム「日本晴」(MSUv7 版)からは 2,052 個のインタクトな LTR-RT が得られたが、次世代シークエンシング(NGS)技術を用いた同一ゲノムからは 239 個のインタクトな LTR-RT のみが同定された((12)から得られたもの)。Jiaoらは、PacBioのロングリード技術を用いて配列決定された新しいトウモロコシ(Zea mays)のリファレンスゲノム(v4)でも同様の結果を報告している(13)。また、Al-Dousらは、ショートリード法によるゲノムシークエンシングでは、ナツメヤシ(Phoenix dactylifera)ゲノムのLTR-RTのような長いリピートのごく一部しか解決できないことを示している(14)。これらの知見は、より連続的なゲノムアセンブリを行えば、より多くの無傷のLTR要素が同定されることを示唆している。このように、識別可能なインタクトなLTR要素の量は、ひいては、遺伝子間および反復配列空間のアセンブリ品質を示すことができる。
LTRレトロトランスポゾン(LTR-RT)は、ドラフトゲノムでは貧弱にしかアセンブリされていないリピートであることが知られている。ここでは、LTR-RTを用いてアセンブリの連続性を評価するLTR Assembly Index (LAI)と呼ばれるリファレンス不要のゲノム指標を提案する。LTR-RTの増幅ダイナミクスを補正した後、LAIはゲノムサイズ、ゲノムLTR-RT含有量、遺伝子空間評価指標(BUSCOやCEGMA)に依存しないことを示す。
LAIは、ほとんどの植物ゲノムにおいて最大のゲノム構成要素を占めるLTRレトロトランスポゾンに基づいて標準化された指標である(raw LAI定義はResult sectionに記述されている)。 定義中の"Intact LTR-RT length"sは、完全なLTR、LTR領域に隣接するジヌクレオチド末端(通常5′-TG...CA-3′)、要素に隣接する4-6 bpの標的部位の重複、および内部領域のタンパク質配列のアラインメントなど、多くの配列特徴を認識するLTR_retriever(4)によって同定される。分母の"Total LTR sequence length"の推定には、LTR_retrieverで生成した非冗長LTR-RTライブラリ(exemplars)を用いて、相同性ベースのRepeatMaskerプログラムでゲノムを検索し、ゲノム内の全アノテーション配列の長さを合計したものとした。LTR レトロトランスポゾンの分解により認識できない配列断片が残っている場合には、この推定値の把握が困難な場合がある。ゲノム内のすべてのLTR配列を同定するために、RepeatMaskerを用いた相同性検索において、発散閾値を段階的に上げていった。イネとシロイヌナズナの両方のLTR-RTアノテーションにおいて、配列発散率が40%まで上昇すると、生のLAIスコアは安定化した(補足図S2)。したがって、本研究では、40%の発散率をLTR-RTの総含有量の推定に用いている。
raw LAIスコアは、LTR-RTの増幅および除去などのLTR-RTの活性(結果参照)と相関しているので、これらの影響を補正するために、一倍体(1×)ゲノムの LTR配列の平均同一性を使用した。LTRの平均同一性を推定するために、LTR領域としてアノテーションされたゲノム配列を抽出し、all vs all BLASTを行った。最も高いクエリカバレッジ(自己アラインメントを除く)を持つ各配列ヒットの同一性を、全ゲノムのLTR同一性を推定するために使用した。20の高品質ゲノムを用いて推定した補正係数2.8138を用いて、raw LAIスコアをadjusted LAI = raw LAI + 2.8138 × (94 - 全ゲノムLTR同一性)の式で補正した。raw LAI = 0、または調整により負の値が得られた場合は adjusted LAI を 0 とした。LTR同一性の推定およびraw LAIの補正は、LAIプログラムによっても行われた。LTR_retrieverによって推定されたintact LTR-RTの平均年齢もLTR-RT活性の指標として使用されたが、若いLTR-RTはアセンブリの悪いものの一つであるため、ドラフトゲノムでは年齢が過大評価される可能性がある。LAIは総LTR-RT含有量とは無関係であるが、総LTR-RT含有量が5%未満、intact LTR-RT含有量が0.1%未満の場合、LAIの推定は経験的に正確ではない。異常に高い LAI スコアを制御するために、全ゲノムおよびローカルな LAI 推定において、LTR-RT 含有量の合計が 1%未満の場合、ローカルな LAI は元のスコアの 10%にダウンスケールされる。LAIはバージョン1.5以降のLTR_retrieverのデフォルト出力であり、GNU General Public License v3.0 (https://github.com/oushujun/LTR_retriever)の下、GitHubを通じて自由に利用できる。
インストール
ubuntu18.04でテストした。
#以下3つはltr_retrieverで使用するファイルを作成するために必要
#genometools
sudo apt-get install -y genometools
conda install -c bioconda ltr_finder -y
git clone https://github.com/oushujun/LTR_FINDER_parallel.git
#bioconda (link)
conda create -n ltr_retriever python=3.7 -y
conda activate ltr_retriever
conda install -c bioconda ltr_retriever -y
> LAI
$ LAI
Input file(s) not exist!
The LTR Assembly Index (LAI) is developed to evaluate the assembly continunity of repetitive sequences
Usage: ./LAI -genome genome.fa -intact intact.pass.list -all genome.out [options]
Options:
-genome [file] The genome file that is used to generate everything.
-intact [file] A list of intact LTR-RTs generated by LTR_retriever (genome.fa.pass.list).
-all [file] RepeatMasker annotation of all LTR sequences in the genome (genome.fa.out).
-window [int] Window size for LAI estimation. Default: 3000000 (3 Mb)
-step [int] Step size for the estimation window to move forward. Default: 300000 (300 Kb)
Set step size = window size if prefer non-overlapping outputs.
-q Quick estimation of LTR identity (much faster for large genomes, may sacrifice ~0.5% of accuracy).
-qq No estimation of LTR identity, only output raw LAI for within species comparison (very quick).
-mono [file] This parameter is mainly for ployploid genomes. User provides a list of sequence names that represent a monoploid (1x).
LAI will calculated only on these sequences if provided. So user can also specify sequence of interest for LAI calculation.
-iden [0-100] Mean LTR identity (%) in the monoploid (1x) genome. This parameter will inactivate de novo estimation (same speed to -qq).
-totLTR [0-100] Specify the total LTR sequence content (%) in the genome instead of estimating from the -all RepeatMasker file.
-blast [path] The path to the blastn program. If left unspecified, then blastn must be accessible via shell ENV.
-t [number] Number of threads to run blastn.
-h Display this help info.
実行方法
1、create a suffix array index
#最初に必要なインデックス等を作成
gt suffixerator -db genome.fa -indexname genome_index -tis -suf -lcp -des -ssp -sds -dna
#ゲノム配列中のLTRレトロトランスポゾンをde novoで予測(manual)
gt ltrharvest -index genome_index -minlenltr 100 -maxlenltr 7000 -mintsd 4 -maxtsd 6 -motif TGCA -motifmis 1 -similar 85 -vic 10 -seed 20 -seqids yes > genome.fa.harvest.scn
#ゲノム配列中の完全長LTRレトロトランスポンソンを見つけるLTR_FINDER(開発終了)のparallel版をラン
perl LTR_FINDER_parallel/LTR_FINDER_parallel -seq genome.fa -threads 10 -harvest_out -size 1000000 -time 300
#2つのscnを結合
cat genome.fa.harvest.scn genome.fa.finder.combine.scn > genome.fa.rawLTR.scn
LTR_FINDER_parallelのランにより、genome.fa.rawLTR.scnが出力される。
2、LTR_retrieverの実行
LTR_retriever -genome genome.fa -inharvest genome.fa.rawLTR.scn -threads 20
3、LAIの実行
LAI -genome genome.fa -intact genome.fa.pass.list -all genome.fa.out
テスト
P.patensのゲノムアセンブリ(Ensembl plantに2020/11/06にアクセスして取得)を使ってLAIを算出してみた(*1)。

LAIは、raw LAIが9.86、adjusted LAIが15.49だった。
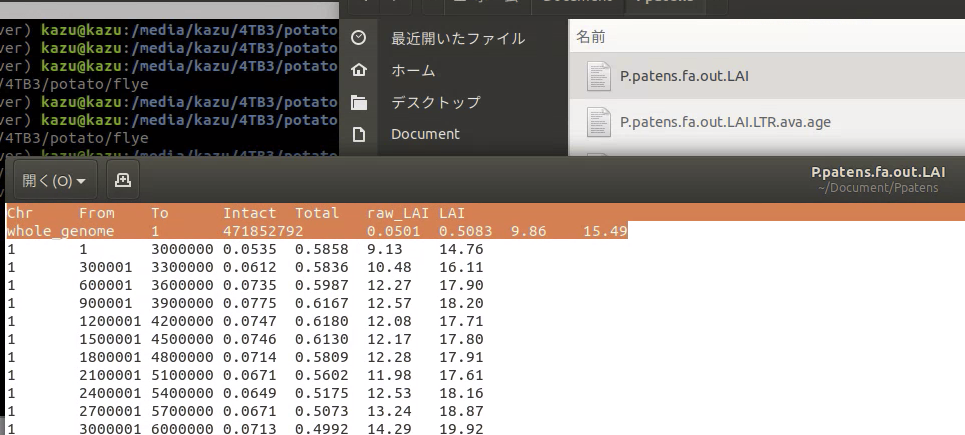
良好なスコアだった。
感想
このLAIスコアは、LTRレトロトランスポゾンがゲノム中に一定数あれば利用できる指標です。ショートリードアセンブリなどで、ゲノム中のLTRレトロトランスポゾンの再構成できている割合が低ければ小さく(5前後)、ロングリードやHi-C技術などの併用でLTRレトロトランスポゾンの再構成できている割合が高ければ大きくなります(15前後)。raw LAIは種内比較のみに利用でき(アセンブラ間の比較など)、adjusted LAIは種間比較もできる測定基準です。不完全かつ長いリピートを対象にした厳しい評価方法のため、他のアセンブリ評価手法では飽和してしまって何とも言えないようなアセンブリ(例えば飽和量ロングリードでシーケンスしたデータのアセンブリ)にも適用することができます。Hi-Cなどの技術が商用キットでも利用できるようになってきており、今後は植物でもより(Pseudo)chromosome length genome assemblyの報告が増えてくると予想されますが、そのような時代において、アセンブリ品質を客観的に判断するために重要な指標になると感じました。
引用
Assessing genome assembly quality using the LTR Assembly Index (LAI)
Shujun Ou, Jinfeng Chen, Ning Jiang
Nucleic Acids Res. 2018 Nov 30;46(21)
関連
*1
論文中ではシロイヌナズナゲノムも評価しているが、Github記載の設定でシロイヌナズナの公開ゲノムをテストすると、LTRレトロトランスポゾンが少なすぎて評価できなかった。ゲノムによってLTRレトロトランスポゾン探索のパラメータ設定最適化が必要と思われる。