2019 8/31 docker imageのhelpコマンドのエラー修正
2021 6/15 データベースのダウンロード追記
2025/01/15追記
腸内微生物叢は、ホストの健康に寄与する多様な代謝特性を付与する数百種の豊かなコミュニティを持っている(ref.1)。また、抗生物質耐性遺伝子の貯蔵庫としても機能し、数え切れないほどの細菌が絶え間なく選択されて(たとえば、食事や抗生物質を介して)、耐性遺伝子が種間で容易に移動する動的環境を作り出す(ref.3)。多剤耐性生物の有病率の増加、および細菌のclass(綱(こう))間で伝達可能なプラスミド上の耐性遺伝子のtransfer(カルバペネム耐性腸内細菌科など)により、伝播を促進するリザーバーとしての腸内微生物叢の役割は、科学的および公衆衛生上の重要な関心事になっている。
多剤耐性生物の伝播研究は主に培養分離株に依存してきたが、この問題にメタゲノム解析がますます適用されている(ref.4,5)。培養バイアスを回避することにより、メタゲノミクスは細菌および真菌株と抗生物質耐性遺伝子のより完全な見方を約束する。ただし、特に複数の株が存在する場合、短い第2世代のリードの実用的な制限とメタゲノムアセンブリの複雑さにより、アセンブリが不正確または不完全になる可能性がある(ref.6)。シーケンス構成またはカバレッジを使用したクラスタリング手法により、断片化されたアセンブリを種のビンに集約できる(ref.7,8)。大きな進歩は、これらのアイデアを複数のサンプルに拡張して、ほぼ完全な種レベルのゲノムをアセンブリすることである(ref.9,10)。これらの方法は、新種のコンセンサスゲノムに合わせて調整されたが、系統と遺伝子を正しくアセンブリするためではない(ref.11)。株の変化の領域は伝播研究の有益なマーカーとして役立つので(ref.12)、個々のサンプルの株レベルでメタゲノムアセンブリを解決する方法が依然として必要である。
ロングリードシーケンスは、個々のサンプルのメタゲノムアセンブリのあいまいさを直接解決し、アセンブリの連続性を高める(ref.13、14、15)。その使用は、高コスト、シーケンシングバイアス、厳しいDNA要件、および低いシーケンスクオリティのために制限されている。最近、ウルトラロングナノポアリードがヒトゲノムアセンブリの改善に使用され(ref.16)、大幅なスループットの改善が新しいプラットフォーム(PromethIONなど)で利用可能になった。ナノポアシーケンスは、いくつかのサンプルタイプのメタゲノムプロファイリングに使用されているが(ref.17)、ヒトメタゲノムアセンブリへのその使用は、広く調査されていない(ref.18,19)。
ここでは、臨床研究における糞便メタゲノムのナノポアシーケンスの実現可能性と有用性を確立し、メタゲノムの多様性を確実に表すロングリードを取得する。ナノポアとイルミナのリードとハイブリッドメタゲノムアセンブラを組み合わせることで、両方のシーケンスアプローチの長所を活用し(ref.20、21)、subspeciesレベル(論文補足表1)でこれまで達成できなかった高い塩基対(bp)精度とほぼ完全なゲノムの目標を達成する。これらのアセンブリは、菌株の変異や新しいプラスミドの同定など、腸の抵抗ゲノムの進化を研究するための貴重な参考資料として役立つことを実証する。
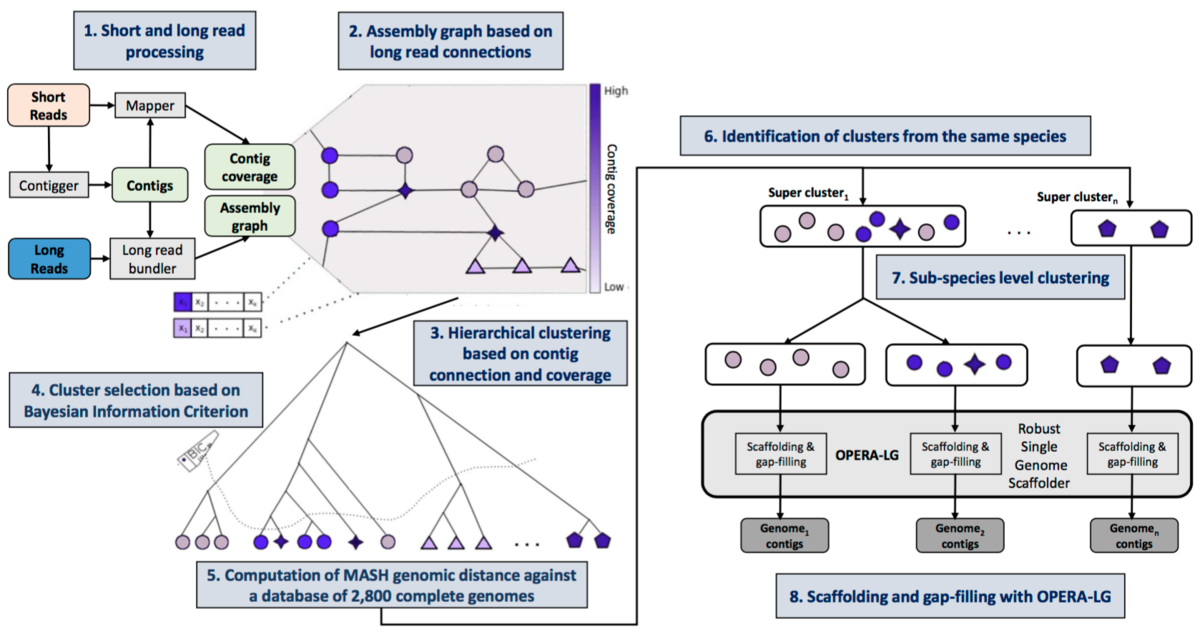
Schematic depicting the steps and workflow for OPERA-MS. Preprintより転載
MethodのセクションではStool samplesからの高品質DNAの抽出プロトコルについて記載されています。
Check out the new release of our hybrid metagenomic assembler OPERA-MS https://t.co/4TJto4lTje and please do give us feedback @dbertran78
— Denis Bertrand (@dbertran78) July 29, 2019
Excited to have our work on next-gen metagenomics with OPERA-MS finally published! Read all about it here: https://t.co/RRXMC8Puin and check out the software https://t.co/sXeVSAhdsM. Great collaboration w/ @dbertran78 @kalisvar @OonTek1 @astar_research https://t.co/dX0H2xKWYR pic.twitter.com/EQcNkKwEZS
— Niranjan Nagarajan (@NiranjanTW) July 30, 2019
インストール
All other required programs come either pre-compiled with OPERA-MS or are built during the installation process. Binaries are placed inside the utils folder:
依存
- The only true dependency is cpanm
本体 Github
git clone https://github.com/CSB5/OPERA-MS.git
cd OPERA-MS
make -j 8
#Once cpanm is installed, simply run the following command to install all the perl modules:
perl utils/install_perl_module.pl
#check dependency
perl OPERA-MS.pl CHECK_DEPENDENCY
dockerイメージビルド
git clone https://github.com/CSB5/OPERA-MS.git
cd OPERA-MS
docker build -t operams .
#ラン例
docker run -itv $PWD:/data --rm kazumax/operams perl OPERA-MS.pl --contig-file /data/final.contigs.fa --short-read1 /data/R1.fq.gz --short-read2 /data/R2.fq.gz --long-read /data/ONT.fq --out-dir /data/OPERA-MS_outdir 2> log.err
contacts: Denis Bertrand <bertrandd@gis.a-star.edu.sg>
Chengxuan Tong <Tong_Chengxuan@gis.a-star.edu.sg>
Usage:
perl OPERA-MS.pl [options] --illumina-read1 <pe1> --illumina-read2 <pe2> --long-read-file <lr> --output-directory <out_dir>
Required arguments:
--short-read1 STR fasta file of illumina read1 <pe1>
--short-read2 STR fasta file of illumina read2 <pe2>
--long-read STR fasta file of long reads <lr>
--out-dir STR output directory for scaffolding results <out_dir>
Optional arguments:
Algorithm options:
--no-ref-clustering disable reference level clustering
--no-strain-clustering disable strain level clustering
--polishing enable assembly polishing (currently using Pilon)
--long-read-mapper STR software used for long-read mapping i.e. blasr or minimap2 [blasr]
--kmer-size INT kmer value used to assemble contigs [60]
--contig-len-thr INT contig length threshold for clustering; contigs smaller than the threshold will be filtered out [500]
--contig-edge-len INT during contig coverage calculation, number of bases filtered out from each contig end, to avoid biases due to lower mapping efficiency [80]
--contig-window-len INT window length in which the coverage estimation is performed. We recommend using contig-len-thr - 2 * contig-edge-len as the value [340]
Other arguments:
--contig-file STR path to the contig file, if the short-reads have been assembled previously [default assembly using MEGAHIT]
--num-processors INT number of processors to use (note that 2 is the minimum) [2]
buildしたイメージは念のためdocker hubに置いておきます(なるべく自分でbuildしてください、古くなってる可能性があります)。
docker pull kazumax/operams
#help
docker run -it kazumax/operams perl OPERA-MS.pl -h
データベース
perl OPERA-MS.pl install-db
テストラン
cd test_files
perl ../OPERA-MS.pl \
--contig-file contigs.fasta \
--short-read1 R1.fastq.gz \
--short-read2 R2.fastq.gz \
--long-read long_read.fastq \
--out-dir RESULTS 2> log.err
#Dockerなら
cd cd OPERA-MS/test_files/
sudo docker run -itv $PWD:/data/ operams perl OPERA-MS.pl \
--contig-file /data/contigs.fasta \
--short-read1 /data/R1.fastq.gz
--short-read2 /data/R2.fastq.gz \
--long-read /data/long_read.fastq \
--out-dir /data/RESULTS 2> log.err
実行方法
configファイルをコピーしておく。
cp test_files/test.config config
ファイルを開き、必要なら直す。 入力fastq部分についてはフラグを立てることで上書きされる。オプション指定するなら直す必要はない。
ランする。ショートリードのアセンブリを別に行なっているなら、"--contig-file”フラグを立ててcontig.fastaのパスを指定する。ここではアセンブル後のpolishingもする。
perl OPERA-MS.pl \
--contig-file contigs.fasta \
--short-read1 R1.fastq.gz \
--short-read2 R2.fastq.gz \
--long-read long_read.fastq \
--out-dir RESULTS \
--num-processors 40 \
--polishing \
- --contig-file : path to the contig file, if the short-reads have been assembled previously
-
--short-read1 : path to the first read for Illumina paired-end read data (fasta/fastq/fasta.gz/fastq.gz)
-
--short-read2 : path to the second read for Illumina paired-end read data (fasta/fastq/fasta.gz/fastq.gz)
-
--long-read : path to the long-read file obtained from either Oxford Nanopore, PacBio or Illumina Synthetic Long Read sequencing (fasta/fastq)
-
--out-dir : directory where OPERA-MS results will be outputted
- --polishing enable assembly polishing (currently using Pilon)
configファイルで全てのファイルとパラメータを一括指定することもできる(参考 test.config)。
引用
Hybrid metagenomic assembly enables high-resolution analysis of resistance determinants and mobile elements in human microbiomes
Denis Bertrand, Jim Shaw, Manesh Kalathiyappan, Amanda Hui Qi Ng, M. Senthil Kumar, Chenhao Li, Mirta Dvornicic, Janja Paliska Soldo, Jia Yu Koh, Chengxuan Tong, Oon Tek Ng, Timothy Barkham, Barnaby Young, Kalisvar Marimuthu, Kern Rei Chng, Mile Sikic & Niranjan Nagarajan
Nature Biotechnology volume 37, pages937–944 (2019)
関連