2019 5/8 インストール追記, 11/29 インストール追記
2020 7/12 仮想環境を作ってインストールするように変更, help追記(コメントいただいたstepも修正しました。)。8/11 リンク追加、誤字修正
2023/01/01 追記、diamond 追記
2023/04/15 step2のコマンドの誤り修正
2023/11/15 追記
TransDecoderはアセンブリなどで作ったDNAもしくはcDNA配列からコード領域を見つけるツール。 RNA seq実験でdo novo assemblyした配列や、cuflinksなどのgenome guide assemblyなツールで作った配列からコード領域を探す時などに使われる。trinityや Trinotateにも取り込まれている。*1
マニュアル
https://transdecoder.github.io
インストール
Bioconda (link)
mamba create -n TransDecoder -y
conda activate TransDecoder
mamba install -c bioconda -y TransDecoder
#homebrew
brew install TransDecoder
#hmmer(optional)
> TransDecoder.LongOrfs -h
$ TransDecoder.LongOrfs -h
########################################################################################
# ______ ___ __
# /_ __/______ ____ ___ / _ \___ _______ ___/ /__ ____
# / / / __/ _ `/ _\(_-</ // / -_) __/ _ \/ _ / -_) __/
# /_/ /_/ \_,_/_//_/___/____/\__/\__/\___/\_,_/\__/_/ .LongOrfs
#
########################################################################################
#
# Transdecoder.LongOrfs|http://transdecoder.github.io> - Transcriptome Protein Prediction
#
#
# Required:
#
# -t <string> transcripts.fasta
#
# Optional:
#
# --gene_trans_map <string> gene-to-transcript identifier mapping file (tab-delimited, gene_id<tab>trans_id<return> )
#
# -m <int> minimum protein length (default: 100)
#
# -G <string> genetic code (default: universal; see PerlDoc; options: Euplotes, Tetrahymena, Candida, Acetabularia)
#
# -S strand-specific (only analyzes top strand)
#
# --output_dir | -O <string> path to intended output directory (default: basename( -t val ) + ".transdecoder_dir")
#
# --genetic_code <string> Universal (default)
#
# Genetic Codes (derived from: https://www.ncbi.nlm.nih.gov/Taxonomy/Utils/wprintgc.cgi)
#
Acetabularia
Candida
Ciliate
Dasycladacean
Euplotid
Hexamita
Mesodinium
Mitochondrial-Ascidian
Mitochondrial-Chlorophycean
Mitochondrial-Echinoderm
Mitochondrial-Flatworm
Mitochondrial-Invertebrates
Mitochondrial-Protozoan
Mitochondrial-Pterobranchia
Mitochondrial-Scenedesmus_obliquus
Mitochondrial-Thraustochytrium
Mitochondrial-Trematode
Mitochondrial-Vertebrates
Mitochondrial-Yeast
Pachysolen_tannophilus
Peritrich
SR1_Gracilibacteria
Tetrahymena
Universal
#
#
# --version show version tag (5.5.0)
#
#########################################################################################
> TransDecoder.Predict -h
$ TransDecoder.Predict -h
########################################################################################
# ______ ___ __
# /_ __/______ ____ ___ / _ \___ _______ ___/ /__ ____
# / / / __/ _ `/ _\(_-</ // / -_) __/ _ \/ _ / -_) __/
# /_/ /_/ \_,_/_//_/___/____/\__/\__/\___/\_,_/\__/_/ .Predict
#
########################################################################################
#
# Transdecoder.LongOrfs|http://transdecoder.github.io> - Transcriptome Protein Prediction
#
#
# Required:
#
# -t <string> transcripts.fasta
#
# Common options:
#
#
# --retain_long_orfs_mode <string> 'dynamic' or 'strict' (default: dynamic)
# In dynamic mode, sets range according to 1%FDR in random sequence of same GC content.
#
#
# --retain_long_orfs_length <int> under 'strict' mode, retain all ORFs found that are equal or longer than these many nucleotides even if no other evidence
# marks it as coding (default: 1000000) so essentially turned off by default.)
#
# --retain_pfam_hits <string> domain table output file from running hmmscan to search Pfam (see transdecoder.github.io for info)
# Any ORF with a pfam domain hit will be retained in the final output.
#
# --retain_blastp_hits <string> blastp output in '-outfmt 6' format.
# Any ORF with a blast match will be retained in the final output.
#
# --single_best_only Retain only the single best orf per transcript (prioritized by homology then orf length)
#
# --output_dir | -O <string> output directory from the TransDecoder.LongOrfs step (default: basename( -t val ) + ".transdecoder_dir")
#
# -G <string> genetic code (default: universal; see PerlDoc; options: Euplotes, Tetrahymena, Candida, Acetabularia, ...)
#
# --no_refine_starts start refinement identifies potential start codons for 5' partial ORFs using a PWM, process on by default.
#
## Advanced options
#
# -T <int> Top longest ORFs to train Markov Model (hexamer stats) (default: 500)
# Note, 10x this value are first selected for removing redundancies,
# and then this -T value of longest ORFs are selected from the non-redundant set.
# Genetic Codes
#
#
# --genetic_code <string> Universal (default)
#
# Genetic Codes (derived from: https://www.ncbi.nlm.nih.gov/Taxonomy/Utils/wprintgc.cgi)
#
#
Acetabularia
Candida
Ciliate
Dasycladacean
Euplotid
Hexamita
Mesodinium
Mitochondrial-Ascidian
Mitochondrial-Chlorophycean
Mitochondrial-Echinoderm
Mitochondrial-Flatworm
Mitochondrial-Invertebrates
Mitochondrial-Protozoan
Mitochondrial-Pterobranchia
Mitochondrial-Scenedesmus_obliquus
Mitochondrial-Thraustochytrium
Mitochondrial-Trematode
Mitochondrial-Vertebrates
Mitochondrial-Yeast
Pachysolen_tannophilus
Peritrich
SR1_Gracilibacteria
Tetrahymena
Universal
#
# --version show version (5.5.0)
#
#########################################################################################
動作に必要なcd-hitもインストールされる。テストデータやユーティリティコマンド群を使うならソースコードもダウンロードする。
https://github.com/TransDecoder/TransDecoder/releases
git clone https://github.com/TransDecoder/TransDecoder.git
cd TransDecoder/util/

> perl gtf_genome_to_cdna_fasta.pl -h
$ perl gtf_genome_to_cdna_fasta.pl -h
usage: gtf_genome_to_cdna_fasta.pl cufflinks.gtf genome.fasta
> perl gtf_to_alignment_gff3.pl
$ perl gtf_to_alignment_gff3.pl
usage: gtf_to_alignment_gff3.pl cufflinks.gtf
実行方法
1、de novo assemblyで作ったfastaを使う。
sample-dataのTrinityのアセンブルデータからコード領域を探す。(TransDecoder-3.0.1/sample_data/simple_transcriptome_target/Trinity.fasta)。
step1
コード領域を探索する。
TransDecoder.LongOrfs -G universal -m 100 -t Trinity.fasta
- -t transcripts.fasta
- -m minimum protein length (default: 100)
- -S strand-specific (only analyzes top strand)
- -G genetic code (default: universal)
デフォルトでは100アミノ酸以上のORFが抽出される。strand specific sequenceなどでリード鎖が決まっている場合-Sをつける。
解析が終わると、Trinity.fasta.transdecoder_dir/ができる。中身は以下のようになった。
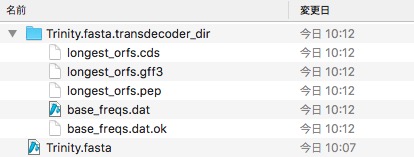
100アミノ酸以上になるcode領域を記載したgff3ファイルができる。またそのアミノ酸配列ファイル(.pep)もできている。
step2 (option)
見つかったコード領域をクエリにしてデータベースを検索。
blastxで相同なタンパク質を検索。(公式マニュアルの例)
blastp -query transdecoder_dir/longest_orfs.pep -db uniprot_sprot.fasta -max_target_seqs 1 -outfmt 6 -evalue 1e-5 -num_threads 10 > blastp.outfmt6
Pfamで相同なタンパク質を検索(データベース作成)。(公式マニュアルの例)(*2 databaseについて)
hmmscan --cpu 12 --domtblout pfam.domtblout /path/to/Pfam-A.hmm transdecoder_dir/longest_orfs.pep >& hmmscan_log
- --cpu Set the number of parallel worker threads to
- --domtblout Save a simple tabular (space-delimited) file summarizing the per-domain output, with one data line per homologous domain detected in a query sequence for each homologous model.
追記
遅いならDIamond blasstpを使う(モデル生物から遠いなら推奨されない)。
diamond makedb --in plasnts_orthoDB.faa -d diamondDB
diamond blastp --query transdecoder_dir/longest_orfs.pep --db diamondDB --evalue 1e-5 --sensitive --max-target-seqs 1 --outfmt 6 --threads 30 > diamond.blastp.outfmt6
step3
blastやpfamの結果をもとに妥当なcode領域を選抜する。
TransDecoder.Predict -t target_transcripts.fasta --retain_blastp_hits blastp.outfmt6
#転写産物配列1つにつきベストの1つのみにするには"--single_best_only"をつける。
TransDecoder.Predict --single_best_only -t target_transcripts.fasta --retain_blastp_hits blastp.outfmt6
- -t transcripts.fasta
- -G genetic code (default: universal)
- --retain_pfam_hits domain table output file from running hmmscan to search Pfam.
- --retain_blastp_hits blastp output in '-outfmt 6' format.
- --single_best_orf Retain only the single best ORF per transcript. (Best is defined as having (optionally pfam and/or blast support) and longest orf)
Trinity.fasta.transdecoder_dir/にさらに中間ファイルが作成され、最終的にカレントディレクトリに上記の条件を元に選ばれたcode領域と除去されたcode領域のスコアを記載したファイルが出力される(下の写真の一番上の4つのファイル)。

一番上に.bed、.gff3、.pep(fasta)、.cds(fasta)ができている。
gff3とbedには最終的に選ばれた領域を記載されている。また最終的に選ばれたコード領域のアミノ酸配列は.pepに記載され、コード領域の塩基配列は.cdsに記載され出力される。
この最終段階では選ばれなかったコード領域のアミノ酸配列は完全に除去されている。
igvで結果を確認する。
igv -g Trinity.fasta Trinity.fasta.transdecoder.bed
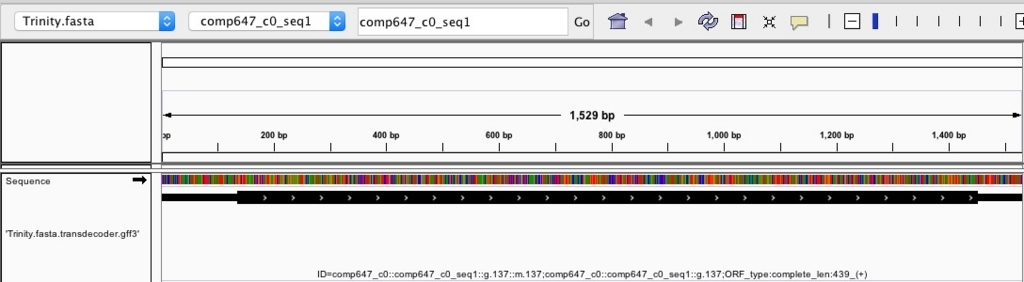
太い部分が予想されたコード領域になる。
ORF type:completeとあるのが完全長(開始コドンとストップコドンがある)の配列。

2、genome guide assemblyで作った gtfファイルと参照したgenome配列を使う。
sample-dataのcufflinksのゲノムガイドアセンブルで作られたgtfからコード領域を探す。(TransDecoder-3.0.1/sample_data/cufflinks_example/)。
step1
gtfで指定した領域をfastaにして抽出する。
util/gtf_genome_to_cdna_fasta.pl transcripts.gtf test.genome.fasta > transcripts.fasta
transcripts.fastaができる。このコマンドはgtfからfasta配列を作るツールとしても使えそうである。
step2
gff3も作る。最後に必要。
util/gtf_to_alignment_gff3.pl transcripts.gtf > transcripts.gff3
コマンドを間違って記載しておりました。修正しました。hosaka_TE様、ありがとうございます。(2023/04/15)。
step3
コード領域を探索する。de novo で始めた場合のstep1にあたる。
TransDecoder.LongOrfs -t transcripts.fasta
解析が終わると、transcripts.fasta.transdecoder_dir/ができる。

step4
妥当なcode領域を選抜する。de novo で始めた場合のstep3にあたる。
TransDecoder.Predict -t transcripts.fasta
#blastサーチ結果があるなら指定することで判定精度が上がる(任意)
TransDecoder.Predict -t stringtie2.fasta --retain_blastp_hits blast.outfmt6
カレントディレクトリに.gff3、.bed、.fasta、.cdsファイルが出力される。

step5
最後にゲノムベースのコード領域のアノテーションファイル(gff3)を作る。
util/gtf_to_alignment_gff3.pl transcripts.fasta.transdecoder.gff3 transcripts.gff3 transcripts.fasta > transcripts.fasta.transdecoder.genome.gff3

igvで結果を確認する。
igv -g test.genome.fasta transcripts.gtf,transcripts.fasta.transdecoder.genome.gff3

上がcufflinksのmergeで作ったgtfファイル(transcripts.gtf)、下がコード領域を予想して最終的に得られたtranscripts.fasta.transdecoder.genome.gff3。太い部分が予想されたコード領域である。
igvについては以前紹介しています。
こちらのツールも確認してみて下さい。
引用
De novo transcript sequence reconstruction from RNA-Seq: reference generation and analysis with Trinity
Brian J. Haas,#1,# Alexie Papanicolaou,#2 Moran Yassour,1,3 Manfred Grabherr,4 Philip D. Blood,5 Joshua Bowden,6 Matthew Brian Couger,7 David Eccles,8 Bo Li,9 Matthias Lieber,10 Matthew D. MacManes,11 Michael Ott,2 Joshua Orvis,12 Nathalie Pochet,1,13 Francesco Strozzi,14 Nathan Weeks,15 Rick Westerman,16 Thomas William,17 Colin N. Dewey,9,18 Robert Henschel,19 Richard D. LeDuc,19 Nir Friedman,3 and Aviv Regev1,20,#
Nat Protoc. 2013 Aug; 8(8): 10.1038/nprot.2013.084.
関連
*1
サポートされているgenetic code (v 3.01)
universal (default)
Euplotes
Tetrahymena
Candida
Acetabularia
Mitochondrial-Canonical
Mitochondrial-Vertebrates
Mitochondrial-Arthropods
Mitochondrial-Echinoderms
Mitochondrial-Molluscs
Mitochondrial-Ascidians
Mitochondrial-Nematodes
Mitochondrial-Platyhelminths
Mitochondrial-Yeasts
Mitochondrial-Euascomycetes
Mitochondrial-Protozoans
デフォルトではuniversalになっており、-Gで変更できる。
*2
ここではデータベースとしてPfam FTPサーバ (link:/pub/databases/Pfam/current_release)からPfam-A.hmm.gzをダウンロードして使用した(Pfam-A.fullではない方)。hmmfetchコマンドが使えると思われるが、ここでは手動で実行した。
wget ftp://ftp.ebi.ac.uk/pub/databases/Pfam/current_release/Pfam-A.hmm.gz
#full
wget ftp://ftp.ebi.ac.uk/pub/databases/Pfam/current_release/Pfam-A.full.gz
解凍してindexを作成
hmmpress Pfam-A.hmm