2021/11/17 dockerについて追記
2023/08/08 引用修正
Funannotateはゲノム予測、アノテーション、比較のためのソフトウェアパッケージである。元々は真菌ゲノム(真核生物の中では小さいもので30 Mb程度のゲノム)のアノテーション用に書かれていたが、より大きなゲノムにも対応できるように進化してきた。このソフトウェアパッケージは、NCBI GenBankにsubmitするためのゲノムのアノテーションを正確かつ簡単に行うことができるようにするために開発された。既存のツール(Makerなど)では、GenBankへの投稿ルールに準拠するために、かなりの手作業での編集が必要となるが、funannotateはゲノム投稿プロセスを簡素化することを目的としている。
また、Funannotateは軽量な比較ゲノムプラットフォームでもある。funannotate annotateコマンドで機能的アノテーションを追加したゲノムは、funannotate compareスクリプトでhtmlベースの全ゲノム比較を実行することができる。このソフトウェアは、オーソロガスクラスタリングの実行、全ゲノム系統図の構築、Gene Ontologyエンリッチメント解析の実行、正の選択下でのオーソロガスクラスタのdN/dS比の計算を行うことができる。
Documentation
Installation — Funannotate 1.7.0 documentation
インストール
#bioconda (link)
mamba create -n funannotate python=3.7 -y
conda activate funannotate
mamba install -c bioconda -y funannotate
> funannotate
$ funannotate
Usage: funannotate <command> <arguments>
version: 1.8.1
Description: Funannotate is a genome prediction, annotation, and comparison pipeline.
Commands:
clean Find/remove small repetitive contigs
sort Sort by size and rename contig headers
mask Repeatmask genome assembly
train RNA-seq mediated training of Augustus/GeneMark
predict Run gene prediction pipeline
fix Fix annotation errors (generate new GenBank file)
update RNA-seq/PASA mediated gene model refinement
remote Partial functional annotation using remote servers
iprscan InterProScan5 search (Docker or local)
annotate Assign functional annotation to gene predictions
compare Compare funannotated genomes
util Format conversion and misc utilities
setup Setup/Install databases
test Download/Run funannotate installation tests
check Check Python, Perl, and External dependencies [--show-versions]
species list pre-trained Augustus species
database Manage databases
outgroups Manage outgroups for funannotate compare
Written by Jon Palmer (2016-2019) nextgenusfs@gmail.com
> funannotate predict
$ funannotate predict
Usage: funannotate predict <arguments>
version: 1.8.1
Description: Script takes genome multi-fasta file and a variety of inputs to do a comprehensive whole
genome gene prediction. Uses AUGUSTUS, GeneMark, Snap, GlimmerHMM, BUSCO, EVidence Modeler,
tbl2asn, tRNAScan-SE, Exonerate, minimap2.
Required:
-i, --input Genome multi-FASTA file (softmasked repeats)
-o, --out Output folder name
-s, --species Species name, use quotes for binomial, e.g. "Aspergillus fumigatus"
Optional:
-p, --parameters Ab intio parameters JSON file to use for gene predictors
--isolate Isolate name, e.g. Af293
--strain Strain name, e.g. FGSCA4
--name Locus tag name (assigned by NCBI?). Default: FUN_
--numbering Specify where gene numbering starts. Default: 1
--maker_gff MAKER2 GFF file. Parse results directly to EVM.
--pasa_gff PASA generated gene models. filename:weight
--other_gff Annotation pass-through to EVM. filename:weight
--rna_bam RNA-seq mapped to genome to train Augustus/GeneMark-ET
--stringtie StringTie GTF result
-w, --weights Ab-initio predictor and EVM weight. Example: augustus:2 or pasa:10
--augustus_species Augustus species config. Default: uses species name
--min_training_models Minimum number of models to train Augustus. Default: 200
--genemark_mode GeneMark mode. Default: ES [ES,ET]
--genemark_mod GeneMark ini mod file
--busco_seed_species Augustus pre-trained species to start BUSCO. Default: anidulans
--optimize_augustus Run 'optimze_augustus.pl' to refine training (long runtime)
--busco_db BUSCO models. Default: dikarya. `funannotate outgroups --show_buscos`
--organism Fungal-specific options. Default: fungus. [fungus,other]
--ploidy Ploidy of assembly. Default: 1
-t, --tbl2asn Assembly parameters for tbl2asn. Default: "-l paired-ends"
-d, --database Path to funannotate database. Default: $FUNANNOTATE_DB
--protein_evidence Proteins to map to genome (prot1.fa prot2.fa uniprot.fa). Default: uniprot.fa
--protein_alignments Pre-computed protein alignments in GFF3 format
--p2g_pident Exonerate percent identity. Default: 80
--p2g_diamond_db Premade diamond genome database for protein2genome mapping
--transcript_evidence mRNA/ESTs to align to genome (trans1.fa ests.fa trinity.fa). Default: none
--transcript_alignments Pre-computed transcript alignments in GFF3 format
--augustus_gff Pre-computed AUGUSTUS GFF3 results (must use --stopCodonExcludedFromCDS=False)
--genemark_gtf Pre-computed GeneMark GTF results
--min_intronlen Minimum intron length. Default: 10
--max_intronlen Maximum intron length. Default: 3000
--soft_mask Softmasked length threshold for GeneMark. Default: 2000
--min_protlen Minimum protein length. Default: 50
--repeats2evm Use repeats in EVM consensus model building
--repeat_filter Repetitive gene model filtering. Default: overlap blast [overlap,blast,none]
--keep_no_stops Keep gene models without valid stops
--keep_evm Keep existing EVM results (for rerunning pipeline)
--SeqCenter Sequencing facilty for NCBI tbl file. Default: CFMR
--SeqAccession Sequence accession number for NCBI tbl file. Default: 12345
--force Annotated unmasked genome
--cpus Number of CPUs to use. Default: 2
ENV Vars: If not specified at runtime, will be loaded from your $PATH
--EVM_HOME
--AUGUSTUS_CONFIG_PATH
--GENEMARK_PATH
--BAMTOOLS_PATH
データベースの準備
funannotate setup -i all -d funannotate_database
# $FUNANNOTATE_DBを設定
export FUNANNOTATE_DB=/<your>/<funannotate>/<download>/<dir>
実行方法
funannotate predict - 遺伝子予測
ゲノムと推定transcritsのFASTAファイルを指定する。genome.fastaのヘッダー名が長いとエラーを起こすので注意する。
funannotate predict -i inputgenome.fasta --species "Genome awesomenous" --isolate T12345 \
--transcript_evidence trinity.fasta --rna_bam alignments.bam -o outdir --cpus 20
以下のステップで実行される。
- Align Transcript Evidence to genome using minimap2
- Align Protein Evidence to genome using Diamond/Exonerate.
- Parse BAM alignments generating hints file
- Parse PASA gene models and use to train/run Augustus, snap, GlimmerHMM
- Extract high-quality Augustus predictions (HiQ)
- Run Stringtie on BAM alignments, use results to run CodingQuarry
- Pass all data to Evidence Modeler and run
- Filter gene models (length filtering, spanning gaps, and transposable elements)
- Predict tRNA genes using tRNAscan-SE
- Generate an NCBI annotation table (.tbl format)
- Convert to GenBank format using tbl2asn
- Parse NCBI error reports and alert user to invalid gene models
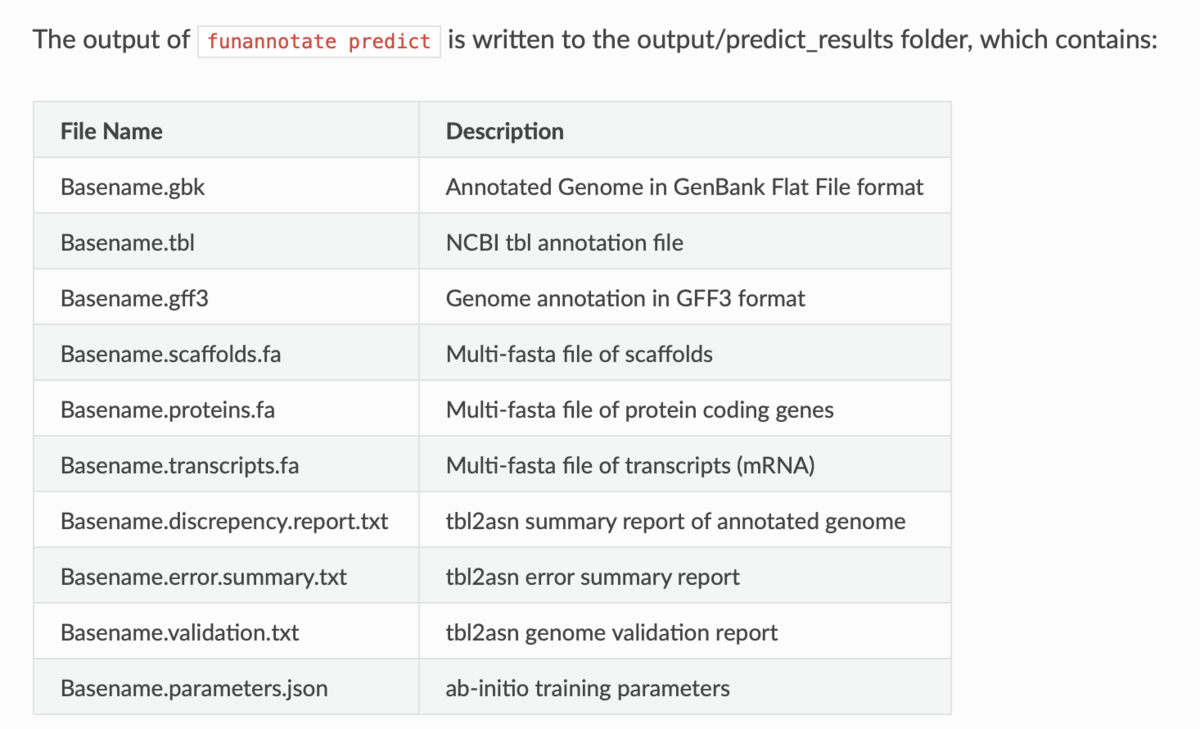
出力

出力ファイル(Githubより)
PASAで得たGFF3ファイルも指定する(--pasa_gff)。
funannotate predict -i inputgenome.fasta --species "Genome awesomenous" --isolate T12345 \
--transcript_evidence trinity.fasta --rna_bam alignments.bam --pasa_gff pasa.gff3 -o outdir --cpus 20
pre-trainされたAugustusのトレーニングファイル(hint file)を使用することもできる(ランタイムが大きく短縮される)。まず利用できるセットを確認する。
funannotate species
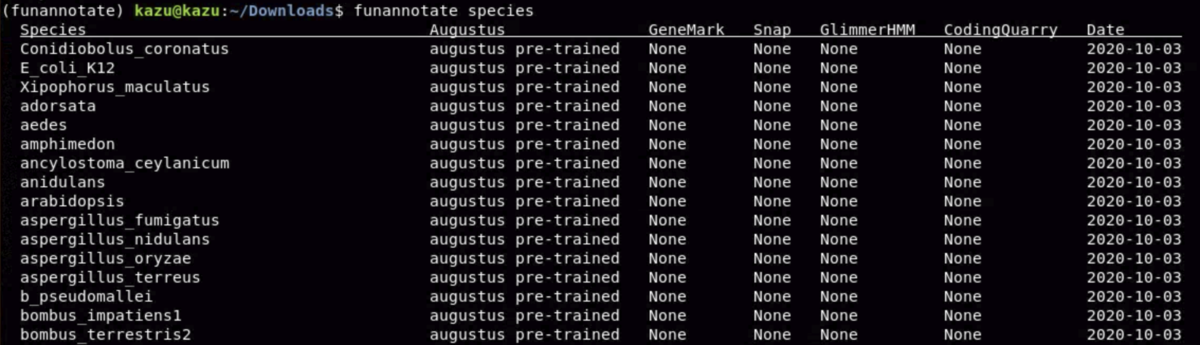
(以下省略)
実行する(bam指定は不要)。
funannotate predict -i inputgenome.fasta -o outdir -s "Aspergillus nidulans" --augustus_species anidulans --cpus 20
Augustus or GeneMarkのアノテーションファイルを持っている場合、--augustus_gffと--genemark_gtfで指定する。
funannotate predict -i inputgenome.fasta -o outdir -s "Aspergillus nidulans" --augustus_gff augustus.gff --genemark_gtf genemark.gtf --cpus 20
Evidence Modelerはエビデンスの各セットにウエイトをつける。デフォルトではab initioの遺伝子予測と転写産物/タンパク質のアラインメントの入力は1に設定されている。PASAの高品質遺伝子モデルを--pasa_gffで渡した場合、デフォルトでは6の重みが設定される。 一方、別のGFFファイルからのエビデンスを--other_gffで渡した場合、それらのモデルはデフォルトでは1に設定される。入力にコロンを使用することで、PASAのエビデンスと他のエビデンスの両方の重みを制御することができる。また、-w, -weightsオプションを利用してab-initioツールの重みを制御することができる。
funannotate predict -i inputgenome.fasta -o outdir -s "Aspergillus nidulans" --pasa_gff mypasamodels.gff3:8 --other_gff prediction.gff3:5
#multiple GFF files can be passed to --other_gff funannotate predict -i inputgenome.fasta -o outdir -s "Aspergillus nidulans" --pasa_gff mypasamodels.gff3:8 --other_gff prediction1.gff3:5 prediction2.gff3:1
#controlling the weights directly funannotate predict -i inputgenome.fasta -o outdir -s "Aspergillus nidulans" --weights augustus:2 pasa:8 snap:1
docker
dockerを使ってfunannotateを実行することもできる(GeneMarkがdockerイメージに含まれていない点に注意)
docker pull nextgenusfs/funannotate
wget -O funannotate-docker https://raw.githubusercontent.com/nextgenusfs/funannotate/master/funannotate-docker
chmod +x /path/to/funannotate-docker
#test run
./funannotate-docker test -t predict --cpus 12


sudo docker run -itv $PWD:/data -w /data --rm nextgenusfs/funannotate:latest funannotate predict -i softmasked.fa -o outdir --cpus 40 --species "Genome awesomenous" --isolate T12345
引用
Palmer J. 2016. Funannotate: pipeline for genome annotation.
https://zenodo.org/record/2604804#.ZNIMJC_3Ia8
関連