2022/06/26 追記
分子生物学、とりわけ、微生物群集(メタゲノミクス分野)のハイスループットシーケンシングは、気候変動、環境汚染、人間の健康などに関わる微生物群集の組成と機能的内容の理解を急速に進歩させている。メタゲノミクスは、以前は実験室制御の培養に難しかったネイティブにサンプリングされた微生物群集の分類学的(すなわち、どのような微生物が存在するか)および機能的(すなわち、微生物が何をしているか)の最も完全な絵を研究者に提供する。現在、これらは地球上のすべての微生物の大部分を占めている。環境サンプルに含まれる全ての生物は、サンプル非依存性コミュニティの機能的な内容を調査するために、分類学的または全ゲノムのショットガンベースの方法を調査するために、培養に依存しない方法で、最も頻繁には16Sリボソームアンプリコン法でシーケンシングされる。メタゲノミクスは、研究者が微生物群集のコミュニティ構成と機能的内容を特徴付けることを可能にするが、どの機能的プロセスが活性であるかを示すことはできない。しかし、transcriptomicsの平行進化により、この分野でも知見が劇的に増加することが約束される。
2008年から、MG-RAST(Meyer et al、BMC Bioinformatics 9:386、2008)は、メタゲノム配列データのアノテーションと分析のための公共リソースとして役立っており、現在150,000以上のデータセットを提供している。 MG-RAST、またはメタゲノミクスRASTサーバーは、ユーザーが(好ましくは)fastqまたはfasta形式で生メタデータとシーケンシングデータをアップロードすることを可能にする。ポストアノテーション分析と視覚化は、Webインターフェイスを介して直接行うことが可能で、さらに、MG-RAST APIを利用するmatRを使用して、MG-RAST処理パイプラインの任意のステージからデータを簡単にダウンロードすることもできる。MG-RASTは、メタゲノミクスおよび関連する分子化学(例えば、メタトランスクリプトーム)における絶えず進化し続ける発展に伴って大幅な改訂を行った。

v2とv3のワークフロー。マニュアルより転載。
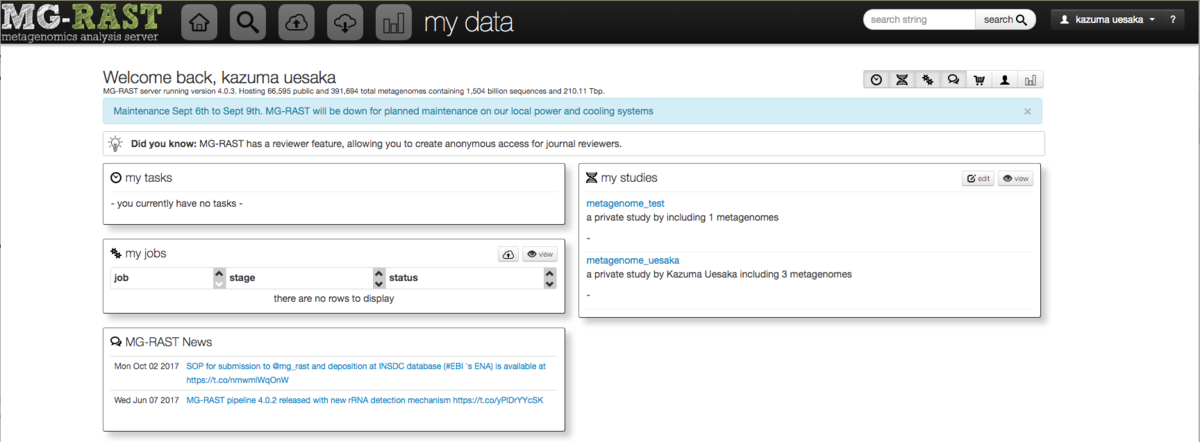
MG-RAST
マニュアル
ftp://ftp.metagenomics.anl.gov/data/manual/mg-rast-manual.pdf#page=28
使い方
パブリックデータを閲覧する機能と、マイデータをアップロードして分析する機能がある。
https://www.mg-rast.org/mgmain.html?mgpage=mydataにアクセスする。
初回はアカウント作成する。
新しく解析を始めるには上のアップロードボタンをクリックする。

fastq(.gzも対応)をアップロードする。左上の↑ボタンをクリックしてfastqを選択する。複数同時選択も可。
右側のウィンドウに表示されるのでstart ボタンをおしてアップロードする。

このステップはかなりの時間がかかる。1GB程度なら1時間くらいでアップできるが、データサイズがHiseqクラスのサンプルとなると、サンプル数が多ければ1週間くらいはかかるかもしれない。落ち着いて進めること。
また、たまに接続が切れることがあるので、その時はやり直す(途中から継続できる)。
右上のboxには常に対応したhelpが表示されるので、わからないことがあれば積極的に調べていく。

全てUPし終えたら、サンプルの詳細な情報をまとめたmetadataをアップロードする( *1)。
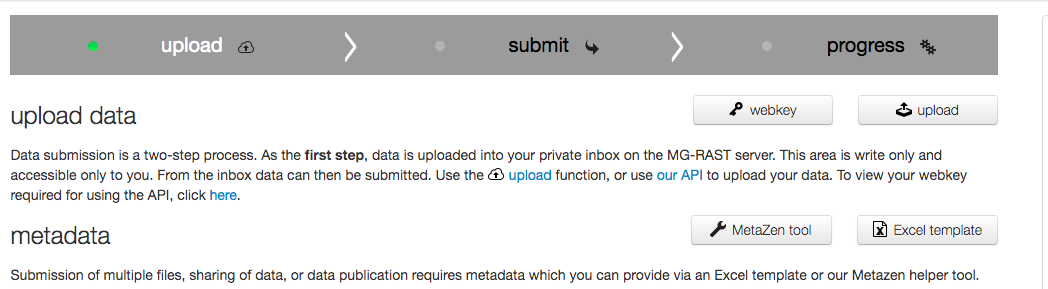
submitに切り替える。submitをクリック。
プロジェクト名、どのサンプルを使うかなどを順番に指定していく。先ほどのステップでmetadataをアップしていない場合は、ここで用意することもできる。

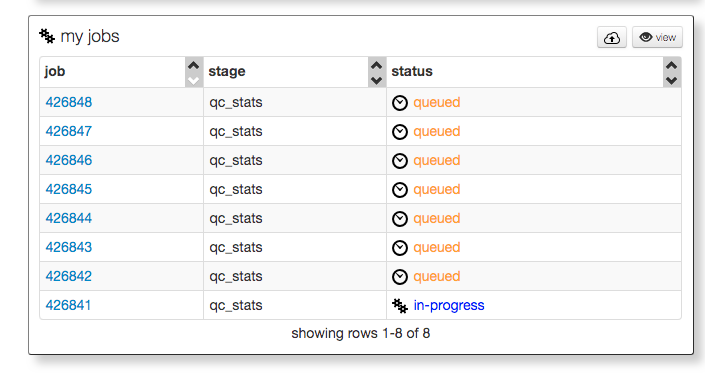
最後にsubmitをクリック。問題なければジョブが開始される。
進捗は画面で確認できるようになっている。
パブリックデータはサーチから探せる。
https://www.mg-rast.org/mgmain.html?mgpage=search
自分のデータを非公開にしていても、ログインIDが同じならばここに表示される。勘違いしないこと。
2022/06/26 追記
サーチでは、右端のメニューで絞り込み検索できる。門レベルでシアノバクテリアのアバンダンス10%以上、光合成遺伝子を含むで検索した。

805ヒットした。

ヒットした中には、非公開データ(ダウンロードできない)、アセンブリ配列が登録されているものなども含まれる点に注意。
引用
MG-RAST, a Metagenomics Service for Analysis of Microbial Community Structure and Function.
Keegan KP, Glass EM, Meyer F
Methods Mol Biol. 2016;1399:207-33.
*1
メタデータは非常に細かな項目まで記入することができますが、その分、登録する時はかなり苦労します。個人的には、しんどさはゲノム登録と同じくらいです。半日使う覚悟で気合いを入れて取り組んでください。
例を見たい方は、サーチでから自分のサンプルに似た公共データを探し、そのメタデータを参考にすると良いでしょう。サンプル一覧の右端のカラムからダウンロードできます。