10/5 3stepコマンドの誤り修正 及びコマンド変更、コメント追加
正確で完全でアノテーションのついたゲノムは、種や個体の過去、現在、未来に関する豊富な情報を提供するため、医療や生物学の研究にとって非常に貴重なリソースとなっている[論文より ref.1]。過去10年にわたるDNAシーケンシング技術の進歩により、ゲノムの構造と系統学、そして遺伝子の機能、RNAおよび他のゲノム特徴を含んだ、生命の樹木(tree of life)の多様な枝の多種多様な生物のゲノムシーケンシングとアセンブリが可能になっている。少なくとも染色体レベルの分解能を持つアセンブリは、特に、遺伝子座が染色体に沿ってどのように整列して配向されているかに関して情報をもたらすため、ゲノム生物学を理解する上で重要である。したがって、染色体レベルのアセンブリは「gold standard」として熱望されているが、長く連続したストレッチをアセンブリするのは難しいため、このスタンダードには届きにくい[ref.3]。今日、ますます多くのゲノムがシーケンシングされ、染色体レベルにアセンブリされているが、大きなゲノムのアセンブリ配列はしばしば高度に断片化されたままである[ref.4]。したがって、アセンブリの連続性 (contiguity) の改善は、ゲノム研究における中心的な問題である。改良された連続性は、アセンブリ全体にわたる遺伝子およびgenomic elementsの完全性を高め、より完全でより完全なアノテーションおよび下流分析を促進する。したがって、アセンブリを評価するための主要な指標の1つとして、連続性が提案されている[ref.5、6]。
シーケンシング技術とゲノムアセンブリアプローチの近年の進歩にもかかわらず、ショートリードからラージゲノムの連続したアセンブリを得ることは困難なままになっている。この理由から、ラージゲノムの連続したアセンブリを提供できる新しい手段やシークエンシング技術は、ゲノミクスコミュニティにとって大きな関心事である。 高品質のロングインサートクローンやsingle-molecule restriction maps [ref.12 optical mapping]と同様に、PacBio [ref.7]やNanopore [ref.8]などの第3世代ロングリードシークエンシング技術は、単独、またはショートリードデータ[ref.9-11]と組み合わせ、より連続したゲノムアセンブリが達成される手段を提供している[ref.13]。しかし、これらのアプローチの利点は単純なショートリードのショットガンシーケンシング技術よりも高コストなことも伴っている。
特にショートリードのみを使用する場合、連続したゲノムのアセンブリを行うための最大の障害は、複雑さの低い領域(low-complexity regions)と転移因子である[ref.14]。いくつかの脊索動物と植物の場合、これらの領域は総ゲノムサイズの50%以上を占める可能性がある[ref.15]。これらの領域は、ゲノム全体に散在する多くの非常に類似したコピーが、基礎をなすアセンブリグラフに多数のあいまいでしばしば解決不可能なグラフをもたらすため、複雑なデノボアセンブリをより複雑にしてアセンブリを妨げてしまう。結果として、得られたゲノムアセンブリは断片化され、さらなる分析への利用が制限される。
連続性を高めるために、染色体レベルのゲノムアセンブリが入手可能なclosely relatedな種からsyntenic information をインポートすることができる[ref.16 pubmed]。Reference-assistedアセンブリは、ゲノム再編成や遺伝子重複により時々エラーを引き起こすが、アセンブリ断片化を大幅に減らし、そのためより効率的にアノテーションやゲノムの特徴の分析を行えるようになる[ref.16,17]。ゲノムアセンブリは、追加のトランスクリプトーム[ref.18 pubmed ,19 link](紹介)またはプロテオームデータ[ref.20 pubmed,21 pubmed]を使用してさらに最適化することができるが、大きなゲノムに関してはそれでも難しく、特に低いカバレッジシーケンシングデータおよび/または遠いリファレンスアセンブリしか利用可能できない場合は困難になる。したがって、ゲノムアセンブリにおける不十分な連続性は、高品質のゲノムリファレンスおよび包括的にアノテーションされた遺伝子レパートリーの探求において永続的な制限因子である[ref.22]。
ペアエンドシーケンシングは通常500bp以下のインサートサイズに制限されているため、長いリピート領域を再解決する場合は効果がないが、メイトペアシーケンシングは数キロbpにまたがることがある。Small、medium、largeインサートサイズのメイトペアライブラリーの効果的な使用により、ラージゲノムのアセンブリが劇的に改善されている[ref.23、24]。いくつかのde novoゲノムアセンブラは、今日、ロングインサートのメイトペア情報を利用することができ、大きなインサートサイズのライブラリー(20〜25kb)があれば、連続性 (contiguity) を大幅に高められる。総合的には、十分なペア情報があれば、より大きなを有するより連続的なアセンブリが容易に得られる[ref.25]。しかしながら、メイトペアライブラリーおよび第3世代のシークエンシング技術の生成には、大量の高品質のDNAが必要であるが、大量の高品質DNAはフレッシュで豊富なサンプルからしか得られない。さらに、ライブラリーの調製およびシークエンシングは、ショートリードシークエンシング単独よりもはるかに高価である。
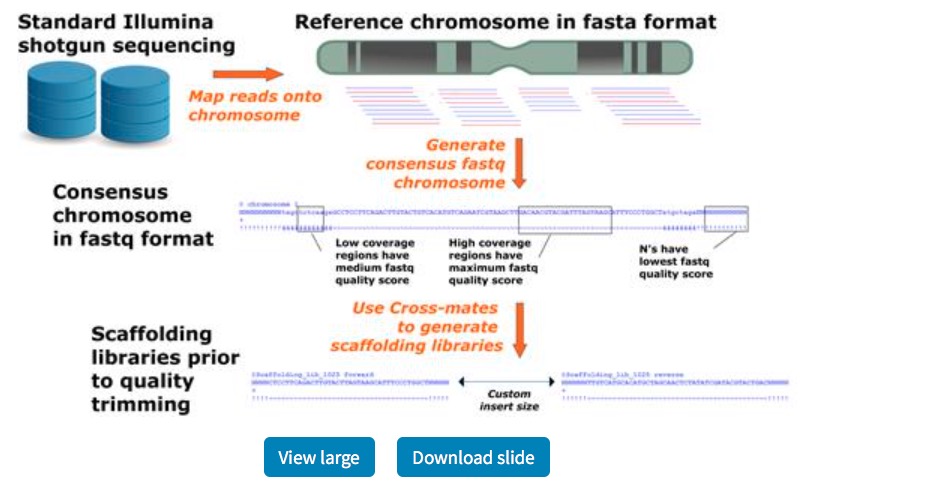
cross-species-scaffoldingの概略。マッピング、 コンセンサス配列生成、in silico mate pairシーケンシングリード発生の順に進む。論文より転載。
cross-species-scaffoldingに関するツイート(2018/10 現在なし)
インストール
ubuntu16.04でテストした。
本ツールを使うために必要
- bwa, seqtk, samtools, bcftools
- seq-scripts
テストランに必要
- curl, gzip
- sratoolkit (https://github.com/ncbi/sra-tools/wiki/Downloads)
seq-scripts
https://github.com/thackl/seq-scripts
本体 Github
git clone https://github.com/thackl/cross-species-scaffolding.git
cd cross-species-scaffolding/
#PATHにない依存を自動でインストール (実行内容はMakefile参照)
make dependencies
cd bin/ #最初からバイナリはある。使えなければmakeし直す。
#perlのライブラリが自動で入らなかったので、seq-fraqが使えない。手動で@INCにパスを通した。
1 Math::Random
sudo cpanm Math::Random
#cpanmがないならcurl -LOk http://xrl.us/cpanm && chmod +x cpanm && cp cpanm /usr/lical/bin/で使えるはず。
#2 perl5lib-Fastq
cd cross-species-scaffolding/ #ツールのルートに入れることにする
git clone https://github.com/BioInf-Wuerzburg/perl5lib-Fastq.git
export PERL5LIB=/path_to/cross-species-scaffolding/perl5lib-Fastq/lib/:$PERL5LIB
永続的に通すなら~/.bashrcや~/.bash_profileにexport~の行を書き込む。
#3 perl5lib-Fasta
git clone https://github.com/BioInf-Wuerzburg/perl5lib-Fasta.git
export PERL5LIB=/path_to/cross-species-scaffolding/perl5lib-Fasta/lib/:$PERL5LIB
#4 seq-scripts本体(mate pairなどのリードシミュレータ)
git clone https://github.com/thackl/seq-scripts.git
cd seq-scripts/bin/
#動作するか確認。
./seq-fraq
#OKならパスの通ったディレクトリコピーするかシンボリックリンクをパスの通ったディレクトリにはる(パスの通ったディレクトリを調べるにはecho $PATH)
> ./cross-mates
$ ./cross-mates
Usage:
cross-mates REF.fa TARGET-READS_1.fq [TARGET-READS_2.fq]
Generate in-silico mate-pair (and paired-end) libraries for super-scaffolding
from target genome reads and a closely related reference.
-o output directory [cross-mates-2018-10-04]
-t number of threads / parallel processes [10]
-i insert sizes, defaults: [500,1000,1500,2000,5000,10000,20000,50000,100000,200000]
-l read length [100]
-c coverage of mate-pair libraries [10]
-p paired-end vs. mate-pair insert size threshold [1000]
pairs with insert < -p will be fw/rev, pairs >= -p rev/fw
-z gzipped output libraries
-s write SOAP-denovo config for libraries to log
-V show script version
-h show this help
テストラン
#cross-species-scaffolding/のルートに移動して
cd cross-species-scaffolding/
make sample-yeast #実行内容はMakefile参照
実行方法
in silico generated mate pairシーケンシングリードを発生させる。
cross-mates ref.fa pair1.fq pair2.fq
ラッパーツールで、論文図1の3つのステップが順番に実行される。しかしエラーが起きたので個別に実行した。
または順番に実行する。
bwa mem -t 10 ref.fa pair1.fq pair2.fq | samtools view -bS - | samtools sort -@ 10 -o mapping.bam
samtools mpileup -uf mapping.bam > pileup
bcftools call -c pileup | vcfutils.pl vcf2fq |seqtk seq -l0 > consensus.fq
#すぐ上のコマンドでゲノムサイズの長いfastqが生成される。これをfastaに変える。オリジナルのスクリプトにはこのstepが存在しない。
seqtk -a consensus.fq > consensus.fa
#例えば50X, insert size 2000-bp、カバレッジ10の mate pairリード (github)
seq-frag mp -l 100 -c 50 -i 2000 < consensus.fa | interleaved-split 1>reads_1.fq 2>reads_2.fq
(seqtk seq の-l(エル)と-0フラグ: Convert multi-line FASTQ to 4-line FASTQ)
ミスアセンブリ、キメラアセンブリも発生します。精度を上げるには、できる限り似たゲノムを使うこと、できるだけディープシーケンスのデータでコンセンサスfastaを発生させることが推奨されています。scaffoldsを作成してから、チェックのためアセンブリの誤りが疑われる部位を壊すようなパイプラインも有効かもしれないです。
引用
Improving draft genome contiguity with reference-derived in silico mate-pair libraries
Grau JH, Hackl T, Koepfli KP, Hofreiter M
Gigascience. 2018 May 1;7(5).
関連ツール
*1
emperical error発生やエラートレーニングなしのシンプルなNGSリードシミュレーター