DNAシーケンシング解析では、通常、リードはただ1つのリファレンスゲノムにマッピングされる。 しかしながら、起源となるゲノムの確認を必要とする場合、複数のゲノムに対するマッピングが必要である。 複数のゲノムに対するマッピングは、汚染を検出するため、または検出されないまま誤って実験的結論につながる可能性のあるサンプルスワップを特定するためにも推奨される。 ここでは、リファレンスゲノムパネルにマップされるリードの割合を定量化することによってDNAサンプルの起源を検証するツールであるFastQ Screenを提示する。 FastQ Screenは、品質管理手段として、またDNAの起源が不明であるか複数の情報源を持つサンプルを分析するために日常的に使用されることを想定して実装されている。
2018年10月3日現在、F1000Researchで査読中になっています。
https://f1000research.com/articles/7-1338/v1
- HP
https://www.bioinformatics.babraham.ac.uk/projects/fastq_screen/
- Documentation
https://www.bioinformatics.babraham.ac.uk/projects/fastq_screen/_build/html/index.html
- 動画Tutorial
他にも3つ動画がある。
インストール
mac os10.12でテストした。
#Anaconda環境ならcondaで導入できる
conda install -c bioconda fastq-screen
> fastq_screen
$ fastq_screen
FastQ Screen - Map sequences against multiple genomes
www.bioinformatics.babraham.ac.uk/projects/fastq_screen
Contact: steven.wingett@babraham.ac.uk
Synopsis
fastq_screen [OPTIONS]... [FastQ FILE]...
Function
FastQ Screen is intended to be used as part of a QC pipeline.
It allows you to take a sequence dataset and search it
against a set of bowtie databases. It will then generate
both a text and a graphical summary of the results to see if
the sequence dataset contains the kind of sequences you expect.
Options
--aligner <func> Specify the aligner to use for the mapping. Valid
arguments are 'bowtie', bowtie2' (default) or 'bwa'.
Bowtie maps with parameters -k 2, Bowtie 2 with
parameters -k 2 --very-fast-local and BWA with mem -a.
Local aligners such as BWA or Bowtie2 will be better
at detecting the origin of chimeric reads.
--bisulfite Process bisulfite libraries. The path to the
bisulfite aligner (Bismark) may be specified in the
configuration file. Bismark runs in non-directional
mode. Either conventional or bisulfite libraries may
be processed, but not both simultaneously. The
--bisulfite option cannot be used in conjunction with
--bwa.
--bowtie <text> Specify extra parameters to be passed to Bowtie.
These parameters should be quoted to clearly
delimit bowtie parameters from fastq_screen
parameters. You should not try to use this option
to override the normal search or reporting options
for bowtie which are set automatically but it might
be useful to allow reads to be trimmed before
alignment etc.
--bowtie2 <text> Specify extra parameters to be passed to Bowtie 2.
These parameters should be quoted to clearly
delimit Bowtie 2 parameters from FastQ Screen
parameters. You should not try to use this option
to override the normal search or reporting options
for bowtie which are set automatically but it might
be useful to allow reads to be trimmed before
alignment etc.
--bwa <text> Specify extra parameters to be passed to BWA.
These parameters should be quoted to clearly
delimit BWA parameters from FastQ Screen
parameters. You should not try to use this option
to override the normal search or reporting options
for BWA which are set automatically but it might
be useful to allow reads to be trimmed before
alignment etc.
--conf <path> Manually specify a location for the configuration.
--filter <text> Produce a FASTQ file containing reads mapping to
specified genomes. Pass the argument a string of
characters (0, 1, 2, 3, -), in which each character
corresponds to a reference genome (in the order the
reference genome occurs in the configuration file).
Below gives an explanation of each character.
0: Read does not map
1: Read maps uniquely
2: Read multi-maps
3: Read maps (one or more times)
4: Passes filter 0 or filter 1
5: Passes filter 0 or filter 2
-: Do not apply filter to this genome
Consider mapping to three genomes (A, B and C), the
string '003' produces a file in which reads do not
map to genomes A or B, but map (once or more) to
genome C. The string '--1' would generate a file in
which reads uniquely map to genome C. Whether reads
map to genome A or B would be ignored.
A read needs to pass all the genome filters to be
considered valid (unless --pass specified).
When --filter is used in conjuction with --tag, FASTQ
files shall be mapped, tagged and then filtered. If
the --tag option is not selected however, the input
FASTQ file should have been previously tagged.
--force Do not terminate if output files already exist,
instead overwrite the files.
--get_genomes Download pre-indexed Bowtie2 genomes for a range of
commonly studied species and sequences.
--help Print program help and exit.
--illumina1_3 Assume that the quality values are in encoded in
Illumina v1.3 format. Defaults to Sanger format
if this flag is not specified.
--inverse Inverts the --filter results i.e. reads that pass
the --filter parameter will not pass when
--filter --inverse are specified together, and vice
versa.
--nohits Writes to a file the sequences that did not map to
any of the specified genomes. This option is
equivalent to specifying --tag --filter 0000 (number
of zeros corresponds to the number of genomes
screened). By default the whole input file will be
mapped, unless overridden by --subset.
--outdir <text> Specify a directory in which to save output files.
If no directory is specified then output files
are saved in the current working directory.
--pass <int> Used in conjunction with --filter. By default all
genome filters must be passed for a read to pass
the --filter option. However, a minimum number
of genome filters may be specified that a read
needs pass to be considered to pass the --filter
option. (--pass 1 effecitively acts as an OR
boolean operator for the genome filters.)
--quiet Suppress all progress reports on stderr and only
report errors.
--subset <int> Don't use the whole sequence file, but create a
temporary dataset of this specified number of
reads. The dataset created will be of
approximately (within a factor of 2) of this size.
If the real dataset is smaller than twice the
specified size then the whole dataset will be used.
Subsets will be taken evenly from throughout the
whole original dataset. By Default FastQ Screen
runs with this parameter set to 100000. To process
an entire dataset however, adjust --subset to 0.
--tag Label each FASTQ read header with a tag listing to
which genomes the read did, or did not align. The
first read in the output FASTQ file will list the
full genome names along with a score denoting
whether the read did not align (0), aligned
uniquely to the specified genome (1), or aligned
more than once (2). In subsequent reads the
genome names are omitted and only the score is
printed, in the same order as the first line.
This option results in the he whole file being
processed unless overridden explicitly by the user
with the --subset parameter
--threads <int> Specify across how many threads bowtie will be
allowed to run. Overrides the default value set
in the configuration file
--top <int>/<int,int> Don't use the whole sequence file, but create a
temporary dataset of the specified number of
reads taken from the top of the original file. It is
also possible to specify the number of lines to skip
before beginning the selection e.g.
--top 100000,5000000 skips the first five million
reads and selects the subsequent one hundred thousand
reads. While this option is usually faster than
comparable --subset operations, it does not prevent
biases arising from non-uniform distribution of
reads in the original FastQ file. This option should
only be used when minimising processing time is of
highest priority.
--version Print the program version and exit.
2018 Babraham Institute, Cambridge, UK
——
ランには、configurationファイルを必要とする。 configurationファイルにパラメータやマッパーのパスなどを記載して、fastq_screenのバイナリと同じディレクトリにセーブする。または"--conf"でconfigurationファイルのパスを指定する。
まずは、付属するfastq_screen.conf.exampleをfastq_screen.confにリネームする(fastq_screen.conf.exampleの場所はfindで検索して見つけてください, e.g, "find / -name fastq_screen.conf.example")。それからfastq_screen.confを開いて編集する。マッパー先頭のコメントアウト(#)を消し、パスを記載する。以下では3つのマッパーのパスを記載しているが、使用するマッパーだけでOK。

スレッドは8にした。
configurationファイル後半には使用するリファレンスを記載する。

複数のリファレンスがあるなら、複数のリファレンスのパスを記載する。ここではEcoli.fastaとref2.fastaのパスを記載した。出力名はEcoliとbacteriaXXXとした。リファレンスのfastaは、所定のディレクトリに入れて、indexをつけておく(BWAがマッパーなら、前もって"bwa index ref.fasta"しておく)。
/data/genome/の状態。2つのref.faとそのindexファイルがある。

詳細は2つ目の動画を見てください。
実行方法
どのリファレンスにどのくらいマッピングされるか調べる。上で作成したconfigファイルに書かれたリファレンス にマッピングしている。
fastq_screen --aligner BWA sample1.fastq sample2.fastq
どのリファレンスにどれくらいマッピングされたのかグラフで示したレポート.htmlファイルとリポート.PNGファイルが出力される。
.html
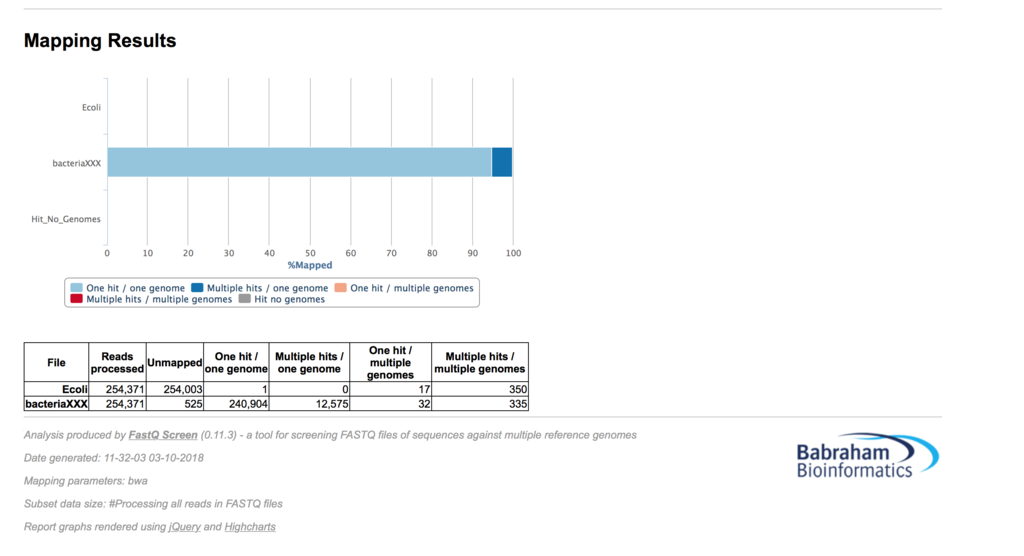
.PNG

マッピングを実行する。
fastq_screen --tag sample1.fastq sample2.fastq
ヘッダーがリネームされ、 どのリファレンスにマッピングされたかリードのヘッダーに追加された.fastq.gzが出力される。
このfastqを使って、フィルタリングを実行する。
fastq_screen --filter 1000 sample1.tagged.fastq.gz
--filter Produce a FASTQ file containing reads mapping to specified genomes. Pass the argument a string of characters (0, 1, 2, 3, -), in which each character corresponds to a reference genome (in the order the reference genome occurs in the configuration file). Below gives an explanation of each character.
0: Read does not map
1: Read maps uniquely
2: Read multi-maps
3: Read maps (one or more times)
4: Passes filter 0 or filter 1
5: Passes filter 0 or filter 2
-: Do not apply filter to this genome
"--filter 1000"なら、ゲノム1にユニークにはマッピングされ、ゲノム2、3、4にはマッピングされないリードを抽出する。"--filter 030"なら、ゲノム2には1回以上マッピングされ、ゲノム1、3にはマッピングされないリードを抽出する。"--filter 555"なら"0 & 2"になるので、3ゲノムいずれにもユニークにマッピングされないか(0)、マッピングされてもユニークでない(2)リードだけ抽出される。
taggingとフィルタリングを同時実行する。例えばどのリファレンスにもマッピングされていないリードを抽出する。
fastq_screen --nohits sample1.fq.gz sample2.fq.gz
例えば3ゲノムいずれにも完全ノーマップのリードを抽出するなら、"--filter 000"か "--nohits"
同じ名前のツールが他にもあるようです。ご注意ください。
引用
FastQ Screen: A tool for multi-genome mapping and quality control
Wingett SW, Andrews S
[version 1; referees: 3 approved, 1 approved with reservations].
F1000Research 2018, 7:1338 (doi: 10.12688/f1000research.15931.1)
