近年のAIを用いた手法の進歩は、構造生物学の分野に革命をもたらした。それと同時に、ハイスループットシークエンシングと機能的ゲノミクス技術により、バリアントの検出と生成がかつてない規模で可能になった。しかし、バリアントをタンパク質構造に「マッピング」し、バリアントがどのように疾患を引き起こすかをよりよく理解し、それによって治療法を設計するためには、これら2つの異種データを結びつける効率的なツールとリソースが必要である。ここでは、Genomics 2 Proteins Portal (G2P; g2p.broadinstitute.org/)を紹介する。G2Pは、19,996,443個の遺伝的変異を42,413個のタンパク質配列と77,923個のタンパク質構造にマッピングしたヒトプロテオーム全体のリソースであり、構造的・機能的特徴を包括的に網羅している。さらに、G2Pポータルは、ユーザーがタンパク質の残基単位のアノテーション(バリアント、スコアなど)やタンパク質構造をインタラクティブにアップロードして、関連付けを確立できるようにすることで、ゲノムとタンパク質の関連付けをデータベースの枠を超えて一般化している。このポータルは、研究者や科学者が、天然または合成のバリアントとその分子表現型との間の構造-機能関係を仮定するための、使いやすい発見ツールとして機能する。
Documentation
https://g2p.broadinstitute.org/docs
Aboutより
Genomics 2 Proteins (G2P)ポータルは、遺伝子スクリーニングの結果をタンパク質の配列と構造に結びつけるための探索ツールであり、主に2つの機能を提供する:
gnomAD、ClinVar、HGMDなどのデータベースからのヒト遺伝的バリアントの可視化と、UniProtデータベースからのタンパク質配列アノテーション(ドメイン、リガンド結合ポケット、活性部位などの特徴)を、タンパク質の配列と構造上にあらかじめマッピングしたものの視覚化。
バリエーション、タンパク質の特徴、機能的ゲノムアッセイ(飽和変異誘発など)からの情報をタンパク質の配列と構造にインタラクティブに自動マッピングする。
G2Pで使用されているデータの概要については、統計のページで確認できる。
https://g2p.broadinstitute.org/にアクセスする。
(自分のgoogleアカウントでログインして使用する)
チュートリアルで2つの使い方が説明されている。
1,一般に公開されているタンパク質の配列と構造にバリアントやスコアのセットをマッピングする。
2,手持ちのpdbファイルやmmcifファイルで定義された構造に対して、バリアントやスコアのセットをマッピングする。
ここでは1の流れを簡単に確認する。左のウィンドウから、遺伝子/タンパク質名で検索できる。例にあるLDLRで検索。

右側のウィンドウは2の解析向けのもの。
出力例
上側には遺伝子名、HGNC識別子、HGNC遺伝子ファミリー、Pantherタンパク質クラス、UniProt IDとUniProtタンパク質名などが表示されている。それぞれリンクになっていて、リンクからオリジナルのサイトに移動できるようになっている。

下半分にはタンパク質配列ビューアが表示され、該当するタンパク質のフィーチャーとバリアントが表示される。デフォルトの表示では、一番上にアミノ酸残基、Hydropathy plotやAF2の信頼性のpLDDTスコア、ジスルフィド結合の位置、ドメイン、モチーフ、リン酸化、ユビキチン化などの情報が示されている。

画面はドラッグしてタンパク質配列に沿って左右にスクロールできる。
色で情報が区別されている。

左側のメニューから表示する情報をON・OFFできる。

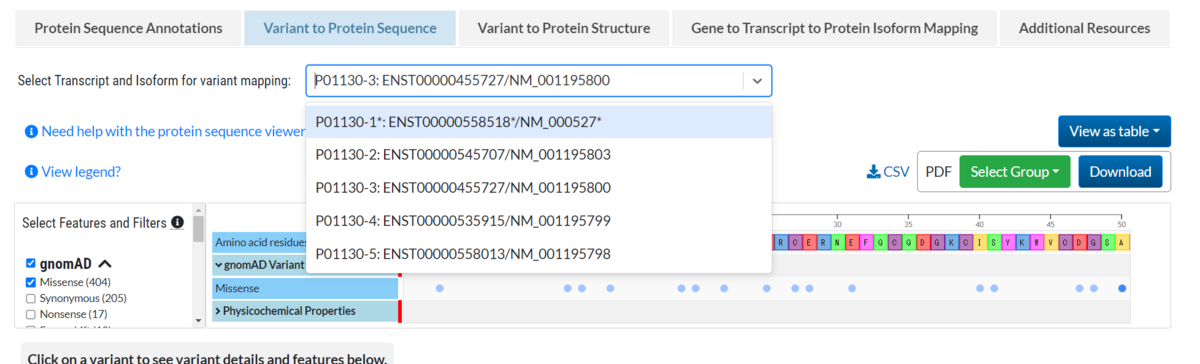
上のボタンからVariantタブに切り替えた。VariantタブではgnomDBやClinVarのバリアント情報をアミノ酸残基に沿ってプロットできる。

アイソフォームがある場合は切り替え可能。マイナーなものだと情報が少ない。
Variant to protein Structureタブでは、構造を選択して "click to view "するとタンパク質構造ビューアが起動してバリアントの位置を構造モデル上にマッピングできる。

1つ選択する。
起動した。左側のウィンドウは先ほどと同じで、右側に構造モデリングのパネルが表示される。

情報が横方向に展開されるのでワイドディスプレイのほうが作業しやすいと思われる。
選択した情報が追加される。ClinvarのIn frame indelをクリックした。

すると右のパネルに赤いプロットが追加(マッピング)された。

これがClinvarのIn frame indelの位置を表している。
構造モデルの拡大したい部分をクリックすると拡大される。再びクリックするとアミノ酸側鎖が表示される。

ビューアは右上のアイコンから全体表示に切り替えできる。
構造上のバリアントとフィーチャーをマッピング後、タンパク質構造の注釈はPyMOL互換ファイルでダウンロードできる。ダウンロードするには、小さいが、画面右上の↓ボタンをクリックする。

構造以外にも、全てのデータは.tsv形式でエクスポートできる。
画面右上の↓ボタンの隣をクリックすると左右分割から上下分割に切り替えできる。

start your proteinでは手持ちのpdbファイルをアップロードして、バリアントやスコアのセットをマッピングして分析することができる。

論文ではケーススタディも説明されています。読んでみて下さい。
引用
Genomics 2 Proteins portal: A resource and discovery tool for linking genetic screening outputs to protein sequences and structures
Seulki Kwon, Jordan Safer, Duyen T. Nguyen, David Hoksza, Patrick May, Jeremy A. Arbesfeld, Alan F. Rubin, Arthur J Campbell, Alex Burgin, Sumaiya Iqbal
bioRxiv, Posted January 02, 2024
