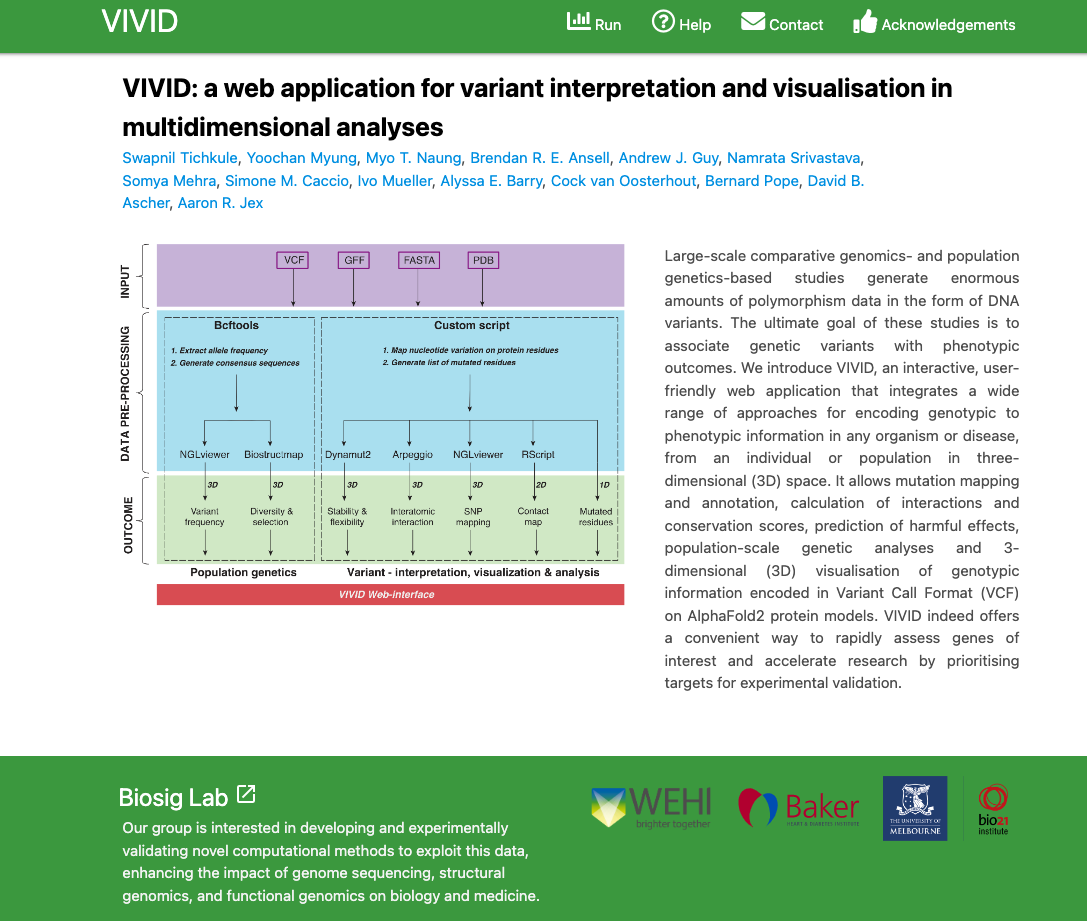
大規模な比較ゲノム研究や集団遺伝学研究では、DNAバリアントという形で膨大な量の多型データが生成される。これらの研究の最終的な目的は、遺伝的変異を表現型やフィットネスに関連付けることである。VIVIDは、あらゆる生物・疾患の遺伝型から表現型までの情報を、個体や集団から3次元空間上でコード化するための幅広いアプローチを統合した、インタラクティブで使いやすいウェブアプリケーションである。変異のマッピングやアノテーション、相互作用や保存スコアの算出、有害作用の予測、多様性や選択の解析、AlphaFold2タンパク質モデル上でVariant Call Formatでコード化された遺伝子型情報の3D可視化などが可能になっている。VIVIDは、適応進化や遺伝的負荷の研究において関心のある遺伝子を迅速に評価することを可能にし、実験的検証のためのターゲットの優先順位付けに役立つ。寄生原虫Plasmodium falciparumの進化遺伝学を探求し、機能的な抗体の潜在的なターゲットにおけるバランス選択のシグネチャーの地理的な差異を明らかにすることで、VIVIDの有用性を実証した。
help
https://biosig.lab.uq.edu.au/vivid/help
https://biosig.lab.uq.edu.au/vivid/static/help/VIVID_user-manual.pdf
上の動画で使い方は説明されています。ここでは使用する流れだけ簡単に紹介します。
webサーバー
https://biosig.lab.uq.edu.au/vivid/にアクセスする。
Runを選択
遺伝子のFASTA形式ファイルをアップロードする。ここではhelpからダウンロードできるテストデータ1を指定した。fastaファイルのヘッダーには遺伝子ID以外の名前が含まれていてはならない(遺伝子IDはGFFの名前; gene_ID=xxxと一致している必要がある)。

VCFファイルを指定する。テストデータの遺伝子では100以上のバリアントがコールされている。続いてGFFファイルも指定する。


GFFファイルは遺伝子のfastaファイルの遺伝子構造を示したアノテーションを使う必要がある。該当遺伝子以外のアノテーションも含まれていても問題ない。VCFは500MB以下のサイズが推奨されている。
AF2などで推論したPDBフォーマットのモデルファイル(変異がないタンパク質のもの)を指定する。PDBに登録されているタンパク質がある遺伝子なら、アクセッションIDで指定することもできる。

chain ID、遺伝暗号、他のパラメータを指定してサブミットする。

chain IDは複数のポリペプチド鎖から構成されるタンパク質において、各ポリペプチド鎖に割り当てられる1文字のID。ここではAとした(上の画像にはない)。
出力例
PDBファイルに存在する残基のみが一次配列で表現され、VCFからの同義変異(黄色)と非同義変異(紫色)がハイライトされる。

使用した遺伝子のUniProtおよびPfamデータベースへのリンクが提供される(画像の上部分)。
コンタクトマップ;タンパク質の3次元構造から得られる原子間の接触(コンタクト)を表す2次元のマップ。
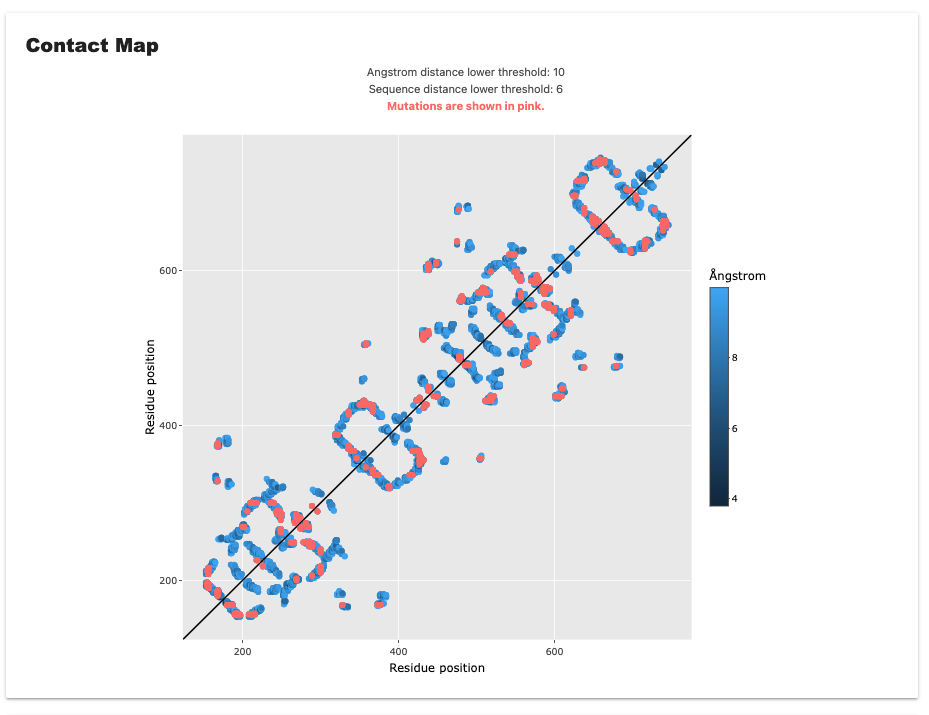
図は自由に拡大・縮小・移動ができる。変異が関与する残基は赤色でハイライトされている。
3D visualization。下のメニューボタンからフルスクリーンにして、自由に閲覧できる。拡大縮小はマウスのホイールボタン、ドラッグで回転、縦横方向のスクロールはCtrl+ドラッグで可能。

右上のメニューからTajima's Dなど様々な統計量で色付けできる。

ArpeggioはPythonで実装されたタンパク質、DNAまたは低分子リガンド内および間の相互作用を計算するWebサーバ(論文)。このパネルでは、各バリアントについてのArpeggioの情報がまとめられている。

Mutational Analysis; アミノ酸置換がタンパク質構造に与える影響を調べるパネル。タンパク質折りたたみの自由エネルギー変化を示す。

テストした時は結果が表示されるまで30分近くかかった。
引用
VIVID: A Web Application for Variant Interpretation and Visualization in Multi-dimensional Analyses
Swapnil Tichkule, Yoochan Myung, Myo T Naung, Brendan R E Ansell, Andrew J Guy, Namrata Srivastava, Somya Mehra, Simone M Cacciò, Ivo Mueller, Alyssa E Barry, Cock van Oosterhout, Bernard Pope, David B Ascher, Aaron R Jex
Molecular Biology and Evolution, Volume 39, Issue 9, September 2022