2022/08/03 誤字修正
2023/07/10 追記
2024/06/08 追記
高精度な構造予測手法により、一般に公開されているタンパク質の構造が雪崩のように増えている。これらの構造を検索することが、構造解析の主なボトルネックになりつつある。Foldseekは、大規模な構造セットを高速かつ高感度に比較することを可能にしする。Foldseekは、最新の構造アライナーと同等の感度を持ちながら、少なくとも2万倍以上の速さを実現している。Foldseekは無料のオープンソースソフトウェアで、foldseek.comやsearch.foldseek.comのウェブサーバとして利用することができる。
2023/04/05
Our #AlphaFold cluster site has new features:
— Martin Steinegger 🇺🇦 (@thesteinegger) April 4, 2023
① Search for clusters using a protein structure via #Foldseek
② Filter candidate clusters
③ Explore the cluster using a pavian-style interactive Sankey taxonomy plot
🌐https://t.co/gZ7B1wlWiR
👏 work by @milot_mirdita @jgyyy15 pic.twitter.com/3gT2ubiCUm
8/12
.@milot_mirdita implemented a taxonomy filter into the Foldseek server. The gif shows a search of the pretty heart-shaped fold found by @nicolabordin searched against the #alphafold/#pdb database filter to show only nematodes 🪱
— Martin Steinegger 🇺🇦 (@thesteinegger) August 10, 2022
🌐https://t.co/yZoYA0ilU1 pic.twitter.com/Ti4FJAPoCN
Our Foldseek server now includes the #AlphaFold UniProt DB clustered to 52M structures at ~50% seq. id & 80% cov. The full Foldseek AlphaFoldDB, including Cα, can be downloaded through the Foldseek databases module (~700GB download, ~950GB extracted) 1/6
— Martin Steinegger 🇺🇦 (@thesteinegger) August 1, 2022
🌐https://t.co/yZoYA0ilU1 pic.twitter.com/AlrH2bSRXN
Foldseek, our local structural aligner, is four orders of magnitude faster than SOA structural aligners at similar sensitivity. Allowing to detect hits in the midnight zone confidently.
— Martin Steinegger 🇺🇦 (@thesteinegger) February 9, 2022
Code: https://t.co/Beq97MYM2w
🌐 https://t.co/yZoYA0iTJz
📄 https://t.co/dElb3lNlGe
ここではFoldseek serverについて紹介します。
web server
https://search.foldseek.com/searchにアクセスする。
AlphaFold DBやPDBに対して類似タンパク質構造を検索できる。現在4つのデータベースが選択できる。
 PDBの他に、Swiss-protの配列をAlphafoldで予測したデータベースが利用できる。また、つい先日からはAlphafold/Uniprot50も追加された。Alphafold/Uniprot50というのは、AlphaFold UniProt DBを50%にクラスタリングしたものになる。AlphaFold UniProt DBは、7月末に公開された、UniRef90(Uniprot紹介)のタンパク質のほぼ全て(HP解説、数が異なるので全部か、あるいはUniRef90+αなのかどうかは分からない)をカバーする推定タンパク質構造データベースになる。このUniRef90ベースで作られたAlphaFold UniProt DBをそのままDBとして使うと相当重たいらしい(関連ツイート)。AlphaFold UniProt DBをクラスタリングして、検索速度を維持し、冗長な構造へのヒットを減らしたAlphafold/Uniprot50がここでは選べる(*1)。
PDBの他に、Swiss-protの配列をAlphafoldで予測したデータベースが利用できる。また、つい先日からはAlphafold/Uniprot50も追加された。Alphafold/Uniprot50というのは、AlphaFold UniProt DBを50%にクラスタリングしたものになる。AlphaFold UniProt DBは、7月末に公開された、UniRef90(Uniprot紹介)のタンパク質のほぼ全て(HP解説、数が異なるので全部か、あるいはUniRef90+αなのかどうかは分からない)をカバーする推定タンパク質構造データベースになる。このUniRef90ベースで作られたAlphaFold UniProt DBをそのままDBとして使うと相当重たいらしい(関連ツイート)。AlphaFold UniProt DBをクラスタリングして、検索速度を維持し、冗長な構造へのヒットを減らしたAlphafold/Uniprot50がここでは選べる(*1)。
2022/11/15追記
利用できるデータベースがさらに増えている。

Mgnify-ESM30 v1は最近公開されたESM Metagenomic Atlasのこと(Meta社の再編成の関係で2023年8月になって開発チームは解散した。今後の更新は厳しそう)。
PDBファイルを貼り付けるかアップロードしてSEARCHをクリックする。PDBかAlphaFold DBにあるタンパク質なら、アクセッションIDでも指定できる。

テストした時は数十秒で結果が得られた。NCBIでデータベースをUniprotにしてBLASTPサーチする時よりも短い時間かもしれない。
出力例
NCBI BLASTに似た出力を得られる。上のほうが構造アラインメントのカバー部位になっている。色はスコアの高さを表す。

(中略)

下の方が詳細になっている。スコアとE-valueが表示されている。
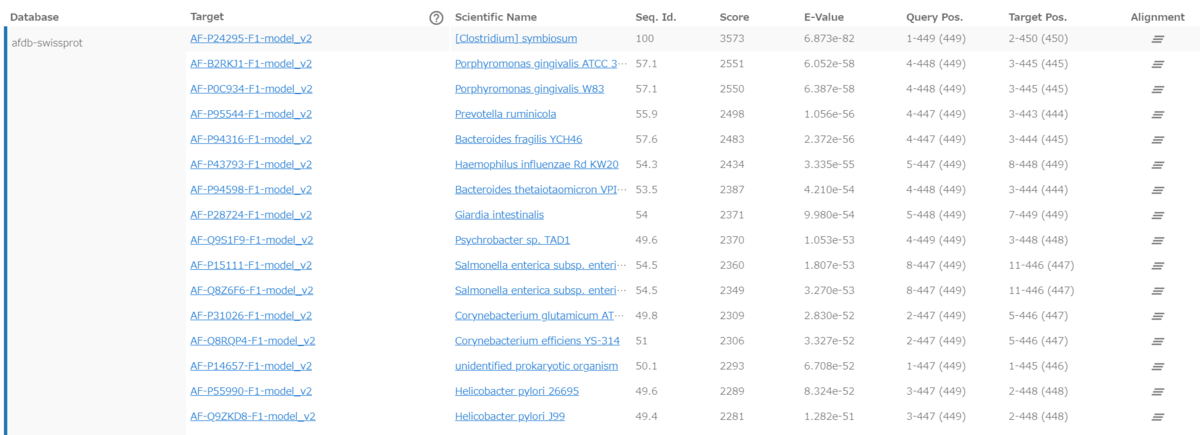
Targetの列のAlphaFoldタンパク質識別子は、AlphaFold DBにリンクしている。Scientific NameはNCBIにリンクしている。
右端のAlignmentをクリックするとアラインメントが表示される。ヒットしたタンパク質構造も表示される。

TM-scoreは、タンパク質構造のトポロジー的な類似性を評価するためのメトリックス。
結果は左端のメニューのDownlaod ~から入手できる(画像が黒いのはダークモードを使用しているため)。
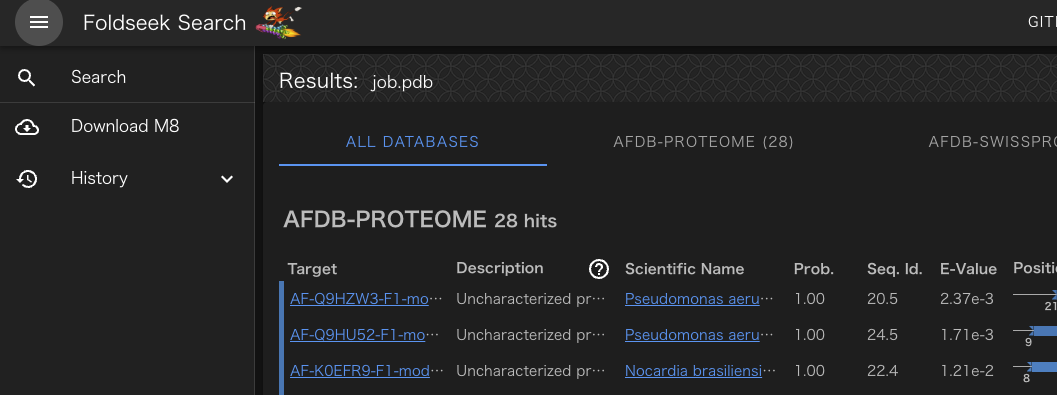
ダウンロードして解凍した。
このHTMLレポートは、CLI環境でFoldseekをランした時にもオプションをつければ出力されます(--format-mode 3使用)。Foldseekについては近いうちに紹介します(リンク)。
2024/06/08追記
しばらく前からタンパク質の1次配列も使用できるようになりました。やり方は、FASTA形式の配列をウィンドウ内にペーストし(400a.a以内)、PREDICTボタンで構造推定の方法を選びます。

試しに高速なESMfoldをクリックします。そのまましばらく待ちます(数秒~数十秒)。
ウィンドウ内が予測結果のPDBフォーマットファイルに上書きされました。

この状態でそのまま問い合わせできます。
引用
Foldseek: fast and accurate protein structure search
Michel van Kempen, Stephanie S. Kim, Charlotte Tumescheit, Milot Mirdita, Johannes Söding, Martin Steinegger
bioRxiv, Posted February 09, 2022
追記
Foldcomp: a library and format for compressing and indexing large protein structure sets
Hyunbin Kim, Milot Mirdita, Martin Steinegger
Bioinformatics, Volume 39, Issue 4, April 2023, btad153
Fast and accurate protein structure search with Foldseek
Michel van Kempen, Stephanie S. Kim, Charlotte Tumescheit, Milot Mirdita, Jeongjae Lee, Cameron L. M. Gilchrist, Johannes Söding & Martin Steinegger
Nature Biotechnology (2023)
2023/06/30 追記
Foldseek gives AlphaFold protein database a rapid search tool
The structural search program makes finding proteins with similar 3D shapes easy.
https://www.nature.com/articles/d41586-023-02205-4
"Foldseekをテストするために、Steinegger氏の研究チームは、AlphaFold 2を使って形状が予測された365,000個のタンパク質のデータベースを使用した。スコアは、アルゴリズムが偽陽性を検索する前に、どれだけ多くの「真陽性」(すなわち、原子モデリングに従って一定の類似性閾値以上のスコアを獲得したタンパク質)を検索したかに基づいている。Foldseekは、2つの一般的な構造ベースの検索ツールであるTM-alignとDaliを凌駕し、それぞれ24%と8%の性能向上、約35,000件と20,000件の高速検索を実現した。CLE-SWと呼ばれる構造アルファベットベースのツールとの比較では、Foldseekの方が23%優れており、11倍高速であった。"
2023/12/13 追記
Is Protein BLAST a thing of the past?
https://www.nature.com/articles/s41467-023-44082-5
"AlphaFoldのようなタンパク質構造検索ツールは、BLASTによるタンパク質配列検索に取って代わるのだろうか?遠隔相同性検出のための構造検索を使用すること、そして、主要な配列検索ツールであるタンパク質BLASTが構造情報を取り入れる努力をすべき理由について、その可能性を議論する。"
関連
*1 一般に、遺伝子配列よりもタンパク質の一次構造、タンパク質の一次構造よりも高次構造で探す方が遠縁な配列間の類似性を探しやすい。よって、Uniref50レベルのデータベースに対してタンパク質一次配列レベルで類似性検索するよりも、Uniref50レベルの推定立体構造データベースに構造レベルで類似性検索するほうが、似たタンパク質の検出感度と検出精度は高くなると考えられる(関連論文)。
例を挙げる。例えば、BLSTPでタンパク質全体に渡って30%類似したタンパク質が見つかり、それが同様の機能を有しているのか、偶然一次配列が似ているだけで、タンパク質の立体構造は異なり、したがって機能も異なるのか判断する状況を考える(短いためE-valueもあまり低くならないとする)。これはコア遺伝子探索などの文脈で良く発生する。従来、このようなケースで精度の高い答えを情報のみから得る事は難しかった。そのため、間接的な証拠を集めたり、interproscanでドメインレベルの保存性を調べたりすることが行われていた(EMBL-EBIサービス)。ドメインファミリーで全く異なるものがヒットしてくるなら、タンパク質の構造や機能は異なることが予想されるからである。しかし、精度の高い推定構造情報が得られ、それをクエリにして短時間で類似性の高いタンパク質を包括的な(推定)立体構造DBに対して問い合わせできるなら、このような作業は不要になる。精度の高い推定立体構造が得られ、超高速な構造アライナーと包括的な(推定)立体構造データベースも利用できるようになり、必要な道具と環境が揃ったことで、研究の進め方が大きく変化してきているのかもしれない(注;大きく変化したかどうかは、後になって当時を振り返らないと分からない。例えば論文の出方を調査するなど)。
関連するツイート
昨年より、AlphaFold DB のすべてのタンパク質ページに「構造類似性クラスタ」が追加されている。
https://twitter.com/paulynamagana/status/1762583367608828157
